次世代シーケンス(NGS)テクノロジーの導入により、大量のゲノムシーケンスデータを生成できるようになった。これにより、個人のゲノムをシーケンシングしてde novoアセンブリすることが可能になった。約10年前、NGSテクノロジーにより、二倍体de novoゲノムアセンブリの比較的高いカバレッジと生成で、ジェームズD.ワトソンとクレイグベンターの2人のゲノムのシーケンスが可能になった[ref.2、3]。これに続いて、アジアの個々のゲノム(YH)、韓国の個人(AK1)、およびサンプルNA18507の2倍体de novoゲノムアセンブリが報告され、以前に利用可能な個々のゲノムと比較された[ref.4、5、6]。さらに最近では、7人の二倍体DNA配列がアセンブリされ、アセンブリ用に開発されたソフトウェアパイプラインとともに報告された[ref.7]。シーケンスがより安価で手頃な価格になったが、精密医療の時代にNGSを日常的に適用するという課題は、特に近い将来の個人のゲノムアセンブリのバイオインフォマティクスソリューションに大きくかかっている。この目的のために、ショートリードおよびロングリードのNGSデータを使用したde novoアセンブリに関するさまざまなアセンブリツールが文献で提案および報告されている。そのようなツールの例としては、SOAPDenovo2 [ref.8]、ALLPATHS-LG [ref.9]、ABySS [ref.10]、MaSuRCA [ref.11]、およびSPAdes [ref.12]があり、これらはショートリードデータとロングレンジのメイトペアライブラリを使う。Canu[ref.13]、MECAT [ref.14]、Celera [ref.15]、およびFalcon [ref.16]は、Oxford NanoporeやPacBioなどのプラットフォームで生成されたロングリードデータを利用して、de novoアセンブリを生成する。これらのアセンブリツールで使用されるアプローチは異なるため、scaffoldsまたはcontigs(連続シーケンス)およびそれらによって生成されるアセンブリの品質も大きく異なる。これらのアセンブリソフトウェアを比較する問題に対処するために、Assemblathonコンソーシアムは、シミュレート(人工)データ[ref.17]および非哺乳類脊椎動物種[ref.18]のデータに対する複数のアセンブラのパフォーマンスを評価するエフォートを行った。ゲノムアセンブリの最適なソリューションとして選ばれたパッケージはなかったが、これらの2つの研究は、アセンブリの全体的な品質の重要な測定値を確立した。別の研究[ref.19]では、著者らは、ヒト14番染色体と小さなゲノムを持つ他の3つの生物について、複数のアセンブリツールによって生成されたde novoアセンブリを比較した。これらの作業により、複数の観点からアセンブリの品質を測定するための、受け入れられているいくつかのメトリックの基礎が確立された。
これらのメトリクスを使用して、新しくアセンブリされたゲノムの品質を評価するためのいくつかのツールが文献で報告されている。REAPR [ref.20]は、アセンブリに使用されたリードをアセンブルされたコンティグにバックアラインメントする手法により品質メトリックを生成する、リファレンス不要のツールである。一方、QUAST [ref.21]とその改良版QUAST-LG [ref.22]は、信頼できるリファレンスゲノムの存在下でde novoゲノムアセンブリを評価できるツールである。これらのツールは、de novoアセンブリのコンティグまたはスキャフォールドを特定のリファレンスゲノムの染色体にアラインメントし、これらのアラインメントに基づいて品質メトリックを報告する。また、N50値などのリファレンスフリーのメトリックもレポートするが、これらのツールの主な目的は、リファレンスゲノムを使用して、ゲノムのカバレッジ率、コンティグ中のミスアセンブリの数など、リファレンスに依存するメトリックを生成することである。主な欠点は、個々のコンティグの品質が完全に評価されておらず、de novoゲノムアセンブリの全体的な品質を反映できる単一の品質メトリックを生成しないことである。これらのツールによって報告される複数のメトリックは互いに矛盾する場合があり、複数のアセンブリを比較およびランク付けするときにエンドユーザーを混乱させる可能性がある。また、これらのメトリックは、ランク付けのために複数のアセンブリ間で一貫性なく実行される可能性があり、全体的な品質の観点からアセンブリをランク付けする際の信頼性が低くなる。
この問題を解決するために、複数の十分に確立されたメトリックの統合により、個々のscaffolds/contigsの品質スコアを使用してデノボアセンブリの品質を評価するデノボアセンブリ品質評価ツール(dnAQET)を開発した。 dnAQETによって生成された品質スコアの有効性を示すために、4つの異なる合成アセンブリセットにツールを適用した。最初のデータセットでは、dnAQETの品質スコアは、より長いscaffoldsのアセンブリの方が優れていることを実証した。2番目のデータセットでは、アセンブリのスキャフォールドで識別されるミスアセンブリの数が増加するにつれて、品質スコアが低下することを示した。 3番目のデータセットでは、アセンブリのリファレンスに対するカバレッジの増加が、それらのアセンブリに対して計算されたdnAQETの品質スコアにプラスの影響を与えることを示すように設計された。これは予想と一致した。最後に、4番目のデータセットを使用して、アセンブリの冗長性が増加すると、それらに割り当てられたdnAQETスコアが低くなる方法を調査した。
また、染色体の個々の品質スコア全体のスコアを5つの生物(哺乳類4匹と魚1匹)の利用可能なリファレンスゲノムビルドで計算した。これらのリファレンスビルドのdnAQETの品質評価は、リファレンスゲノムのより最近のビルドの方が古いものより良くなると予想され、結果は予想と一致した。さらに、サンプルNA24385の6つのde novoアセンブリを使用して、これまでの十分に確立されたメトリックでは互いに矛盾する結果を示す可能性を示した。リファレンスゲノムビルドをランク付けする時には一貫しない結果を示す可能性かあることを示した。(一部略) 一方、dnAQETの全体的な品質スコアは、両方のデータセットのアセンブリのランク付けにおいて非常に一貫しており、アセンブリの品質を評価する信頼性と、de novoアセンブリを比較するベンチマークメトリックとして使用される適合性を証明した。また、dnAQETは非常に高速でスケーラブルであり、合理的な量の計算メモリで、ヒトなどの大規模で複雑なゲノムのアセンブリを処理できることを示した。
(以下略)
インストール
ubuntu18.0.4のLTSでテストした。
依存
The dnAQET is a Java package designed to be used in a Unix based operating system (such as Linux, MacOS, etc.) and it requires Java 1.7 Runtime Environment installed. It depends on Minimap2 tool and MUMmer4 package, which requires the following to run successfully.
HP
HPからtar ballをダウンロードし、解凍する。中にはjavaのバイナリ(.jar)やマニュアルのREADME.pdf等が入っている。
> java -jar dnAQET.jar
# java -jar dnAQET.jar
dnAQET version 1.0.25890
Usage: <main class> [options] [command] [command options]
Command: evaluate Evaluate a de novo assembly by calculating a quality score and also assign quality scores to individual contigs/scaffolds with respect to a reference genome
Usage: evaluate [options]
Options:
-q, --alignmentTool
Tool used for the alignment of the contigs/scaffolds back to reference
Default: minimap2
Possible Values: [minimap2, nucmer]
-d, --evaldestfolder
Evaluation destination directory
Default: RESULT
* -f, --file
Input contig file
-j, --jobcommand
Alignment job submission command
Default: multithread
Possible Values: [sbatch, qsub, multithread]
-p, --jobcommandparams
Alignment job submission command parameters in quotes
Default: <empty string>
-l, --lengthscalingfactor
The length scaling factor constant that is used to scale the quality
value of a scaffold/contig based on its size. The default value is the size of
the smallest chromosome of the reference genome which is used to compute the
regression model
-k, --model
A model file
-n, --npartition
Number of partitions for the input contig file
Default: 1
-m, --ov_dist_threshold
Distance (or overlap) treshold for the alignments of consecutive parts of
a contig to be considered a RELOCATION
Default: 1000
* -r, --ref
A trusted reference genome file
-t, --threadno
Number of threads
Default: 4
Command: model Given a reference compute a multiple linear regression model to be used in evaluation
Usage: model [options]
Options:
-q, --alignmentTool
Tool used for the alignment of the contigs/scaffolds back to reference
Default: minimap2
Possible Values: [minimap2, nucmer]
-j, --jobcommand
Alignment job submission command
Default: multithread
Possible Values: [sbatch, qsub, multithread]
-p, --jobcommandparams
Alignment job submission command parameters in quotes
Default: <empty string>
-d, --modeldestfolder
Model file destination directory
Default: MODEL
* -r, --referencefile
Input reference file path
-t, --threadno
Number of threads
Default: 4
Command: reevaluate Re-compute a previous evaluation with given parameters
Usage: reevaluate [options]
Options:
-d, --evaldestfolder
Re-evaluation destination directory
-l, --lengthscalingfactor
The length scaling factor constant that is used to scale the quality
value of a scaffold/contig based on its size. The default value is the size of
the smallest chromosome of the reference genome which is used to compute the
regression model
-m, --ov_dist_threshold
Distance (or overlap) treshold for the alignments of consecutive parts of
a contig to be considered a RELOCATION
Default: 1000
* -s, --sourcefolder
The results output directory from a previous evaluation
-t, --threadno
Number of threads
Default: 4
実行方法
アセンブリ結果を評価する。アセンブルして得た配列と信頼できるリファレンス配列を指定する。
java -jar dnAQET.jar evaluate -f input_assembly.fa -r trusted_ref.fa \
-q minimap2 -d output_dir -t 12
- -q Tool which will be used for the alignment of the contigs/scaffolds back to reference Default: minimap2
- -r A trusted reference genome file. (this is a required parameter, not optional!)
- -f Input contig file path (this is a required parameter, not optional!)
-
-t Number of threads for alignment (in case -j is selected to be 'multithread') step and for evaluation step. Default: 4
出力ディレクトリ

様々なreportが出力される。
qualityScoreHistogram.jpeg

output_dir/report/

assembly.stat

scaffolds.alignment

scaffolds.stat
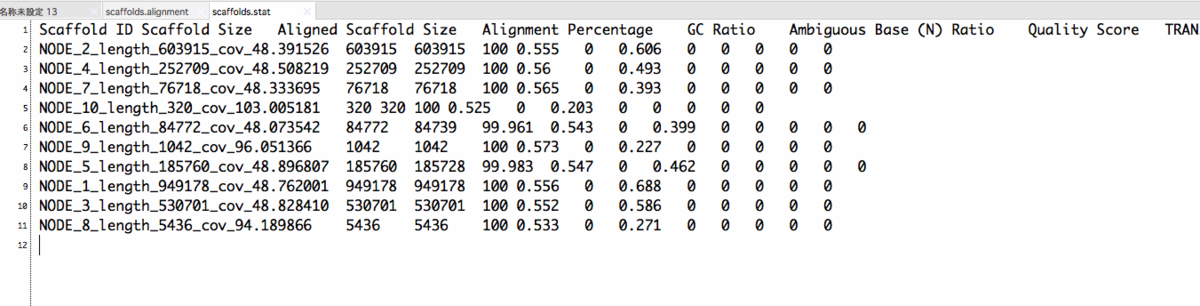
misassembly.report

ミスアセンブリはなかった(小さなゲノムのシミュレーションデータのアセンブルを評価している)
引用
dnAQET: a framework to compute a consolidated metric for benchmarking quality of de novo assemblies
Gokhan Yavas, Huixiao Hong, Wenming Xiao
BMC Genomics volume 20, Article number: 706 (2019)