2020 8/9 論文追記
Visualizationは、現在のゲノム革命において、バリアント、発現パターン、進化による変化、および他の多くの関係を検査し、理解するために非常に重要な役割を果たす(Preprint ref.1~3)。しかし、構造変化可視化時のリードとリファレンス、またはリファレンスとリファレンスとのアライメント情報の大部分は、リファレンスの座標しか示さないほとんどのゲノムブラウザの1次元ビューのため失われる。ロングリードまたはアセンブして作ったコンティグで構造変化を理解するためには、複数の染色体からのアラインメントや他のゲノム情報などのより多くの文脈を理解する必要がある。
この問題は、リファレンスとクエリシーケンスの両方に沿ったアラインメントとサンプル内の関連バリアントコールを表示するインタラクティブなオンラインビジュアライゼーションツールであるRibbon(genomeribbon.com)を作成することで解決する。リボンは、複数の染色体にわたる多くのリードパターンを示し、個々のリードの詳細な検査を可能にする(Preprint 補足1)。例えば、ここでは、CYTH1およびEIF3H遺伝子を連結するSK-BR-3乳癌細胞株における遺伝子融合を示す。これまでにトランスクリプトームref.4-6で見つかっているが、ゲノムシーケンシングではこれら2つの遺伝子間のダイレクトな染色体融合の証拠は提供しなかった。 SMRTシークエンシングref.7の後、リボンは、実際には、2つの遺伝子にまたがるロングリードがあることを示した(図1a)(以下略)。結論として、リボンは、複雑なゲノムアラインメントを表示するための強力でインタラクティブなWebツールである。
Preprintです。
公式ページ
紹介動画
インストール
https://github.com/MariaNattestad/ribbon
XAMPPをいれて自分でサーバーを立ち上げることもできるようになっている。ここではweb版を紹介する。
ラン
SplitThreaderと連携しており、SplitThreaderで構造変化を可視化し、それをRibbonにエキスポートして、実際のロングリードのアライメント状況を分析できるようになっている(SplitThreader紹介)。
Ribbonにアクセスする。
上のメニューのExamplesの1つ、ゴリラのアセンブリをヒトゲノムにアライメントしたデータを表示してみる。

上半分はゴリラアセンブリとヒトゲノムのHarr plot(ドットプロット)になっている。
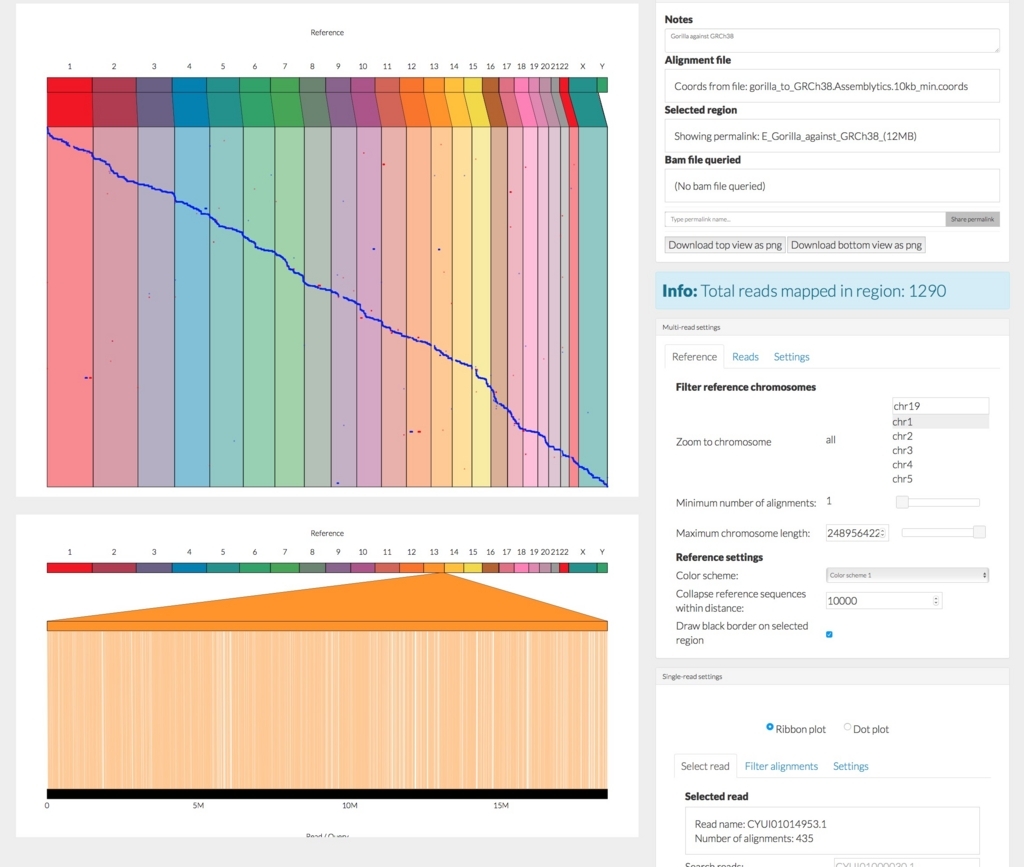
青のプロットをクリックすると、下半分はその染色体だけの表示に切り替わる。chr10。

今現在、下半分に表示されているのは、chr10のうち、上の画像の青が濃くなっている部分だけになる。上の画像のドットをクリックすると、下の画像はその領域に切り替わるようになっている。
メニューのRibbonをDot plot表示に切り替え。

メニューのFilter alignments => Minimum % identityを97%まであげた。
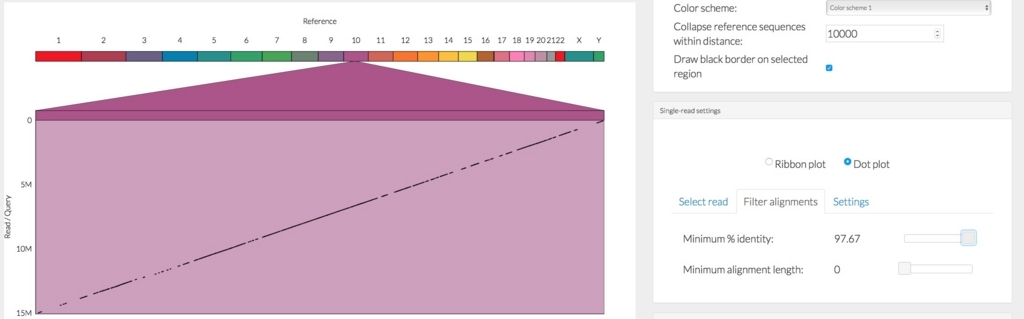
メニューのReference => Reference settingsの数値を1000に修正し、1000 bp以上アライメントがスキップされた領域に線をつけた。

自分のデータを解析するには、sam/bamか、Mummerのshow-coords の解析結果を使う。ショートリードやロングリードのマッピングbamからバリアントコール部位のリードアライメント状況を可視化したり、アセンブリのcontig、ゲノム同士の比較などができる。
データは右のメニューからアップロードする。バクテリアのMiseqシーケンスデータから得たbam、そのバリアントコールのvcf、bedファイルをアップロードして、表示してみた。ゲノムとbedはUCSC genome browser(アーキアとバクテリア)から入手したものをそのまま使った。大きな欠失部位がコールされた部位のリードアライメント状況(関連するリードだけ描画されている)。

コール部位をクリックすればジャンプでき、コールされた構造変化を素早く確認できる。大規模なtranslocationなどの構造変化を見やすく工夫し、直感的に理解しやすくなっている。
引用
Ribbon: Visualizing complex genome alignments and structural variation
Maria Nattestad, Chen-Shan Chin, Michael C Schatz
bioRxiv preprint first posted online Oct. 20, 2016;
doi: http://dx.doi.org/10.1101/082123.
https://www.biorxiv.org/content/biorxiv/early/2016/10/20/082123.full.pdf
2020 8/9 追記
Ribbon: Intuitive visualization for complex genomic variation
Maria Nattestad, Robert Aboukhalil, Chen-Shan Chin, Michael C Schatz
Bioinformatics, Published: 07 August 2020
