2022/03/13 文章修正
2022/03/15 誤字修正
次世代シーケンサーの進歩により、リードに付着したアダプターや低品質の塩基が 直接的に、あるいは暗黙のうちに、ダウンストリーム解析の妨げとなる。たとえば、偽陽性 一塩基多型(SNP)、断片化したアセンブリが生成される。そのため 、アダプターや低品質塩基を正確に除去する高速トリミングアルゴリズムが必要である。本発表ではAtriaという、ペアエンドのリードからマッチするアダプターの重複する可能性のある領域を探す、高速かつ慎重に設計されたバイトベースの マッチングアルゴリズム(O (n) time with O (1) space)を開発した。Atria はまた、マルチスレッドを実装している。配列処理とファイル圧縮の両方を行い、シングルエンドリードをサポートする。シミュレーションデータと実データの様々なトリミングとランタイムベンチマークで、Atriaは良好な結果を出している。また、高速かつ軽量なバイトベースのマッチングアルゴリズムも提供している。Atriaは、プライマー検索や、シードスキャニングなど、様々な用途で使用できる。
特徴(Githubより)
- 圧縮されたFastqsでも高速に処理
- 高精度なイルミナアダプタトリミング
- ペアエンドコンセンサスコール
- 品質トリミング
- ポリXテールトリミング
- 3'末端および5'末端のハードクリップ
- Nテールトリミング
- N塩基の数でリードをフィルタリング
- リードの長さによるフィルタリング
- リードの複雑さによるフィルタリング
インストール
リリースからatria-3.1.1-linux.tar.gzをダウンロードしてテストした(mac os向けはバージョンによってある場合とない場合がある)もしくはソースからビルドする。Julia言語で開発されている。
依存
- pigz and pbzip2 are required.
#パスの通ったディレクトリにシンボリックリンクを張るかbinにパスを通す
sudo ln -s atria-3.1.1/bin /usr/local/bin/atria
export PATH=atria-3.1.1/bin/:$PATH
> ./atria
Atria v3.1.1
≡≡≡≡≡≡≡≡≡≡≡≡≡≡
An ultra-fast and accurate adapter and quality trimming software designed for paired-end sequencing data.
If you use Atria, please cite
│ Jiacheng Chuan, Aiguo Zhou, Lawrence Richard Hale, Miao He, Xiang Li, Atria: an ultra-fast and accurate trimmer for adapter and quality trimming, Gigabyte, 1, 2021 https://doi.org/10.46471/gigabyte.31
Github: https://github.com/cihga39871/Atria
Usage
=======
Try atria -h or atria --help for more information.
Input and Output
––––––––––––––––––
The input files should be paired-end FastQ(.gz|.bz2) files (in the same order), or single-end fastqs:
1. Read 1 files: -r X_R1.FQ Y_R1.FQ.GZ ...
2. Read 2 files (optional): -R X_R2.FQ Y_R2.FQ.GZ ...
Output all files to a directory: -o PATH or --output-dir PATH. Default is the current directory.
Trimming methods
––––––––––––––––––
Atria integrated several trimming and read filtration methods. It does the following sequentially.
1. Poly X Tail Trimming: remove read tail with poly X.
suggest to enable --polyG for Illumina NextSeq/NovaSeq data.
• enable: --polyG, --polyT, --polyA, and/or --polyC (default: disabled)
• trim poly X tail if length > INT: --poly-length 10
2. Adapter Trimming
• specify read 1 adapter: -a SEQ or --adapter1 SEQ (default: AGATCGGAAGAGCACACGTCTGAACTCCAGTCA)
• specify read 2 adapter: -A SEQ or --adapter2 SEQ (default: AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT) (if paired-end)
• disable: --no-adapter-trim
• if adapter is unknown, use --detect-adapter.
3. Paired-end Consensus Calling: the overlapped regions of read pairs are checked and corrected. It is available only when input files are paired-end and Adapter Trimming is on.
• disable: --no-consensus
4. Hard Clip 3' end: resize reads to a fixed length by discarding extra bases in 3' end.
• specify the number of bases to keep: -C INT or --clip-after INT (default: disabled)
5. Hard Clip 5' end: remove the first INT bases from 5' end.
• specify the number of bases to remove: -c INT or --clip5 INT (default: disabled)
6. Quality Trimming: trim low-quality tails. (Trimming read tails when the average quality of bases in a sliding window is low.)
• specify average quality threshold: -q 20 or --quality-score 20 (default: 20)
• specify sliding window length: --quality-kmer 5 (default: 5)
• specify FastQ quality format: --quality-format Illumina1.8, or --quality-format 33 (default: 33, ie. Illumina1.8)
• disable: --no-quality-trim
7. Tail N Trimming: trim N tails.
• disable: --no-tail-n-trim
8. N Filtration: discard a read pair if the number of N in one read is greater than a certain amount. N tails are ignored if Tail N Trimming is on.
• specify # N allowed in each read: -n 15 or --max-n 15 (default: 15)
• disable: -n -1 or --max-n -1
9. Read Length Filtration: filter read pair length in a range.
• specify read length range: --length-range 50:500 (default: 50:500)
• disable: --no-length-filtration
10. Read Complexity Filtration: filter reads with low complexity.
Complexity is the percentage of base that is different from its next base.
• enable: --enable-complexity-filtration (default: disabled)
• specify complexity threshold: --min-complexity 0.3 (default: 0.3)
Parallel computing
––––––––––––––––––––
Atria has two parallel methods: multi-threading and multi-core. You can enable them both.
1. Multi-threading uses multiple threads to process one sample. The speed marginal gain drops as the threads increase: -t 8 or --threads 8. (Default: 8)
2. Multi-core processes multiple samples at the same time. No marginal gain loses if IO and memory are sufficient: -p 4 or --procs 4. (Default: disabled)
3. If memory is not sufficient, use --log2-chunk-size INT where INT is from 23 to 25. Memory usage reduces exponentially as it decreases.
実行方法
ペアエンドFastq、またはシングルエンドFastqに対応している (.gz|.bz2)。
- シングルエンドは-rで指定し、ペアエンドFastqは-rと-Rでそれぞれ指定する。
- Poly Xトリミングには--polyGを付ける(デフォルトではオフ)。イルミナのNextSeq/NovaSeqでは付ける事が提案されている。
- アダプタートリミングには-aと-Aでペアエンドそれぞれのアダプター配列を指定する。もしアダプター配列が不明なら、--detect-adapterを付けて、まずアダプターを確認する(下の例参照)。
- ペアエンドの場合は、ペアエンドがオーバーラップする領域が認識され、低品質側の塩基のエラーが修正される(アダプタートリミングがオンの時だけ)。オフにするには--no-consensusを付ける。
- Q20以下の低品質塩基のクオリティトリミングを行うなら-q 20をつける。オフにするなら--no-quality-trimを付ける。スライディングウィンドウサイズはデフォルトでは--quality-kmer 5となっている。
- FastQの品質形式は--quality-format Illumina1.8, or --quality-format 33。 デフォルトでは33, i. Illumina1.8)
- Nテイルのトリミングは自動で行われる。オフにするなら--no-tail-n-trimを付ける。
- 末端の塩基をハードトリミングできる。5’側の末端1塩基をトリミングするには-c 1を付ける。3’側の末端1塩基をトリミングするには残す長さを指定する。151-bp読んで1-bpトリミングするなら-C 150を付ける。
- N塩基が多い(ペアエンド)リードをフィルタリングする。-n 15ならNが16以上のペアは捨てられる。オフにするには-n -1とする(マイナス1)。デフォルトでは-n 15。
- リード長トリミングは最短:最長の範囲で指定する。--length-range 50:500なら、トリミング後に50塩基以下、500塩基以上のリードはフィルタリングされる。--no-length-filtrationを付けるとリード長トリミングはオフになる(default: 50:500)。
- 低複雑性領域のトリミングを行うには--enable-complexity-filtrationを付ける。トリミングに影響をパラメータ--min-complexity は、デフォルトでは0.3となっている。
- Atriaには、計算を高速化するためマルチスレッドとマルチコアの2つの並列方式が実装されている。どちらも有効にすることができる。マルチスレッドでは、1つのサンプルを処理するために複数のスレッドを使用する。デフォルトでは--threads 8となっている。マルチコアは、複数のサンプルを同時に処理する。デフォルトでは無効になっている。4つ並列にするには --procs 4をつける。
非圧縮のfastq、もしくはgzipかbgzip圧縮されたfastqを指定する。
ここではペアエンドfastq.gzを指定した。Phredクオリティ20でトリミングする。5'末端1塩基をトリムする(-c 1 )。poly Gトリミングも行う(--polyG)。低複雑性領域のトリミングはオンにする(--enable-complexity-filtration)。デフォルトでペアエンドのオーバーラップ領域の塩基の修復とNテイルのトリミングは自動でオンになっている。出力はoutdirに保存。-oを指定しないとカレントパスに出力される。8スレッド指定(-t 8)。
atria -r R1.fastq.gz -R R2.fastq.gz -o out_dir -a SEQ1 -A SEQ2 -t 8 -c 1 --polyG -q 20
- -r, --read1 input read 1 fastq file(s), or single-end fastq files
- -R, --read2 input read 2 fastq file(s) (paired with R1-FASTQ)
- -o, --output-dir store output files and stats to PATH (default: current working directory)
- -g, --compress AUTO|NO|GZ|GZIP|BZ2|BZIP2 compression methods for output files (AUTO: same as input, NO: no compression, GZ|GZIP: gzip with `pigz`, BZ2|BZIP2: bzip2 with `pbzip2`) (default: "AUTO")
- -a, --adapter1 SEQ read 1 adapter (default: "AGATCGGAAGAGCACACGTCTGAACTCCAGTCA")
- -A, --adapter2 SEQ read 2 adapter (default: "AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGT")
- -t, --threads INT use INT threads to process one sample (multi-threading parallel). (type: Int64, default: 1)
- -p, --procs INT process at most INT samples at the same time (multi-core parallel) (default: "1")
- --log2-chunk-size INDEX read at most 2^INDEX bits each time. Suggest to process 200,000 reads each time. Reduce INDEX to lower the memory usage. (type: Int64, default: 26)
- --quality-format FORMAT the format of the quality score (Illumina1.3, Illumina1.8, Sanger, Illumina1.5, Solexa); or the ASCII number when quality score == 0 (default: "33")
- --check-identifier check whether the identifiers of r1 and r2 are the same
- -C, --clip-after INT hard clip the 3' tails to contain only INT bases. 0 to disable. (type: Int64, default: 0)
- -c, --clip5 INT remove the first INT bases from 5' end. (type: Int64, default: 0)
poly X tail trimming:
- --polyG enable trimming poly G tails
- --polyT enable trimming poly T tails
- --polyA enable trimming poly A tails
- --polyC enable trimming poly C tails
- --poly-length the minimum length of poly X (type: Int64, default: 10)
実行すると標準出力に条件が表示される。確認しておく。
┌ Info: ATRIA TRIMMERS AND FILTERS
│ adapter_trimming = true
│ consensus_calling = true
│ hard_clip_3_end = true
│ hard_clip_5_end = true
│ quality_trimming = true
│ tail_N_trimming = true
│ max_N_filtering = true
└ length_filtering = true
chunkごとにパスした(ペアの)リード数が表示されていく。
[ Info: Cycle 1: processed 199657 read pairs (199657 in total), in which 161278 passed filtration (161278 in total). (0/0 reads copied)
[ Info: Cycle 2: processed 200324 read pairs (399981 in total), in which 164354 passed filtration (325632 in total). (667/667 reads copied)
[ Info: Cycle 3: processed 200324 read pairs (600305 in total), in which 167484 passed filtration (493116 in total). (667/1334 reads copied)
[ Info: Cycle 4: processed 200324 read pairs (800629 in total), in which 169979 passed filtration (663095 in total). (667/2001 reads copied)
[ Info: Cycle 5: processed 200324 read pairs (1000953 in total), in which 167820 passed filtration (830915 in total). (667/2668 reads copied)
[ Info: Cycle 6: processed 200324 read pairs (1201277 in total), in which 167874 passed filtration (998789 in total). (667/3335 reads copied)
[ Info: Cycle 7: processed 200324 read pairs (1401601 in total), in which 176154 passed filtration (1174943 in total). (667/4002 reads copied)
[ Info: Cycle 8: processed 200324 read pairs (1601925 in total), in which 178092 passed filtration (1353035 in total). (667/4669 reads copied)
[ Info: Cycle 9: processed 200324 read pairs (1802249 in total), in which 173586 passed filtration (1526621 in total). (667/5336 reads copied)
[ Info: Cycle 10: processed 200324 read pairs (2002573 in total), in which 171527 passed filtration (1698148 in total). (667/6003 reads copied)
[ Info: Cycle 11: processed 200324 read pairs (2202897 in total), in which 170582 passed filtration (1868730 in total). (667/6670 reads copied)
[ Info: Cycle 12: processed 200324 read pairs (2403221 in total), in which 173638 passed filtration (2042368 in total). (667/7337 reads copied)
[ Info: Cycle 13: processed 200324 read pairs (2603545 in total), in which 176535 passed filtration (2218903 in total). (667/8004 reads copied)
[ Info: Cycle 14: processed 200324 read pairs (2803869 in total), in which 177761 passed filtration (2396664 in total). (667/8671 reads copied)
[ Info: Cycle 15: processed 200324 read pairs (3004193 in total), in which 171773 passed filtration (2568437 in total). (667/9338 reads copied)
[ Info: Cycle 16: processed 200324 read pairs (3204517 in total), in which 171264 passed filtration (2739701 in total). (667/10005 reads copied)
[ Info: Cycle 17: processed 200324 read pairs (3404841 in total), in which 170401 passed filtration (2910102 in total). (667/10672 reads copied)
[ Info: Cycle 18: processed 200324 read pairs (3605165 in total), in which 170968 passed filtration (3081070 in total). (667/11339 reads copied)
[ Info: Cycle 19: processed 200324 read pairs (3805489 in total), in which 176159 passed filtration (3257229 in total). (667/12006 reads copied)
[ Info: Cycle 20: processed 200324 read pairs (4005813 in total), in which 176233 passed filtration (3433462 in total). (667/12673 reads copied)
[ Info: Cycle 21: processed 200324 read pairs (4206137 in total), in which 171687 passed filtration (3605149 in total). (667/13340 reads copied)
[ Info: Cycle 22: processed 200324 read pairs (4406461 in total), in which 169872 passed filtration (3775021 in total). (667/14007 reads copied)
[ Info: Cycle 23: processed 200324 read pairs (4606785 in total), in which 161197 passed filtration (3936218 in total). (667/14674 reads copied)
[ Info: Cycle 24: processed 200324 read pairs (4807109 in total), in which 168067 passed filtration (4104285 in total). (667/15341 reads copied)
出力例

アダプターが不明な時は、--detect-adapterを付けてAtriaをランし、まずアダプター配列を確認する(古いバージョンではこのオプションはない)。
atria -r R1.fastq.gz -R R2.fastq.gz --detect-adapter
リード全部は見ないので数秒で終わる。fastqは出力されない。
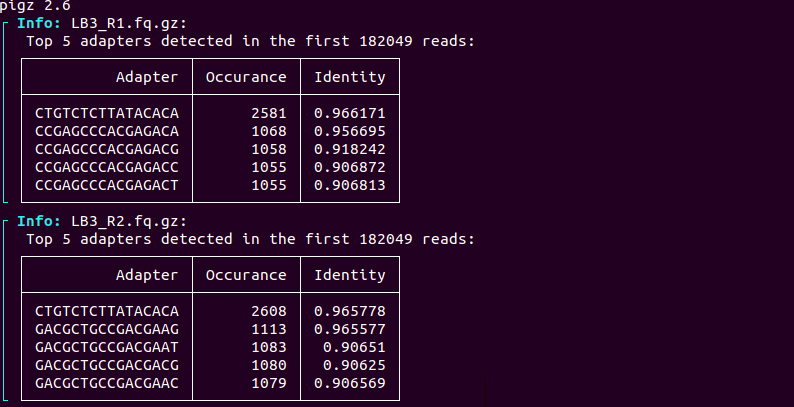
主要なアダプター配列が分かったら、-aと-AでR1とR2のアダプター塩基配列を指定してAtriaをランする。
より高速化するには、--procs をつけて複数のサンプルを並列処理する。ここでは4つずつ。1つあたり4スレッド指定。
atria -r sample*_R1.fastq.gz -R sample*_R2.fastq.gz --procs 4 -o out_dir -a SEQ1 -A SEQ2 -t 4 -q 20 -c 1 -C 1 --polyG
メモリが十分利用できる環境で、かつI/Oが高速なディスクを使っているなら、かなり高速化できる。Optane SSD上で試した時は、1つずつの時より4倍ほど早く終わった(リニアに高速化した)。メモリが足りないなら--log2-chunk-size <INT>を指定する。INTは23~25の整数。
引用
Atria: An Ultra-fast and Accurate Trimmer for Adapter and Quality Trimming
Jiacheng Chuan, Aiguo Zhou, Lawrence Richard Hale, Miao He, Xiang Li
Gigabyte, 1, 2021 https://doi.org/10.46471/gigabyte.31
関連