糖質活性酵素(CAZymes)は、様々な生物によって作られ、複雑な糖質代謝を担っている。バイオエネルギー、マイクロバイオーム、栄養、農業、地球規模の炭素循環におけるCAZymesの重要性から、CAZymesのゲノムマイニングは(メタ)ゲノムプロジェクトにおけるルーチンデータ解析となっている。2012年にCAZymeアノテーションを自動化するためのオンラインWebサーバーとしてdbCANが提供され、2018年には複数のツールを組み合わせてCAZymeアノテーションを改善するためのメタサーバーとしてdbCAN2(https://bcb.unl.edu/dbCAN2)に発展した。dbCAN2には(メタ)ゲノム中のCAZyme gene clusters (CGCs)を識別するツールCGC-Finderも含まれている。今回dbCAN3に更新し、以下の新機能とコンポーネントを追加した: (i) CAZymeサブファミリーレベルの基質予測のためのプロファイル隠れマルコフモデルデータベース(HMMdb)としてのdbCAN-sub 、(ii) CGCレベルでの基質予測のために、dbCAN-PULデータベースの既知の糖鎖サブステートを持つ実験的に特徴付けられたpolysaccharide utilization loci (PULs) の検索、 (iii) dbCAN-subから予測された基質を持つすべてのCAZymをCGCレベルの基質予測に考慮する多数決法の採用、 (iv) ウェブサイト上での基質予測結果のデータ閲覧および可視化の改善。以上、dbCAN3は、dbCAN2の機能をすべて受け継ぐだけでなく、糖鎖基質予測のための3つの新しい手法を統合している。
help
https://bcb.unl.edu/dbCAN2/help.php
https://bcb.unl.edu/dbCAN2/にアクセスする。
Annotateを選択。
配列を入力する。dbCAN2と同様、原核生物のゲノム(fnaファイル)またはメタゲノムアセンブリゲノム(MAGs)、真核生物ゲノムではタンパク質配列(faaファイル)を使用できる。

探索にはHMMERやDIAMONDなどが選べ、感度が異なっている。CGC-Finderを選択すると遺伝子位置ファイルのアップロードボタンが表示される。遺伝子位置ファイルはGFFかBED形式(例)をアップロードする必要がある。FASTAファイルで使用される遺伝子IDは、BED/GFFファイルのものと完全に一致する必要がある。GFFファイルを使用する場合、タイプカラムに'CDS'が含まれる行のみが考慮される。GFFファイルでは遺伝子IDはNameタグのあるnotes欄に記載する。Nameタグがない場合、遺伝子IDはIDタグの中に記載する。
それぞれのツールの感度(マニュアルより)
- DIAMOND: E-Value < 1e-102, hits per query (-k) = 1.
- HMMER(dbCAN): E-Value < 1e-15, coverage > 0.35.
- HMMER(dbCAN-sub): E-Value < 1e-15, coverage > 0.35.
- CGC-Finder: Distance = 2, signature genes = CAZyme+TC. DIAMOND for TC and TF: E Value = 1e-10, hits per query (-k) = 1. CAZymes are defined as those predicted by at least 2 tools.
- Prodigal: Defaults.
- CGC substrate prediction based on dbCAN-PUL search: unique genes homologous >= 2, bitscore >= 50, total signature pairs >= 2 and CAZyme signature pair >= 1.
- CGC substrate prediction based on eCAMI-subfam: major voting score >= 2.
(マニュアルより)CGCとは、CAZyme遺伝子、トランスポーター/TC遺伝子(TCDBで検索して予測)、転写因子/TF遺伝子(collectf DB, RegulonDB, DBTBSで検索して予測)を少なくとも1つ含むゲノム領域と定義する。その根拠は、CAZymesはしばしば互いに、また他の重要な遺伝子(TFsや糖輸送体など)と協力して、様々な高度に複雑な糖質を相乗的に分解・合成しているためである。なぜCGCの同定が面白いかについては、著者らの論文を参照されたい。
example配列を指定した。HMMERが選択されている。

メールアドレスを入力するとジョブ完了時にメールが届く。
example result
Overview;このタブでは、各タンパク質がどのツールでCAZymesのアノテーションが付けられたか、どのCAZyファミリーで注釈を付けられたかなどが表示される。またCAZymesの基質へのリンクやシグナルペプチドの予測結果も表示される。
 2つ以上のツールで予測されたタンパク質はCAZymeのアノテーション性能がより優れていることが示唆されている。EC番号はBRENDAに、HMMERとDIAMONDの結果はCAZY DBにリンクしている。アノテーションでCE4(103-271)など書かれている時の()はドメインのポジション。
2つ以上のツールで予測されたタンパク質はCAZymeのアノテーション性能がより優れていることが示唆されている。EC番号はBRENDAに、HMMERとDIAMONDの結果はCAZY DBにリンクしている。アノテーションでCE4(103-271)など書かれている時の()はドメインのポジション。
gene IDをクリックするとドメインの図が表示される。このタンパク質はCE4とGH153がアノテートされている。

HMMER dbCAN;このタブでは、MMERの実行結果とdbCANデータベースの結果を表示している。タブの上部から全出力をダウンロードできる。

CGC-Finder;このタブはCGC-Finderを実行した場合のみ表示される。CGC-Finderの出力となる。CAZyme gene clusters (CGCs)のそれぞれのクラスタを構成する遺伝子数などが表示されている。CGCは、上でも書いたが、少なくとも1つのCAZyme遺伝子、1つのトランスポーター/TC遺伝子(TCDBとの検索により予測)、1つの転写因子/TF遺伝子(collectf DB, RegulonDB, DBTBSとの検索により予測)を含むゲノム領域と定義されている。パラメータを変えて再度探索することができるようにもなっている。
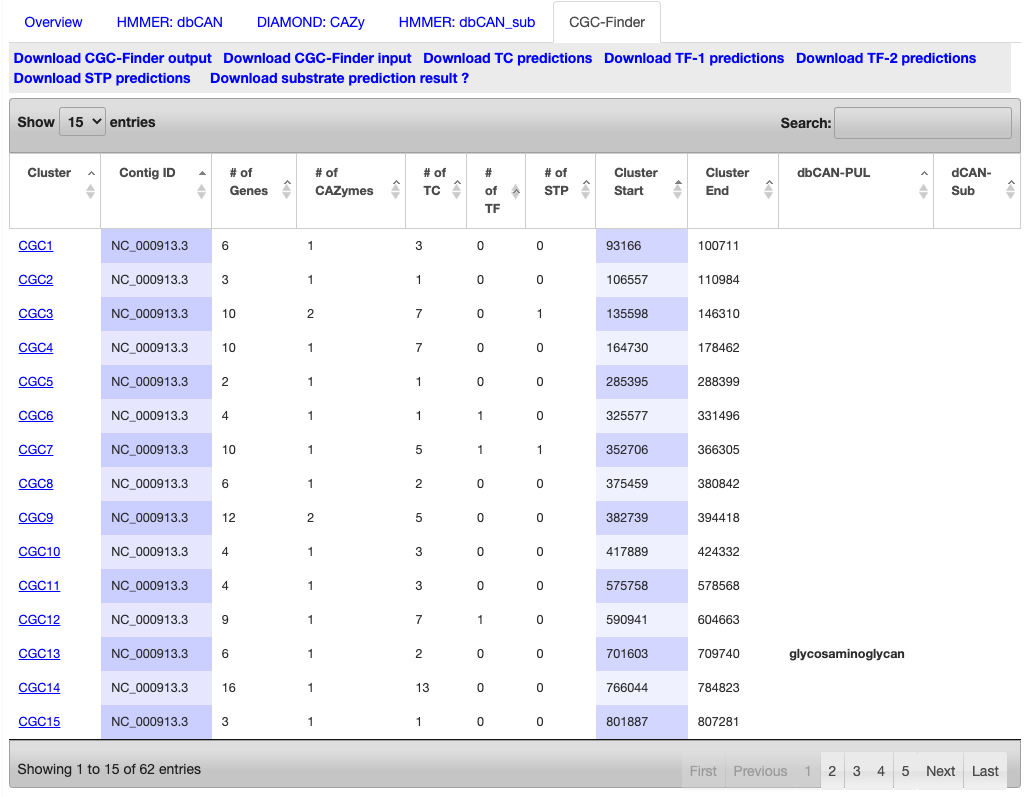

Rerunの詳細はhelpで確認してください。各パラメータについて詳しく説明されています。
引用
dbCAN3: automated carbohydrate-active enzyme and substrate annotation
Jinfang Zheng, Qiwei Ge, Yuchen Yan, Xinpeng Zhang, Le Huang, Yanbin Yin Author Notes
Nucleic Acids Research, Published: 01 May 2023
関連
