2020 7/2 誤字修正
2021 4/27 v5の論文リンク追加
2022 8/27追記
2024/04/21 v6論文追加
系統樹は、生物学やその他の科学分野において重要なツールであり、様々なデータタイプのコンテキスト化としても機能している。このことは、このような系統樹を作成するためのツールが頻繁に使われていることからもわかる(MEGA, (ref.2))。このようなツリーの可視化は、長年にわたって様々なソフトウェアツールによってカバーされてきたが、iTOL(ref.5)では、様々なタイプの追加データを含むツリーのアノテーションを導入した。現在では、ETE toolkit(ref.6)、Dendroscope(ref.7)、Evolview(ref.8)などのように、オンラインでもスタンドアロンでも、様々なソフトウェアパッケージがツリーのアノテーション機能を提供している。ここでは、iTOLの機能を拡張・合理化し、より強力で使いやすいものにしたiTOLの最近の開発について報告する。
iTOLは、最新のWebブラウザからアクセス可能なオンラインツールである。ツリー表示エンジンは純粋なJavascriptで実装されており、可視化のためにHTML5 Canvasを使用している。
iTOLは、一般的に使用されている系統樹のフォーマットをサポートしている。Newick、Nexus、phyloXMLである。EPAとpplacerによって作成されたPhylogenetic placements filesもサポートしている。現在のバージョンでは、QIIME 2 のツリーとアノテーションファイルのサポートが導入されている。(一部略)
iTOLは、他の系統樹ビューアで利用可能なほとんどの一般的な機能を提供する。iTOL v4では、標準的な表示形式(rectangular, circular and unrooted)に加えて、系統樹の斜め表示モードをサポートしている。ツリーは様々な方法で操作することができ、基本的な編集機能では、単一ノードやクレード全体の削除や移動をインタラクティブに行うことができる。また、様々なパラメータ(関連するブートストラップ値や平均枝長距離など)に基づいて、手動または自動でブランチをカットしたり、縮めたりすることができる。また、任意のノードで手動で、または中点ルート法を使って自動的にツリーの re-rootができる。ツリーのleavesは、手動または自動で様々な方法でソートすることができる。
現在のバージョンでは、いくつかの新しいアノテーション機能が導入されており、個々の表示要素に対するユーザーのコントロールが拡張され、4つの新しいデータセットタイプが追加されている(論文図2)。
iTOLでは、ツリー内の個々のノードやラベルに対して、個々のスタイルや色をサポートしている。現在のバージョンでは、ユーザーインターフェース全体でUTF-8文字セットを完全にサポートし、Google Web Fontsリストから任意のフォントを使用できるようになり、さまざまなフォントやフォントスタイルのサポートが大幅に拡張されている。さらに、任意のテキストラベルの背景色を独立して変更できる。
https://twitter.com/search?q=iTOL%20v4&src=typed_query
video tutorial
https://itol.embl.de/video_tutorial.cgi
help
gallary
https://itol.embl.de/gallery.cgi
テストデータ
helpからexample annotation data(ダイレクトリンク)をダウンロードできる。その中にツリーファイルも含まれる。ここではこのデモデータを使う。
https://itol.embl.de/ にアクセスする。気づかなかったが、いつの間にかv5v6になっている。=> 2024/12/02 v7に更新
写真中央下のUpload treeボタン、または上のメニューからtreeファイルをアップロードする。Newick、Nexus、phyloXMLフォーマットやQiime2のツリーファイル等に対応している。
アカウントを作ってloginしておくと複数ツリーを管理でき便利になる。loginしない場合、この管理画面は表示されずそのままツリーが表示される。

1、基本操作
exampleデータを読み込んだ。管理画面でツリーファイルを選ぶとツリーが視覚化される。画面はインタラクティブにマウスホイールで拡大縮小したり、ドラッグで移動できる。これらの操作は左端のボタンでも可能になっている。

右のウィンドウからツリーの表示方法を変更できる。

DisplayモードはdefaultではCircularになっている。

Normal。長方形の形状の普通の系統樹。

Unrooted

Normal、Slanted => ON

Normal、Dashed line => 3、Branch lines => 2

Labels => At tips
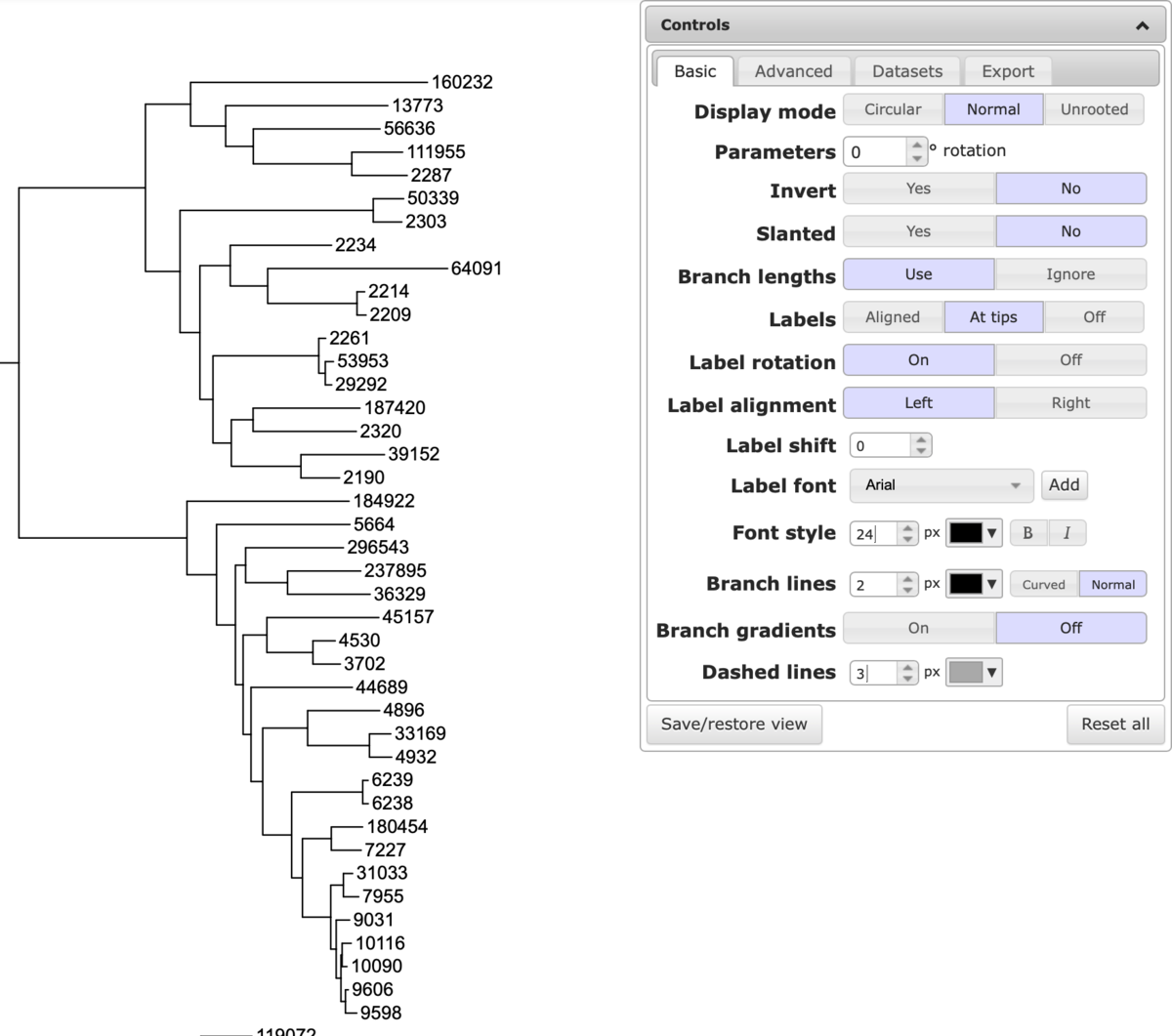
Labels => At tips & Label shift 60、Label Font => Times New Roman。

Advancedの機能も簡単に確認していく。
Branch length => Display、Fout size 16、round to 2 decimals(小数点以下の表示桁数)

Branch length => Display、Display as age => ON。

年齢表示オプションを選択すると、枝の長さの値の代わりにノードの年齢が表示される。ツリー内で最も遠いノードの年齢はゼロで、ツリーのルートに向かって年齢が上昇していく。
ブートストラップ値は4つの方法で表示できる。 symbol modeのほか、text、color、widthモードになる。
bootstrap => Display & symbol mode、Legend On。

ブートストラップ値を表示できるのはツリーにブートストラップ値の情報が含まれている場合のみ。
bootstrap => Display & text mode、Font size 13、position on branch 30。

bootstrap => Display & color mode、Legend On。Display range 50-100。
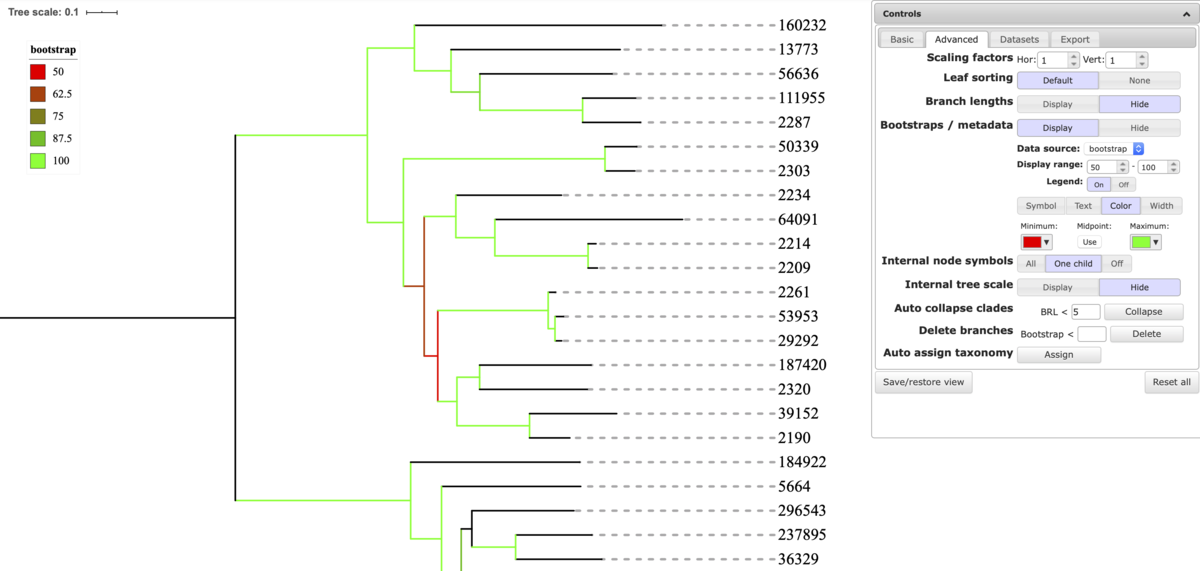
bootstrap => Display & width mode、max width 3、min width 1。

bootstrap値が50以下の信頼性が低い枝は消す(実験によって閾値は変わる)。枝長が短いノード(<0.1)をcollapseする。
Auto collapse clades < 0.2 、Delete branches < 50。

collapseしているとcollapseした部分の形状を選べるメニューが表示される。
Collapsed nodes => circle。

Collapsedした枝はクリックすることで再び展開できる。

Internal tree scale => Display、interval1 => 0.2 & dark blue、font size 27、Set root to 1997 & Scaling factor 10。

タイムスケールモードでは、ツリーのスケールをカスタマイズして、枝の長さではなく他の値を表示することができる(データに合わせた適切な方法でツリーが推定・校正されている時にだけ利用できるオプションであることに注意する)
対応するノードIDを使って系統樹を描画している場合、右下のAuto aasign taxnomyボタンを押し、Reset treeを一度行うことで対応する名前に変更できる。ここでは数値が種名に変わる。

メニュー左下のSave/restore ボタンで名前をつけて設定を保存する。現在の設定をデフォルト設定として利用できるようになる。

Save as => 名前を決めて保存する。

設定はいつでも呼び出せる。非常に便利な機能。

枝をクリックすることで、クレード全体/特定のノードのフォントカラーやバックグラウンドを変更できる。
Color => Set clade color

赤を選択した。

選択したクレードだけ赤になった。

赤のクレードの枝を実線から破線に変更した。
Stype => Clade => Dashed line。

Leaf labels => set labels colorで色の変更。
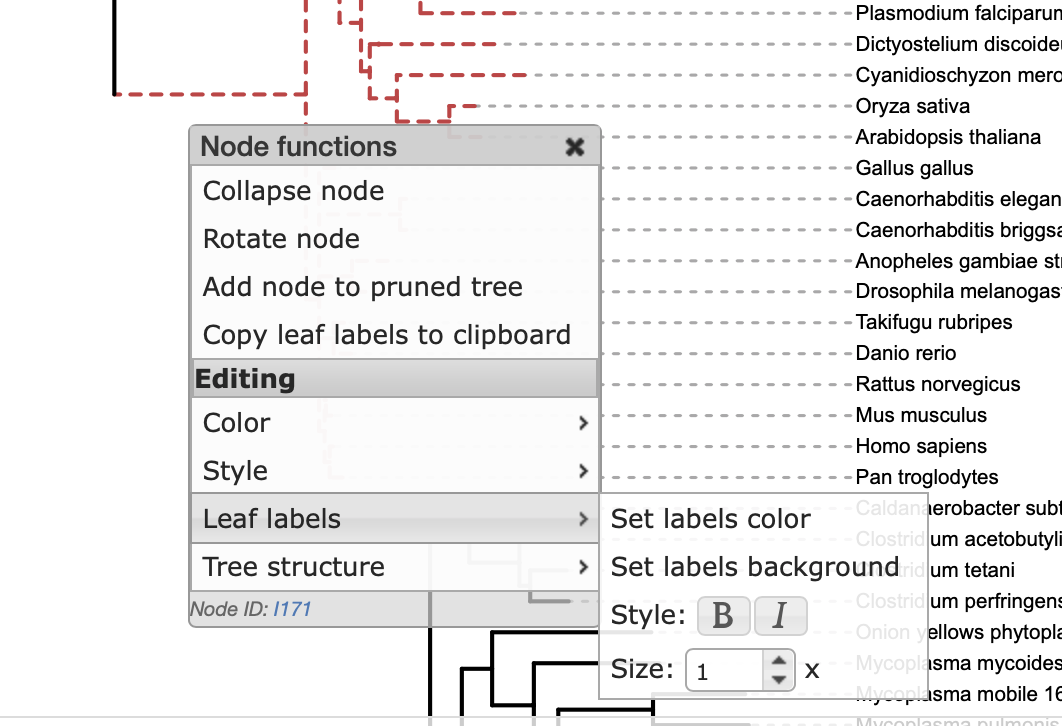
フォントを緑にして、太字、size 3にした。

Leaf labels => set labels backgroundでフォントの背景色の変更。

背景を薄い緑にした。

背景を整えると、tree of lifeの環状ツリーのようにクレードごとの統一性が出る。
追記
クレード全体の背景を変えるにはColor => New color groupで色を設定する。上の方法だとノード間に色がない部分が残る。

不要なクレードを削除する。よく考えて行うこと。
Tree structure => Delete clade。

Copy leaf labels - 選択したクレードのラベルだけコピーする

テキストエディタに貼り付けた。
Rerot tree here

2023/08追記
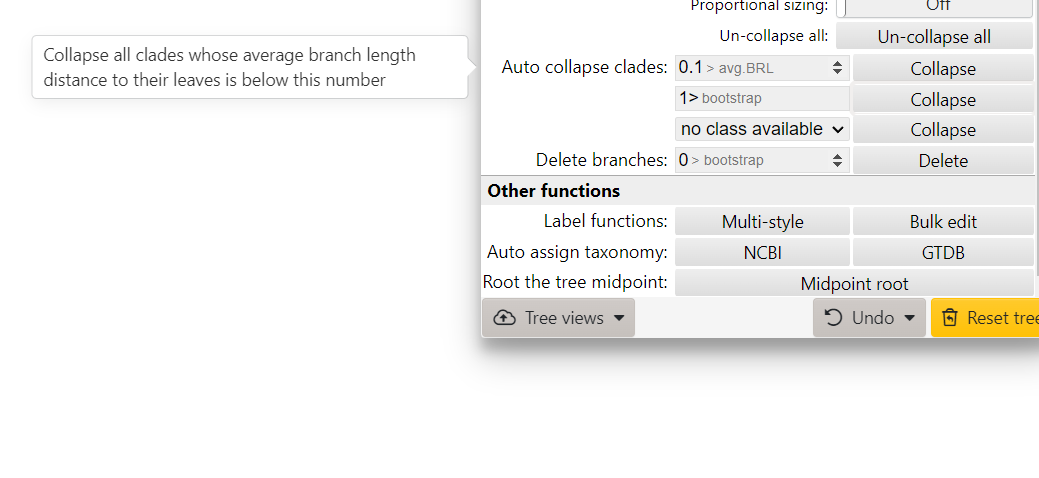
advancedのメニュー下から、指定した枝長以内の姉妹系統を一括してcollapseしたり、特定のbootstrap値以下、以上の枝を一括してcollapseする、learve数が指定値以下のノードを一括削除するなどの機能が利用できるようになっています。collapseはun-collapse allボタンで解除できる。
2、応用操作
ここからは一括設定や注釈ファイルの表示が可能なconfigファイルを使う流れを確認する。使うのは上のexampleファイル。中に様々な設定ファイルが入っているので、これを使ってiTOL v4で表現可能な注釈情報を示す。

exampleフォルダの中身。
実際に使用するには、注釈ファイルを表示中の系統樹の上にドラッグアンドドロップするだけでよい。
試しに colors_tol.txtを画面上にドラッグするとすぐに注釈が反映された。このcolors_tol.txtに記載されているのはノードのフォントサイズや色設定になるため、それらが反映されている。

次はranges.txtを読み込んだ。ranges.txtはそのままレンジを一括指定する。形状はcircularモードにしている。

追記
rangeでクレード全体の背景に色をつけるには、葉の名前の位置をAt tipsにするか、下の画像のように、rangesのウィンドウの一番下のボタンで、CladeかFullを選択する。

tol_alignment.txt。マルチプルシーケンスアラインメントを系統樹の隣に表示する。特定のタンパク質や遺伝子の系統樹で使う。normalモードでしか表示されない。

tol_binary.txt

tol_binary2.txt

tol_binary.txtの後に追加した。複数メタデータを追加した場合、追加された順に内側から外側に向かって順番に表現される。
tol_boxplot.txt
#各ノードには5つの数値の要約が定義され、その後に任意の数の極値が定義されていなければならない。すなわち、カンマ区切りなら
となっている必要がある。

tol_color_strip.txt

tol_domains.txt タンパク質ドメインなどを表示する。
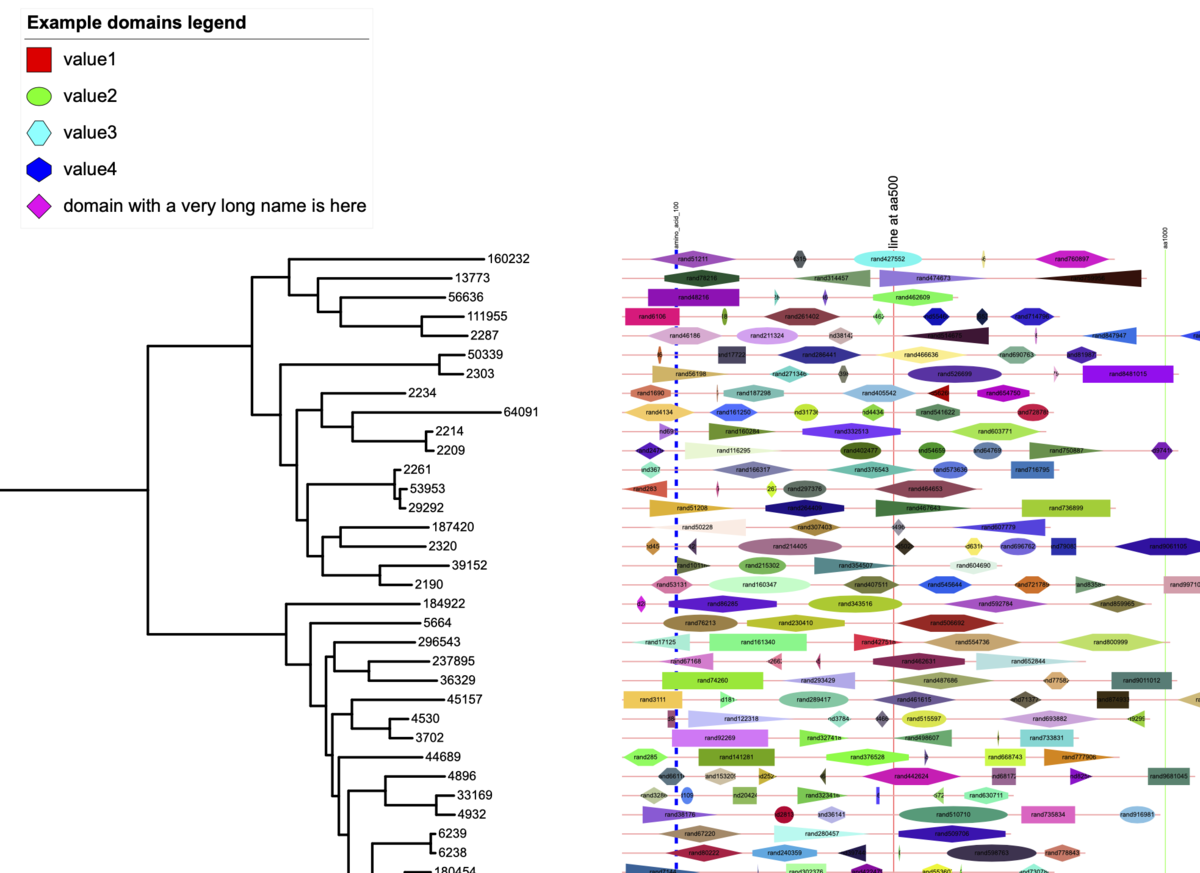
tol_external_shapes.txt

tol_heatmap1.txt

tol_linechart-sine.txt

tol_multibar10.txt

tol_pies1.txt

tol_connections_leaves.txt

複数の設定ファイルを読み込み

左のメニューボタンの一番下には画面に直接半透明の四角や楕円を描いたりするmanual annotationボタンがある。しかしあまり使い勝手は良くなく、出力後にイラストレーターなどで編集する方が簡単に感じた。
ではexampleのtol_binary.txtを例に、実際にはどのようなファイルを用意すれば良いのかを説明する。このtol_binary.txtはバイナリ(1/0)表現可能なメタデータを読み込むときに使う。
tol_binary.txtの中身


このファイルを、iTOL v4のexample treeを表示させた状態で画面にドラッグ&ドロップすると、系統樹横に11個のメタデータが表示される。

これをどう制御しているのか説明する。まずtol_binary.txtの5-6行目。読み込むバイナリ値のセパレータはコンマとする。セパレータをコンマからタブに変える場合、5行目のコメントアウトを消し、7行目をコメントアウトする。

24-26行名は重要。11個のメタデータを追加したいなら、24-26行名それぞれ11指定する必要がある。例えば25行目だけ10個しか指定していないなど、ズレがあると何も表示されない。

24行目はラベル。ユニークな名前をメタデータの順番に11個指定する。

25行目は色。16進数カラーコード指定。メタデータの順番に11個指定する。16進数カラーコードはオンラインのカラーピッカーサイトやグラデーション表示計算サイト(例)を使用して下さい。

26行目はプロットの形状。19-23行目に記載されているように1-5の数値で形が決まる。下だとメタデータの一番内側は長方形。次は右向き矢印。メタデータの順番に11個指定する。

そのほか細かい指定があるが、テキスト自体コメントをつけながら書かれているので、読み込むexampleファイルを読めばわかるようになっている。
バイナリメタデータは最後に記載する。

系統樹のノード155864を拡大して示した。系統樹ではこのように表示されているが、

tol_binary.txtをみると、このノード155864は0,1,1,1,0,1,0,-1,0,1となっている。

11個のマークの形状はtol_binary.txtの26行目で決定しているので、この72行目で指定しているのはマークを表示するかしないかである。1は表示、0は白抜きで枠のみ表示、-1は非表示と決まっている。
結果をみると、左から1番目(◁)のマークは0なので白抜きで表示、2番目(◀︎)は1なので表示と、確かにそうなっている。8番目は-1なのでこれだけ非表示になっている。

(繰り返しになるがマークの形状は26行目で指定、全てのサンプルに同じ形状が反映される)
このようにしてexampleのバイナリメタデータは表現されている。

読み込んだメタデータはDatasetからシンボルの間隔やサイズを変更できる。

configファイルに大半の情報を記載しているため、間違った時も設定をresetして再読み込みするだけでリカバー可能。メタデータのルール詳細は、上にリンクを張ったhelpを参照して下さい。
結果はPDFやSVGなどで出力できる。
 系統樹を保存するだけでなくシェアする機能もある。
系統樹を保存するだけでなくシェアする機能もある。
exampleデータやgallaryファイルを数時間さわれば、大体の操作方法はマスターできます。テスト時は数千ノードあっても機能しました。バージョンアップを重ねているだけあってユーザーフレンドリな素晴らしいphylogenetic tree viewerになっています。
# バグなのか仕様なのか不明ですが、Newickフォーマットのツリーファイルを読み込ませると一部のleave が抜けていたデータがありました。いったんFigtreeに読み込ませて全データをNEXUS形式で出力し、それを使うと一部のleave の抜けは無くなりました。
引用
Interactive Tree of Life (iTOL) v6: recent updates to the phylogenetic tree display and annotation tool
Ivica Letunic, Peer Bork
Nucleic Acids Research, Published: 13 April 2024
Interactive Tree Of Life (iTOL) v4: recent updates and new developments
Ivica Letunic, Peer Bork
Nucleic Acids Research, Volume 47, Issue W1, 02 July 2019
Interactive Tree of Life (iTOL) v3: An Online Tool for the Display and Annotation of Phylogenetic and Other Trees
Ivica Letunic, Peer Bork
Nucleic Acids Res. 2016 Jul 8;44(W1):W242-5
Interactive Tree Of Life v2: Online Annotation and Display of Phylogenetic Trees Made Easy
Ivica Letunic , Peer Bork
Nucleic Acids Res. 2011 Jul;39
Interactive Tree Of Life (iTOL): An Online Tool for Phylogenetic Tree Display and Annotation
Ivica Letunic, Peer Bork
Bioinformatics. 2007 Jan 1;23(1):127-8
参考
2021 4/27
SERVER ACCESSより
iTOLは自立したツールであり、実質的な公的資金を得ずに維持・拡張されてきた。アクティブなユーザー数とアップロードされる樹木の数が増え続けていることを考慮し、iTOLを維持・発展させ、必要なストレージとCPUパワーを提供し、ユーザーベースにタイムリーな技術サポートを提供するための持続可能なモデルを模索してきた。バージョン5では、ツリーのアノテーション機能は無料で利用できるが、iTOLのアカウント管理機能の一部は、アクティブなサブスクリプションが必要となった。iTOLサーバーへのツリーアノテーションの保存とバッチアップロードモードを除き、ほとんどのユーザーアカウント管理機能は無料で利用できる。ユーザーがアップロードしたツリーやアノテーションは、会員登録の有無にかかわらず、いつでもアクセスできる。
iTOL subscriptions

