2020 3/21 コマンドの修正と結果追記、タイトル修正、誤字修正
2020 3/23 誤字修正
ゲノムシーケンシングは、現代の分子生物学の不可欠な部分となっている。ただし、利用可能な分析方法の大半は、染色体レベルのリファレンスゲノムと詳細なアノテーションがすぐに利用できる確立されたモデル生物向けに設計されている。対照的に、非モデル生物のゲノムアセンブリは、断片化されている、不完全である、または存在しないことがよくある。さらに、モデル生物は通常、比較的複雑さが控えめで、通常、比較的遺伝的多様性が低く、リピートが少ない半数体または二倍体種でである。逆に、非モデル種はしばしば倍数性が高いか、ヘテロ接合率が高いため、分析が実質的に困難である。結果として、倍数体種または他の異常なゲノム構造を持つ種は、ゲノミクス研究の中で非常に過小評価されている。
これは、そのような研究から収集できる生物学的洞察の一般性を低下させる。倍数体は、特に植物や菌類の間で一般的であることが知られている。リンゴ、バナナ、ジャガイモ、イチゴ、小麦など人間の消費と使用に不可欠な多くの一般的な作物を含む開花植物の70%以上は倍数体である。より高い倍数性レベルは、多くの真菌種でも実証されている(ref.3, link)。動物の倍数体はこれらの他の分類群ほど一般的ではないが、多くの
種のカエル、魚、甲殻類、軟体動物、および多くの線虫を含み、珍しいものではない。倍数体作物の主要な害虫である線虫種も偶然ではあるが倍数体である。より一般的には、倍数体化イベントはゲノム進化に重要な結果をもたらす。したがって、倍数体がゲノムおよび種の進化にどのように影響するかを理解するには、断片化された倍数体ゲノムを分析するツールの開発が不可欠である。
倍数体ゲノムを分析する方法は、通常、リードを一倍体リファレンスにマッピングすることに依存している。ただし、完全な半数体のリファレンスを取得することは通常、困難な作業である。種の基本的なゲノムの特徴が不明な場合(サイズ、ヘテロ接合性、倍数性など)、ゲノムアセンブリにはさらに複雑なレイヤーがある。二倍体生物のコンテキストでは、ゲノムサイズとヘテロ接合または反復性とヘテロ接合を含む、アセンブリされていないシーケンシングリードから直接ゲノム特性を推定するためのいくつかの計算アプローチが開発された。ただし、これらのアプローチはいずれも倍数体ゲノムをモデル化しない。
以前に、GenomeScopeを導入した。これは、k-merスペクトラムとも呼ばれる、アセンブルされていないリードのk-merの統計分析を使用した、二倍体ゲノムのリファレンスフリー分析である。ここでは、GenomeScope 2.0を紹介する。GenomeScope2.0は、このアプローチを倍数体対応の混合モデルで拡張し、アセンブリされていないシーケンスデータからゲノムの特性を計算的に推測する。 GenomeScope 2.0は、負の混合二項分布をシーケンスデータのk-merスペクトルに適合させ、より高い倍数性レベルでk-merをキャプチャする追加コンポーネントを備えている。種の分析をさらに支援するために、非モデル生物ではしばしば未知である倍数性を推定するためのゲノム構造の視覚化技術であるSmudgeplotも開発している。これらのツールが、シミュレートされた倍数体ゲノムのアンサンブルといくつかの実際の倍数体ゲノムからのシーケンスデータを迅速かつ正確に分析することを示す(分析された11種の要約については、論文補足表1を参照)。これらのツールは、ゲノムアセンブリの評価と解釈を改善するために使用でき、倍数体または複雑なゲノムの将来の研究を大幅に支援する。
Interested in genome profiling? In this paper, @t_rhyker, @KamilSJaron & @mike_schatz present GenomeScope 2.0 and Smudgeplot for reference-free profiling of polyploid genomes. R codes and a web-enabled version of GenomeScope are providedhttps://t.co/fS3yEbVV4I
— Nature Communications (@NatureComms) 2020年3月18日
インストール
ubuntu18.04LTSでテストした。
KMCかjellyfishでk-merカウントする必要がある。ここではKMC3を導入。
conda install -c bioconda -y kmc
本体 Github
git clone https://github.com/tbenavi1/genomescope2.0.git
cd genomescope2.0/
mkdir ~/R_libs
echo "R_LIBS=~/R_libs/" >> ~/.Renviron
Rscript install.R
> ./genomescope.R -h
# ./genomescope.R -h
usage: ./genomescope.R [-h] [-v] [-i INPUT] [-o OUTPUT] [-p PLOIDY]
[-k KMER_LENGTH] [-n NAME_PREFIX] [-l LAMBDA]
[-m MAX_KMERCOV] [--verbose] [--no_unique_sequence]
[-t TOPOLOGY]
[--initial_repetitiveness INITIAL_REPETITIVENESS]
[--initial_heterozygosities INITIAL_HETEROZYGOSITIES]
[--transform_exp TRANSFORM_EXP] [--testing]
[--true_params TRUE_PARAMS] [--trace_flag]
[--num_rounds NUM_ROUNDS]
optional arguments:
-h, --help show this help message and exit
-v, --version print the version and exit
-i INPUT, --input INPUT
input histogram file
-o OUTPUT, --output OUTPUT
output directory name
-p PLOIDY, --ploidy PLOIDY
ploidy (1, 2, 3, 4, 5, or 6) for model to use [default
2]
-k KMER_LENGTH, --kmer_length KMER_LENGTH
kmer length used to calculate kmer spectra [default
21]
-n NAME_PREFIX, --name_prefix NAME_PREFIX
optional name_prefix for output files
-l LAMBDA, --lambda LAMBDA, --kcov LAMBDA, --kmercov LAMBDA
optional initial kmercov estimate for model to use
-m MAX_KMERCOV, --max_kmercov MAX_KMERCOV
optional maximum kmer coverage threshold (kmers with
coverage greater than max_kmercov are ignored by the
model)
--verbose optional flag to print messages during execution
--no_unique_sequence optional flag to turn off yellow unique sequence line
in plots
-t TOPOLOGY, --topology TOPOLOGY
ADVANCED: flag for topology for model to use
--initial_repetitiveness INITIAL_REPETITIVENESS
ADVANCED: flag to set initial value for repetitiveness
--initial_heterozygosities INITIAL_HETEROZYGOSITIES
ADVANCED: flag to set initial values for nucleotide
heterozygosity rates
--transform_exp TRANSFORM_EXP
ADVANCED: parameter for the exponent when fitting a
transformed (x**transform_exp*y vs. x) kmer histogram
[default 1]
--testing ADVANCED: flag to create testing.tsv file with model
parameters
--true_params TRUE_PARAMS
ADVANCED: flag to state true simulated parameters for
testing mode
--trace_flag ADVANCED: flag to turn on printing of iteration
progress of nlsLM function
--num_rounds NUM_ROUNDS
ADVANCED: parameter for the number of optimization
rounds
実行方法
1、k-merカウントする。KMCやjellyfishが使える。ここでは KMC3を使う。fastqはgzip圧縮されていても、解凍してあってもOK。リスト指定しない場合、ペアエンドなどのfastqはあらかじめ結合しておく(cat pair*.fq.gz > concatenated.fq.gz)。fastqの場合は”-fq”、fastaの場合は"-fa"フラグを立ててファイルを指定する。
mkdir working_dir
kmc -k21 -m32 -ci1 -fq input.fq.gz database working_dir
- -f<a/q/m> input in FASTA format (-fa), FASTQ format (-fq) or multi FASTA (-fm); default: FASTQ
- -ci exclude k-mers occurring less than <value> times (default: 2)
- -m max amount of RAM in GB (from 1 to 1024); default: 12
- -k k-mer length (k from 1 to 256; default: 25)
- -t total number of threads (default: no. of CPU cores)
database.kmc_preとdatabase.kmc_sufが出力される。21-merはラージゲノムにおいてもユニークな長さが期待できる十分な長さで、十分な頻度出現すると期待されるため推奨値となっている。半数体で10Gbを超すようなより大きなゲノムでは、より大きなk-mer値を検討する必要があるかもしれない。
2、histgramファイルを出力する。database.~はdatabaseと指定すれば認識する。
kmc_tools transform database histogram reads.histo -cx10000
- -cx exclude k-mers occurring more of than <value> times
3、genomescopeの実行。ここでは2倍体を指定。
./genomescope.R -i reads.histo -o output_dir2 -k 21 -p 2
- -k kmer length used to calculate kmer spectra [default 21]
- -i input histogram file
- -o output directory name
- -p ploidy (1, 2, 3, 4, 5, or 6) for model to use [default 2]
3の視覚化はオンラインでも行うことができる。使用したk-mer長、ploidy、primary peakの値などを記載してランする。
http://qb.cshl.edu/genomescope/genomescope2.0/
Rを使えない環境ならweb版は重宝します。コピー数の多いオルガネラゲノムのカバレッジピークが混ざると推定値に大きな誤差が出る可能性がある。よって、データによってはmax k-mer coverageを指定し、ハイコピーなオルガネラゲノムのピークを除いて計算する。
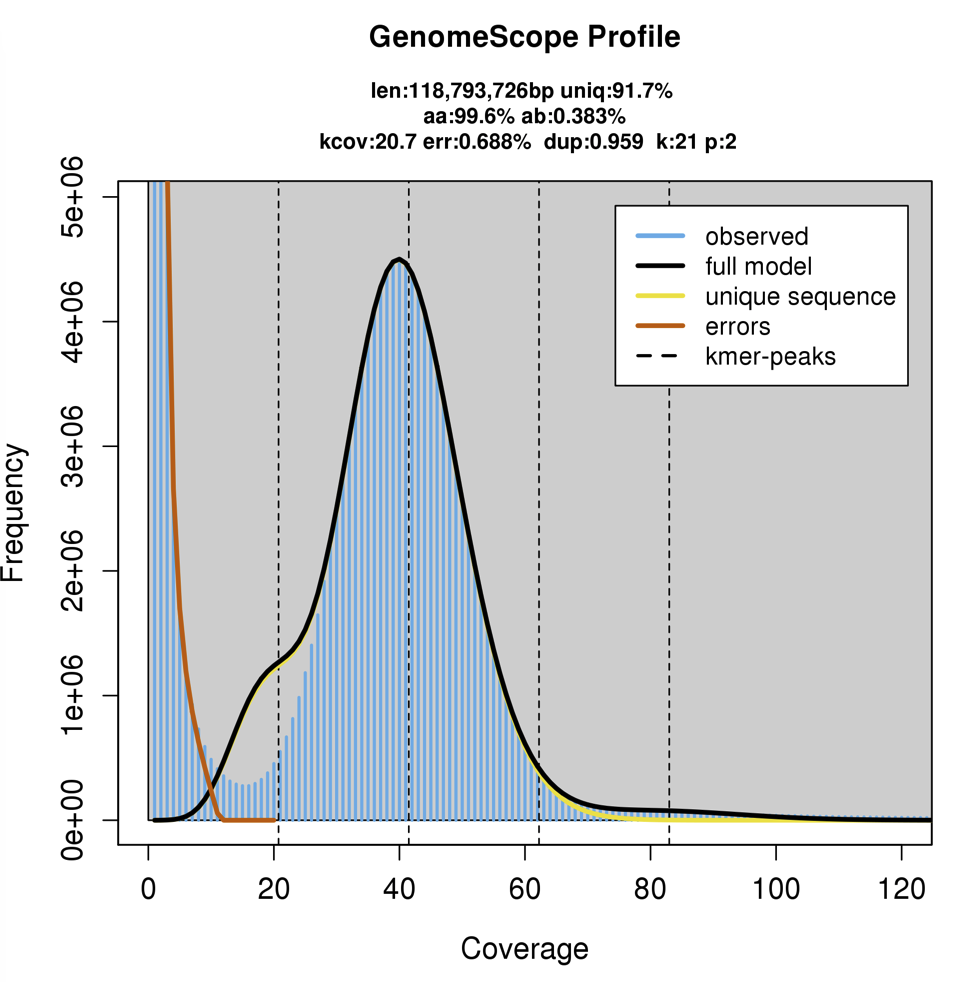
シロイヌナズナのillumina シーケンシングデータ(ERR2173372)を2倍体設定で分析してみた。論文の例ほど顕著ではないが、主要なピークの左側に肩があり、ヘテロ接合性の領域の配列と推測される(観測値にはない)。AA=99.6%、AB=0.383%だった。
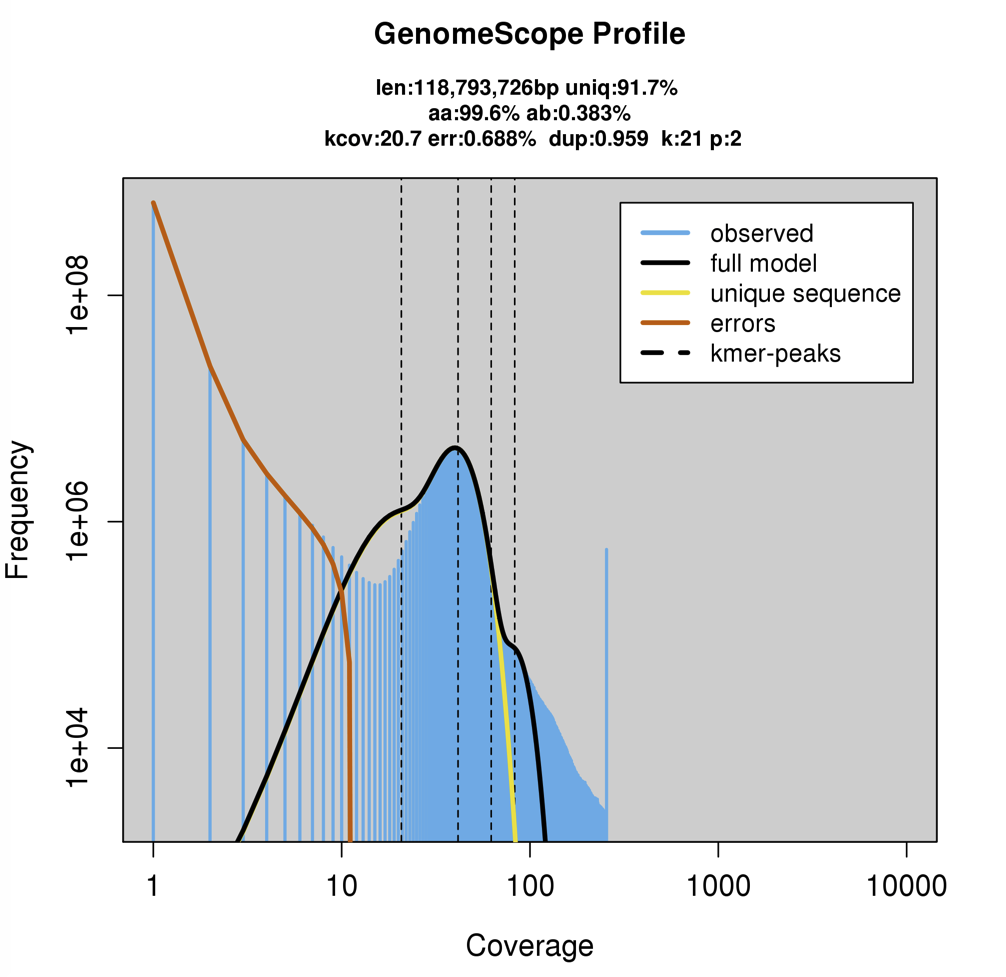
x軸が対数の図も出力される。unique sequence(黄色)外のモデルにフィッティングしきっていない右側の肩部分がrepettitive sequence由来ピークと思われる。
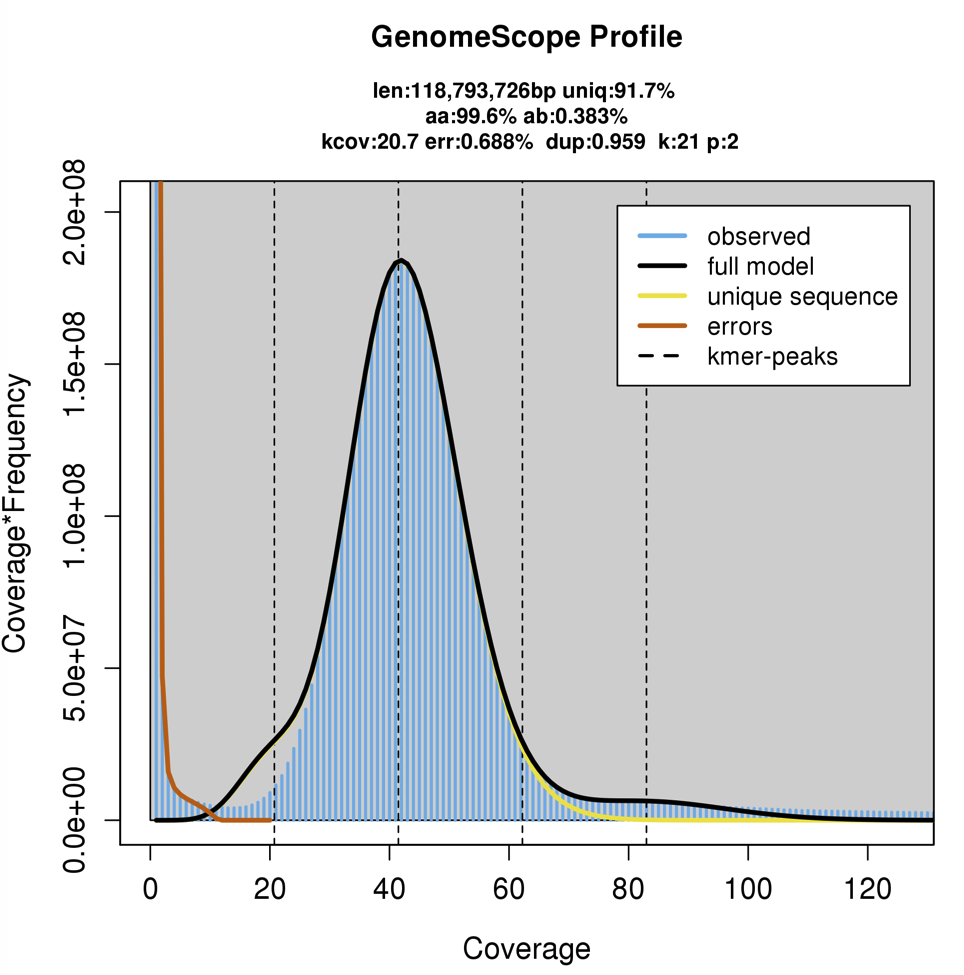
理論モデルへのフィッティング率も表示される。

Haploidゲノムサイズは117.6-Mbpとなった。
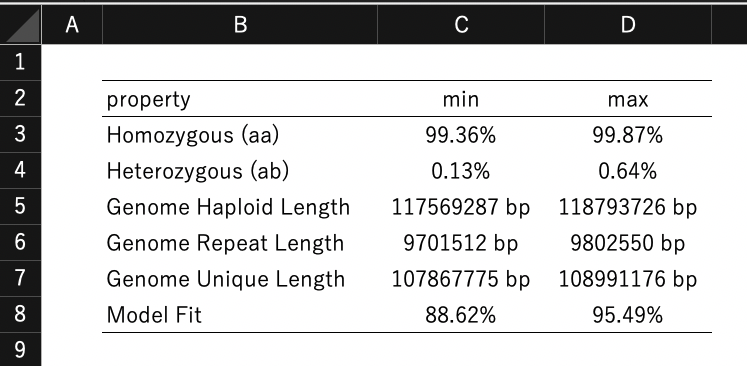
GenomeScope2を走らせる時に、何倍体かの指定を間違えると推定値が変わってしまう。そもそも倍数性が不明で、何倍体であるか、また異質倍数体か同質倍数体かを推測したいなら、先にsmudgeplotを使用する。
GenomeScope 2.0は6倍体ゲノムまでのゲノムサイズ推定を行うことができる。ただし、aneuploid cancer genomesなどの性染色体数が不均等なゲノムサイズの推定には適していない(Q&Aより)。 その他、モデル適合のためには半数体ゲノムの15倍のシーケンシングデータが必要であること、ONTやPacbioはエラー率が高く(数十塩基ごとにエラーが発生する)、GenomeScope 2.0では利用できないことに注意して下さい。
引用
GenomeScope 2.0 and Smudgeplot for reference-free profiling of polyploid genomes
T. Rhyker Ranallo-Benavidez, Kamil S. Jaron & Michael C. Schatz
Nature Communications volume 11, Article number: 1432 (2020)
関連