2019 12/9 タイトル修正
Venntは共通/非共通のDGEsを図示するのに使われるvenn diagramを描画してくれるhtmlベースのツール。
公式ページ
http://drpowell.github.io/vennt/
インストール
https://github.com/drpowell/vennt
brew install npm #npmがない人だけ
npm install -g browserify
npm install -g clean-css
npm install hbsfy@1.3
npm install handlebars-runtime
npm install coffeeify
./build.sh
ラン
ランにはcuffdiffの解析結果を入力するだけでよい。
python vennt.py --cuffdiff gene_exp.diff > my-vennt.html
公式サイトからリンクされているデモを確認してみる。

左上のboxをクリックすることでベン図に落とし込むデータを選択できる。
WT vs MT1とWT vs MT2だけ選択。

全データを選択し、FDRを0.01から0.05に変更、log FCは1に変更(2倍以上変動)。
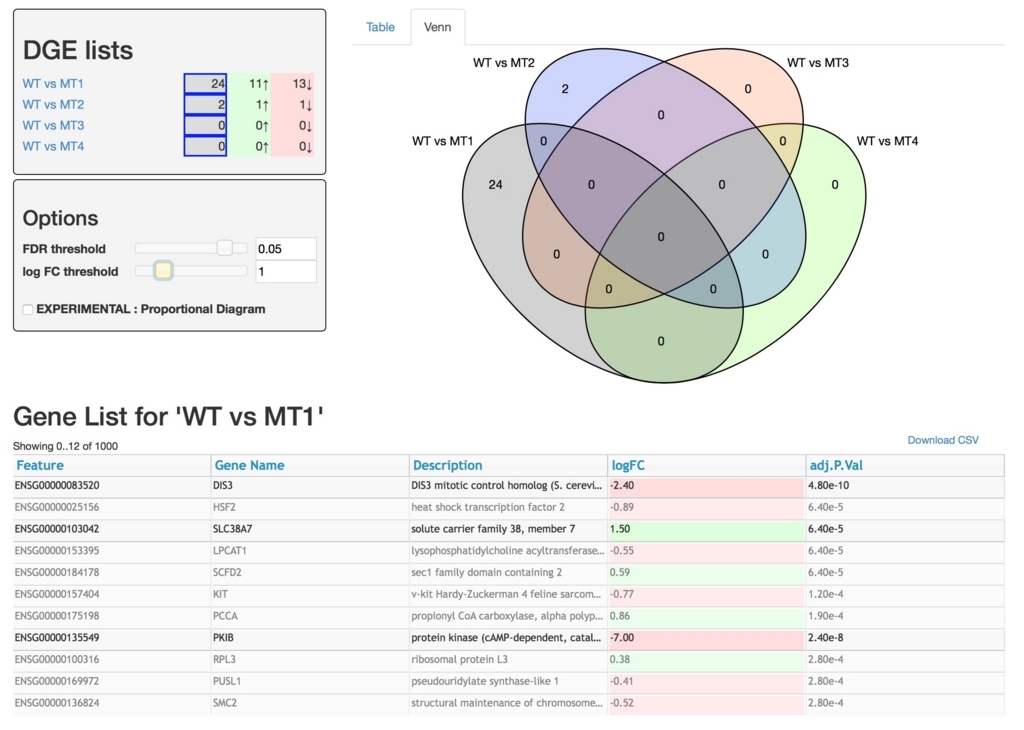
3データ以上で共通するDGEはゼロになった。
ベン図の数値をクリックすれば、該当遺伝子のテーブルに切り替わる。青の2をクリック。

下の表に2遺伝子が表示された。
Download CSVからデータをダウンロードできる。この条件でクリックすると2遺伝子だけの.csvファイルとなる。
引用