大規模な生体分子構造は、結晶学や電子顕微鏡などの確立された技術を用いて、日々実験的に決定されている。さらに、新たな統合的手法やハイブリッド手法(I/HM)により、時には数億個の水素原子以外の原子を含む巨大な高分子機械やアセンブリの構造モデルが作成されている。これらのデータを提供する可視化・解析ツールに要求される性能は急速に高まっている。オンライン、ウェブネイティブの3次元(3D)可視化ツールの開発は、LiteMolスイートとNGL Viewersの導入により大きく進展した。その後、PDBeとRCSB PDBが共同で、LiteMol(PDBeが開発)とNGL(RCSB PDBが開発)の長所を組み合わせて構築するMol*の開発に着手した。WebネイティブのMol* Viewerは、構造品質、機能性、生物学的背景の注釈を表示する機能とともに、高分子の座標および実験データの3D可視化とストリーミングを可能にする。高性能グラフィックスとデータ管理により、ユーザーは最大数百の(重ね合わせた)タンパク質構造を同時に可視化したり、分子動力学シミュレーションの軌跡をストリーミングしたり、細胞レベルのモデルをレンダリングしたり、巨大なI/HM構造を表示したりすることができる。PDBeとRCSB PDBで使用されている主要な3D構造ビューアである。サードパーティのサービスにも簡単に統合できる。Mol* Viewerはオープンソースで、https://molstar.org/ から自由に入手できる。
Documentation
https://molstar.org/viewer-docs/
https://www.youtube.com/watch?v=EdgmALgbJpM
Exploring PDB Structures in 3D with MolStar (Mol*): Introductory Guide
https://molstar.org/にアクセスする。
Open Mol* Viewerを選択するか、表示させたい高分子アイコンをクリックする。
ビューアが起動した。
以下のオンラインリソースから構造データを読み込むことができる。

PDBを選択した。PDBのIDを指定してApplyをクリックする。
読み込まれた。画面中央が3Dビューで、ここに構造が表示される。マウスの左クリックで3Dビュー画面を掴んで引っ張ると構造が回転し、右クリックで引っ張るとスクロールする。マウスホイールで拡大縮小もできる。
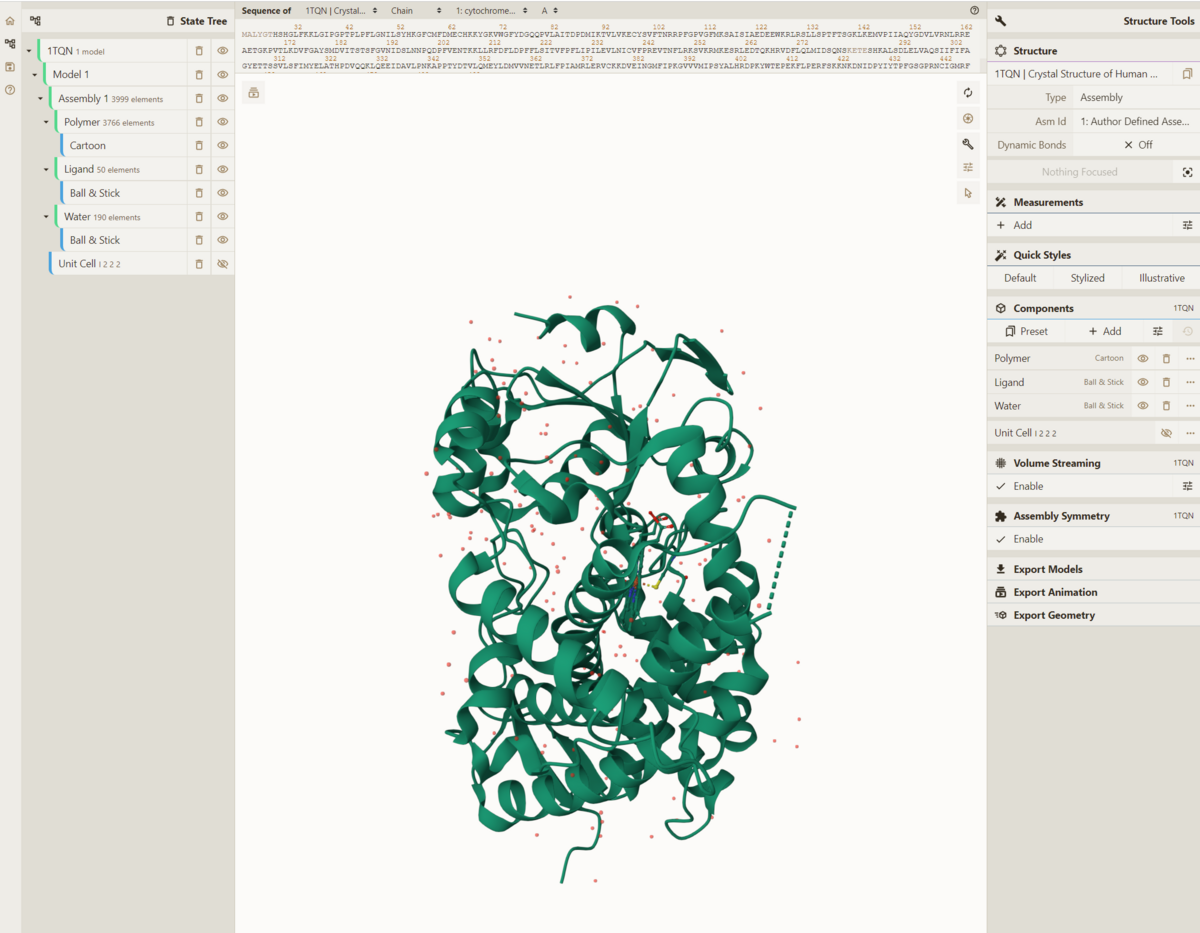
高分子複合体でも軽快に動作する。またスマートフォンやタブレットでも軽快に動作する。
移動はキーボードでもできる。Aで左へスクロール、Dで右にスクロール、Sで縮小、Wで拡大、Qで左回転、Rで右回転など。
別のコンポーネントやリガンドを区別して表示できる。3Dビューの上にある配列パネルでは、別のコンポーネントに切り替えできる。

カーソルを合わせるとそのアミノ酸残基がハイライトされ、上の配列パネルでもハイライトされる。

構造モデル上のカーソルを合わせた残基をクリックするとその周辺のアミノ酸側鎖が浮き出て、どの残基の側鎖と近いのか確認することができる。また同時に遠い領域が部分的に非表示になる(高分子で重なって見にくくなるのを防ぐ目的と考えられる)

解除するには無関係の空間をダブルクリックする。拡大表示も解除される。
特定のアミノ酸残基に素早く拡大する機能は、配列パネルの配列をクリックしても働く。

現在強調されているアミノ酸残基は右のパネルからも確認できる。THR 150と出ている。
右のパネルのQuick styleからはスタイルを一括変更できる。
sylized

illustrative

Componentsパネルでは、Componentやリガンド単位で表示・非表示を調整できる。これによって、リガンドだけを非表示にしたり、複数コンポーネントの一部だけを非表示にできる。周囲のWaterを非表示にした。

Componentsパネルのポリマーは、現在Cartoonになっている。これはコンポーネントごとに異なる色を適応する。

初期化して左上のホームボタンからPDBの別の構造”4V8K”(Crystal structure of the LH1-RC complex from Thermochromatium tepidum in P21 form)(DOI: https://doi.org/10.1038/nature13197)を読み込んでみた。

複雑な構造であっても軽快に動作する。
配列パネルの上から確認すると、この構造が多くのコンポーネントとリガンド、金属イオンからなることが分かる。

コンポーネントパネルでは、コンポーネント、リガンド、金属、配位している分子などの表示を細かくカスタマイズできる。リガンドの表示にGaussianを適用した。

適用したイメージはレイヤーのようになっているので、パネルの下から除外できる。リガンドパネルの下にGaussian Surface~とあるが、その右にあるごみ箱のマークをクリックするとGaussian Surfaceは削除される。その右の眼のマークでは、一時的に非表示できる。Ball &Stickは読み込んだリガンドモデルのデフォルトの表示なので、これまで削除してしまうと何も見えなくなる(再び追加すれば見える)。

右上のボタンアイコンからは、構造を回転表示させたり、シャドウ、アウトラインの表示・非表示の切り替えなどを行える。

回転させてみると分かるが、高いフレームレートで非常にスムーズに動作していることが分かる。回転機能は途中で拡大縮小しても止まらず、右上からOFFにするまで回転し続ける。SPINでなくROCKを選ぶと、実行時の角度で左右に5度程度の首振り回転をし続ける。
左下にはXYZ軸が表示され、どの方向に回転しているか分かる。

左下のXYZ軸は、タンパク質が現在どの向きを向いているのか確認するために便利。被写界深度(現在のズームの程度)には対応していない。
StereoをONにすると左右に同じ画面が2つ表示される。

右上のボタンパネルの上から3番目の工具のアイコンをクリックすると、メニューパネルが隠れてブラウザ上でフルスクリーンになる。

もう一度工具のアイコンをクリックすると元に戻る。
右上のボタンパネルの一番下の矢印アイコンをクリックすると選択モードになる。選択モードでは上にボタンメニューが出現する。

上のパネルからは選択レベルを調整できる。デフォルトでは残基になっており、クリックするたびに加算式で選択された残基を増やしていける。例えばchainに変えると、鎖単位で選択できるようになる。
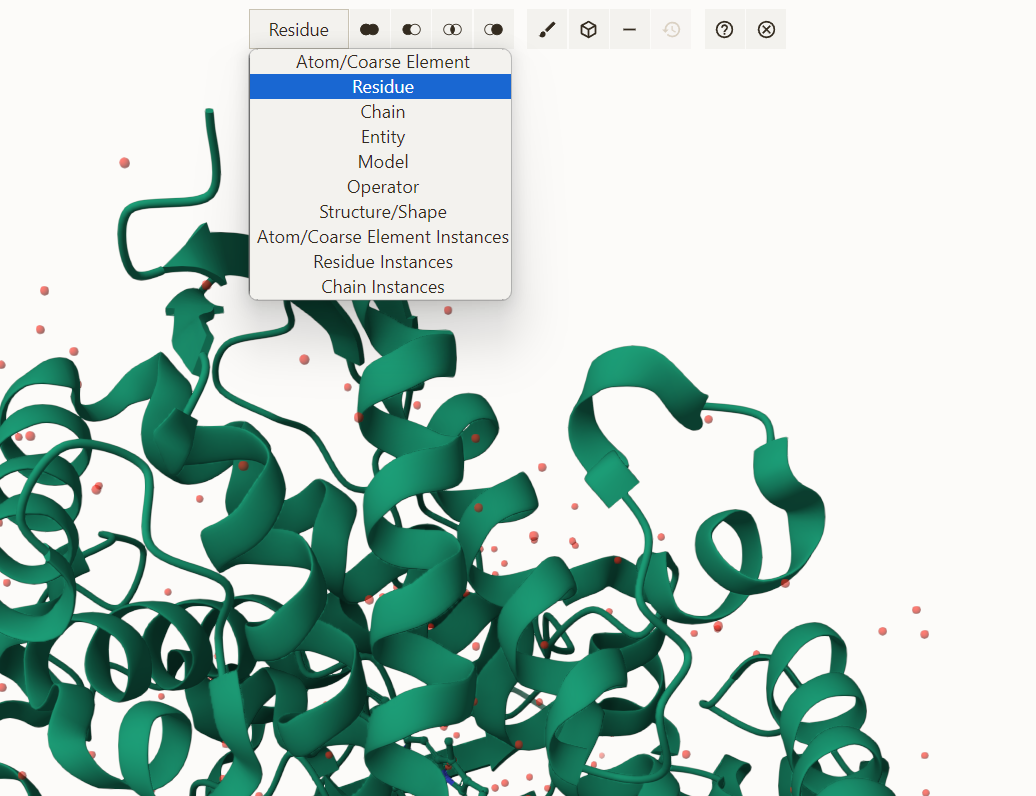
Residueの隣のアイコンからは条件によって自動選択ができる。Structure Propertyのhelixを選んだ。ヘリックス領域が全て選択された。

その右のRemoveアイコンからは条件に一致した領域だけを選択解除できる。細かい条件に対応しており、5Å以下のリガンドなどまである。

4つのボタンは下のようになっている(マニュアルより転載)。
その右の筆マークからは選択した領域の色を変更できる。

その右の立方体ボタンは選択した領域だけコンポーネントとして抽出できる。その右のマイナスボタンからは選択した領域だけ除去できる。その右には操作のUndoボタンも用意されている。

右下のパネルから様々なフォーマットで出力が可能。

ローカルのデータを読み込むには画面上にファイルをドラッグアンドドロップする。

Mol*サイトにあるほかの機能
- VSCoding-Sequence Visual Studio Codeの拡張機能で、エディタ上でタンパク質の構造や分子データを可視化することができる。
- PePr2Vis 周辺膜タンパク質の突起の計算と可視化のためのウェブサイト。
- MolAR 化学構造を拡張現実感で実現し、実世界の環境で見ることができる。
原子電荷電卓IIとαCharges
Atomic Charge Calculator IIとαChargesは、カスタムMol*拡張機能を使用して可視化された部分原子電荷の高速計算を行うウェブアプリケーション。
論文より
- Mol* Viewerは他のウェブ可視化ツールに比べ格段に大きな分子系を可視化することができる。組み込みのBinaryCIF、解凍サポート、モデルおよびボリューム/実験データストリーミングの高度な技術により、大きな構造でも限られた帯域幅でインタラクティブにレンダリングできる。そのため、リボソーム、ウイルスキャプシド、重ね合わせた高分子の集合体(同じタンパク質ファミリーの個々のメンバーの比較など)、あるいはMDシミュレーションシステムなど、多くの種類の大規模システムをレンダリングできる。
引用
Mol* Viewer: modern web app for 3D visualization and analysis of large biomolecular structures
David Sehnal, Sebastian Bittrich, Mandar Deshpande, Radka Svobodová, Karel Berka, Václav Bazgier, Sameer Velankar, Stephen K Burley, Jaroslav Koča, Alexander S Rose
Nucleic Acids Res. 2021 Jul 2;49(W1):W431-W437
追記
Mol* ViewerはUniProtなどでも使用されている。
操作性は同じだが、スムーズさなどMol* Viewerで見たときよりも簡素化されている。

