タンパク質の構造は、共通の祖先のシグナルを持ち、そのため進化の歴史を再構築するのに役立つ。構造情報推論を迅速に行うために、Structomeというウェブサーバーが開発され、ユーザーはクエリータンパク質に類似した構造を迅速に特定し、構造に基づく系統学に有用なデータセットを組み立てることができるようになった。Structomeは、RCSB PDB中の94%を90%の配列同一性でクラスタリングし、各クラスタをセントロイド構造で表現することにより作成された。セントロイド間の構造類似度を計算し、PDB、SCOP、CATHからのアノテーションを統合した。その結果、Structomeが返すタンパク質構造は、ヒストンフォールドの配列と構造の多様性の両方にまたがっていることが示された。さらに、Structomeが提供する計算済みのNexus-formated距離行列により、配列の類似性だけに基づく検索では識別できないタンパク質間の進化的関係を分析することができる。この結果は、Structomeを使用することで、単一の構造から始めて、構造的なneighboursのデータセットを迅速に生成することができ、タンパク質の深い進化の歴史を研究することができることを実証している。Structomeは、https://structome.bii.a-star.edu.sgで入手できる。
Bitbucket
https://structome.bii.a-star.edu.sg/にアクセスする。
キーワードかchain名で検索できる。大文字・小文字は区別されない。
表が出力される。windowsのchromeだと表示がおかしく見えるが、2行の結果が返されている。それぞれの行に重複しないセントロイドが表示されている。列には、PDB/Chainラベル(セントロイド)、サイズ、RCSB PDB description、SCOP/CATH family description、セントロイド構造が得られた生物種の列が表示されている、
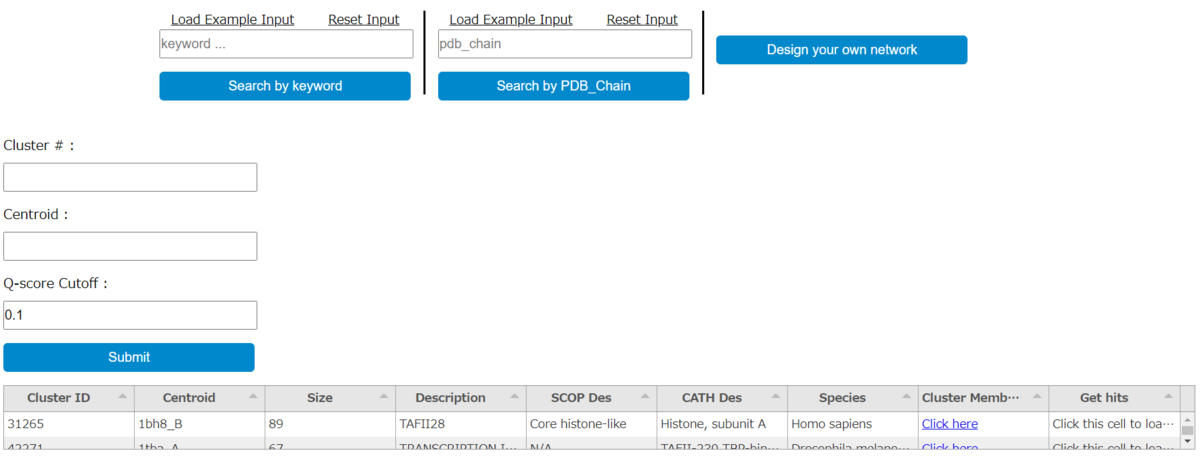
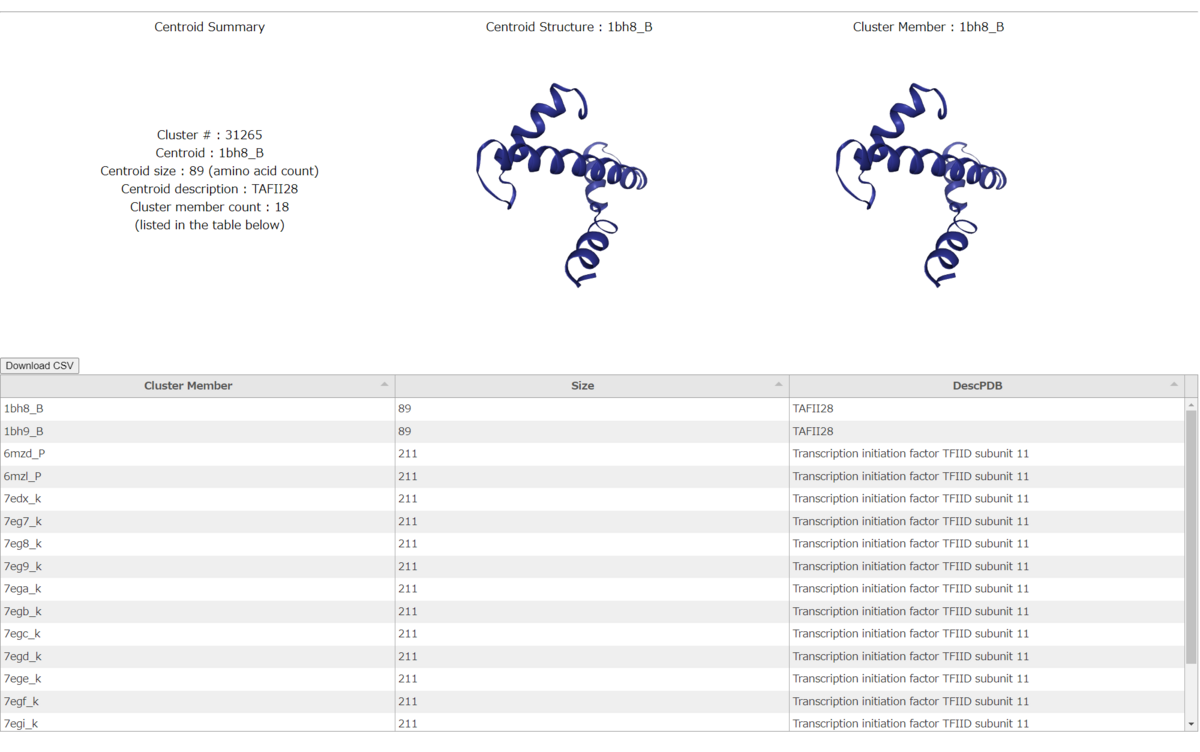
表の右から2番目のセルをクリックするとクラスターの要約ページが表示される。
また、ヒット結果の表の右端の列(Get hits)をクリックすると、表の上のデータ入力フィールドにクラスター番号とセントロイドPDBが読み込まれる。
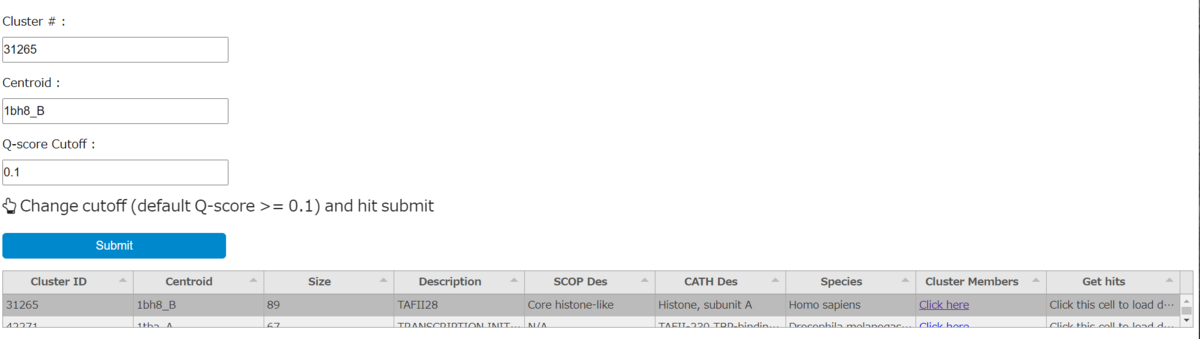
表の右端の列をクリック後、データ入力フィールドに文字と数値が表示されている。
Q-score Cutoffフィールドは、あるセントロイドをStructomeの他のセントロイドと比較したい時に、このカットオフ値よりも良いスコアを出したペアワイズセントロイド構造比較のみを次の結果ページに表示するために使用する(論文より)。
Q-score Cutoffを選択後、上の画像中央にあるSubmitをクリックすると、結果ページが表示される。
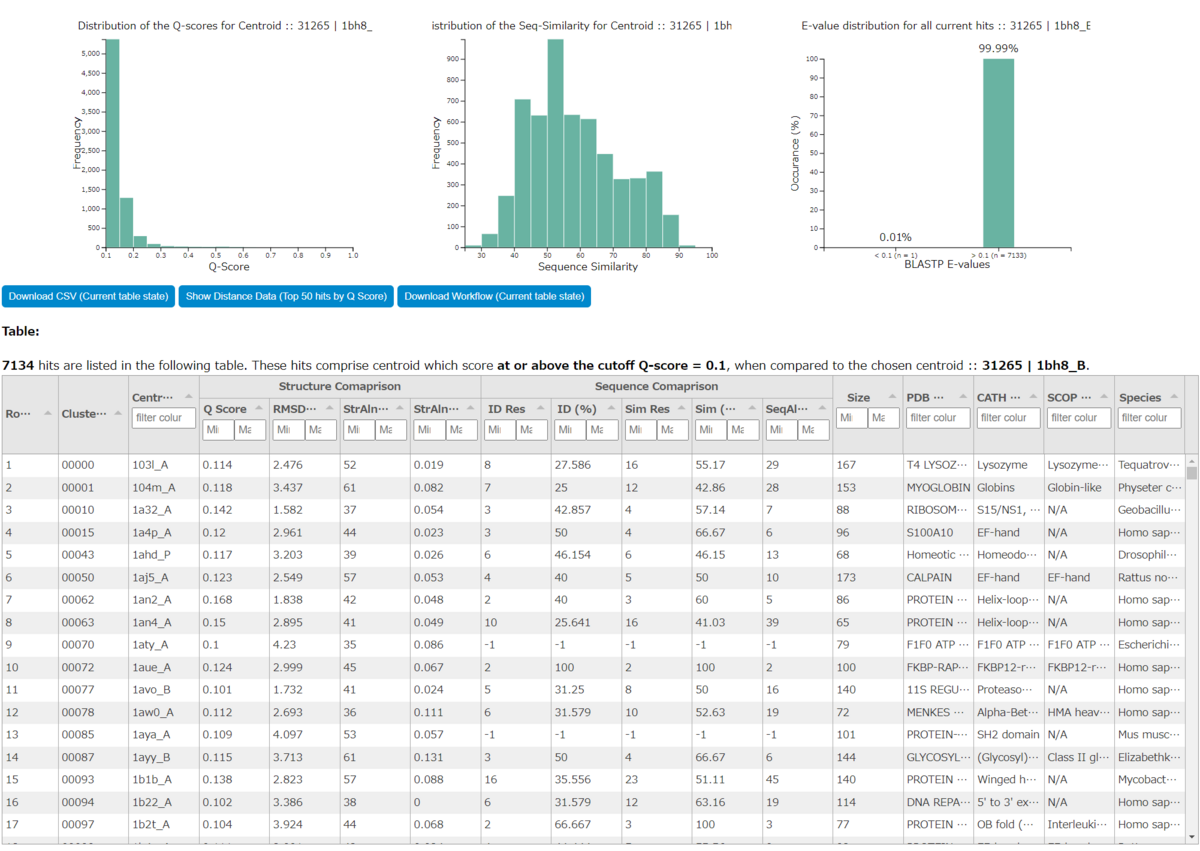
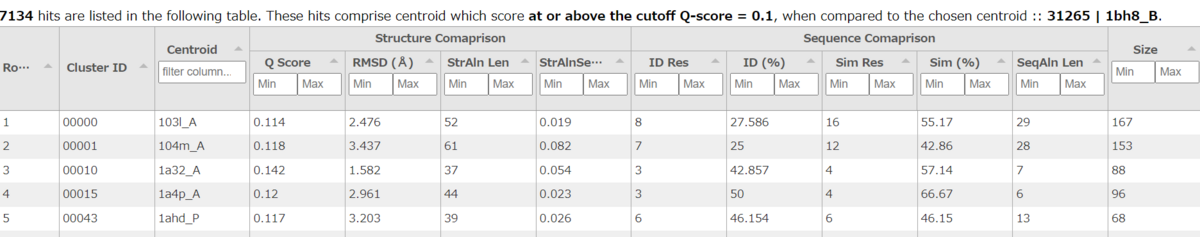
左の図は構造比較に基づくQスコアの分布、中央のグラフはBLASTに基づく配列類似度の分布、右の棒グラフは、BLASTPに基づくE値が任意のカットオフ値である0.1より下と上のヒット数。下の表は、前ステップで選んだセントロイドとStructomeの他のセントロイドとの間で、選んだQ-scoreカットオフ以上のスコアを持つすべてのセントロイド構造のマッチングを示している。
ここではヒトの1bh8_BをクエリにQ-score Cutoff 0.1でサブミットし、7134個のヒットが得られた。
引用
Structome: Exploring the structural neighbourhood of proteins
Ashar J Malik, Chandra S Verma, Anthony M Poole, Jane R Allison
bioRxiv, Posted February 21, 2023