バイオインフォマティクスと計算生物学の分野において、タンパク質の構造モデリングと解析は極めて重要な側面である。しかし、既存のツールのほとんどは高度な専門知識を必要とし、ユーザーフレンドリーなインターフェースを備えていない。この問題を解決するために、本著者らはPROFASAと呼ばれるタンパク質ワークステーションを開発した。PROFASAは革新的なタンパク質ワークステーションであり、最先端のタンパク質構造可視化技術とタンパク質解析のための最先端のツールとアルゴリズムを組み合わせたものである。本著者らの目標は、全てのタンパク質配列と構造解析のための包括的なプラットフォームをユーザーに提供することである。PROFASAは、複雑なタンパク質解析ワークフローをワンクリック操作で簡素化することを念頭に設計されており、同時にプロフェッショナルユーザーのニーズを満たす強力なカスタマイズオプションを提供している。
PROFASAは、タンパク質の構造評価、パラメトリック解析、タンパク質の可視化を可能にするワンストップ・ソリューションを提供する。ユーザーはI-TASSERまたはAlphaFold2を使用して、ワンクリックでタンパク質モデルを構築し、新しいタンパク質配列、モデルを生成し、タンパク質パラメータを計算することができる。さらに、PROFASAはリアルタイムコラボレーション、ノート共有、プロジェクト共有などの機能を提供し、研究者や教育専門家にとって理想的なツールとなっている。PROFASAの革新性は、ユーザーフレンドリーなインターフェースとワンストップ・ソリューションにある。タンパク質計算、解析、可視化ツールへの参入障壁を下げるだけでなく、タンパク質研究と教育に新たな可能性を開く。PROFASAがタンパク質設計と工学の研究を発展させ、新しい研究分野を切り開くことが期待される。
https://profasa.ucc.ie/#/にアクセスする。
最初にアカウントを作りログインする(30秒で出来る)。それから下にスクロールする。
4つの機能が利用できる。

1、Structure Blast
Structure Blastを見てみる。


保存されたドメインの検索にはRPS-BLASTが使用される。RPS-BLASTがConserved Domain Database (CDD)に従ってドメインが一致するタンパク質を検索し、これによってタンパク質プロファイルのセットが得られる。PROFASAでは、RpsBprocプログラムによって結果の解析とフィルタリングが行われ、フロントエンドの出力に表示される。
出力例
クエリの配列(黒線)とヒットした配列(赤とピンクの線)が表示される。

まずクエリの配列。左端にラマチャンドランプロット、中央にCanvasを使ったFunction2Form(F2F) Resultのレーダープロット、分子構造モデリングが示されている。

(全部の結果が出るまで最低数時間以上かかる)
(論文より)タンパク質は複数の「部分」/機能を備えており、ある部分を変化させることを目的としたタンパク質の改変は、通常、他のタンパク質の部分にも影響を及ぼす。最終的なタンパク質の全体的な性能に対する設計パラメータの複合的な影響を予測するシステムは存在しない。Function2Form Bridge (F2F-Bridge)(paper)は、解析対象のタンパク質に関連する様々な設計パラメータのスコアを、全体的な性能を記述する単一の解釈しやすい出力に組み合わせることにより、この問題に対処しようとする。

論文の表2を参照。
ここではクエリとヒットした2つのドメインがある。


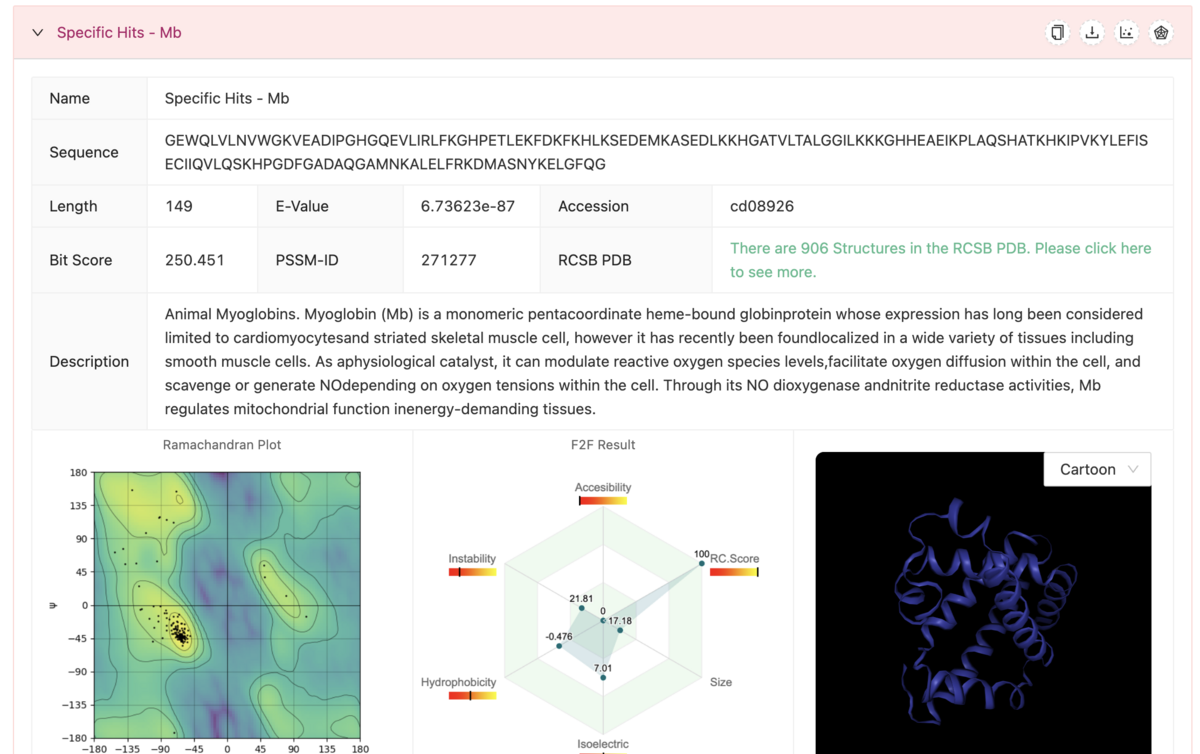
クエリと2つのヒットはRESULTから視覚化することができる。
よりマルチドメインのタンパク質だと、さらに多くのヒットが見つかる。

2、PROTEIN FOLD
PROTEIN FOLDは、AlphaFold2を使って複数のクエリー配列を次々と折りたたんでいく。その結果、折りたたまれたタンパク質モデルといくつかの基本情報が得られる。

3、Analysis
PDBファイルをアップロードして分子構造モデルの基本情報を閲覧したり、ダウンロードできる。閲覧時はモデルの表現方法や色を変更できる。

下の配列パネルを選択することで特定のアミノ酸残基をハイライトできる。

左側にラマチャンドランプロットとF2Fのレーダープロットを表示できる。

Descriptionのclick hereからPDBの同一の構造を確認できる。

右端のボタンからは図をPDF形式でダウンロードしたり、視覚化方法を変更できる。

4、Superimpose
Superimpose(重ね合わせ)機能によって、構造を回転させたり平行移動させたりして、他の構造と一致させたり整列させたりできる。そして、RMSDとGDT_TSを使用してスーパーインポーズを評価できる。

2つ以上のPDBファイルをアップロードする。
2つのPDBファイルをアップロードした時の出力例。右下にRMSDとGDT_TSのスコアが表示される。

(論文より)2つのタンパク質の比較可能なアミノ酸を決定し、タンパク質の位置を再計算してスーパーインポーズを完成させる。RMSD (Root-mean-square Deviation)は、タンパク質の優れたスーパーインポーズの基準と悪いスーパーインポーズの基準を決定するための統計量で、積み重ねられたタンパク質の原子間の平均距離は、RMSDによって測定される(Patel et al, 2019)。この値が小さいほど、重ね合わせた2つのタンパク質は外見的特徴や機能が近く、収束していることを意味する。PROFASAでは、まず配列アラインメントを行って2つの配列のどの残基が同一であるかを決定し、これらの残基に対応するアルファ炭素原子を探し出し、その中の原子の位置座標を更新するタンパク質オブジェクトを使用して位置座標を取得する。原子の位置座標からRMSDが求められる。さらに、これらのアルファ炭素原子の座標を通して、GDT_TS(Global Distance Test-Total Score)を計算することができる。2つの構造が詳細に異なる場合、GDT_TSはRMSDよりも重ね合わせ類似性を検出するのに優れている(Zemla, 2003)。
右端のボタンよりスーパーインポーズさせる領域を手動で選択できる。

RESULTSから全ての結果を確認したりアクセスしたりできる。

Progressの列ではジョブの進捗も確認できる。
引用
PROFASA—a web-based protein fragment and structure analysis workstation
Yanlin Mi, Stefan-Bogdan Marcu, Sabin Tabirca, Venkata V. B. Yallapragada
Front. Bioeng. Biotechnol., 21 July 2023
関連
