2020 7/13 誤字修正、説明の誤り修正
アウトブレイク解析では時間が非常に重要である。最近のシーケンス準備の進歩により、多くの病原体ではシーケンスがボトルネックとなっている。多くの病原体のゲノムサイズが小さいため、MinION シーケンシングにより数分で洞察力のあるシーケンス データが得られる。RAMPART は、そのような病原体の MinION シーケンシングと同時に実行される。RAMPARTは、各バーコードについてゲノム カバレッジとリファレンス マッチングに関するリアルタイムの概要を提供する。
インストール
マニュアル通りcondaの仮想環境を作ってテストした(ubuntu18.04LTS)。
依存
- python>=3.6
#condaを使う。python>=3.6
conda create -n artic-rampart -y nodejs=12
conda activate artic-rampart
conda install -y artic-network::rampart=1.1.0
#dependency
conda install -y anaconda::biopython
conda install -y -c conda-forge -c bioconda snakemake=5.10.0 #snakemake<5.11
conda install -y bioconda::minimap2=2.17
#optional (if you require RAMPART to perform demuxing)
python -m pip install git+https://github.com/artic-network/Porechop.git@v0.3.2pre
python -m pip install binlorry==1.3.0_alpha1
> rampart --help
$ rampart --help
usage: rampart [-h] [-v] [--verbose] [--ports PORTS PORTS]
[--protocol PROTOCOL] [--title TITLE]
[--basecalledPath BASECALLEDPATH]
[--annotatedPath ANNOTATEDPATH]
[--referencesPath REFERENCESPATH]
[--referencesLabel REFERENCESLABEL]
[--barcodeNames BARCODENAMES [BARCODENAMES ...]]
[--annotationOptions ANNOTATIONOPTIONS [ANNOTATIONOPTIONS ...]]
[--clearAnnotated] [--simulateRealTime SIMULATEREALTIME]
[--devClient] [--mockFailures]
RAMPART v1.1.0: Read Assignment, Mapping, and Phylogenetic Analysis in Real
Time
Optional arguments:
-h, --help Show this help message and exit.
-v, --version Show program's version number and exit.
--verbose verbose output
--ports PORTS PORTS The ports to talk to the client over. First: client
delivery, i.e. what localhost port to access rampart
via (default: 3000). Second: socket to transfer data
over (default: 3001)
--protocol PROTOCOL path to a directory containing protocol config files
Config commands:
Override options from config files
--title TITLE experiment title
--basecalledPath BASECALLEDPATH
path to basecalled FASTQ directory (default: don't
annotate FASTQs)
--annotatedPath ANNOTATEDPATH
path to destination directory for annotation CSVs -
will be created if it doesn't exist (default: '.
/annotations')
--referencesPath REFERENCESPATH
path to a FASTA file containing a panel of reference
sequences
--referencesLabel REFERENCESLABEL
the reference header field to use as a reference
label (if not just the reference name)
--barcodeNames BARCODENAMES [BARCODENAMES ...]
specify mapping of barcodes to sample names - e.g.
'BC01=Sample1' (can have more than one barcode
mapping to the same name)
--annotationOptions ANNOTATIONOPTIONS [ANNOTATIONOPTIONS ...]
pass through config options to the annotation script
(key=value pairs)
Runtime commands:
Options to specify how RAMPART behaves
--clearAnnotated remove any annotation files present when RAMPART
starts up (force re-annotation of all FASTQs)
--simulateRealTime SIMULATEREALTIME
simulate real-time annotation with given delay
between files (default none)
Development commands:
--devClient don't serve build (client)
--mockFailures stochastic failures (annotating / parsing)
テストラン
GitHubレポジトリにエボラウイルス(EBOV)のプロトコルの例がある。これを使う。
git clone https://github.com/artic-network/rampart.git
cd rampart/example_data/20181008_1405_EBOV
中身

MinIONのランではfastq/passにbasecallが上手くいったfastqが追加されていくが、RAMPARTはこのfastq ファイル群を認識する。
バーコードとサンプル名のマッピングなど実行のためのパラメータを定義したrun_configuration.jsonファイルが認識される。(ここではファイルは揃っているが、実際のランではリファレンスゲノムも用意する必要がある=>レポジトリの例を確認)。
rampart --protocol ../../example_protocols/EBOV --clearAnnotated
--clearAnnotated remove any annotation files present when RAMPART starts up (force re-annotation of all FASTQs)
localhost:3000 にアクセスする。

クリックすると展開される。ウィルスゲノム全体のリードのカバー率。
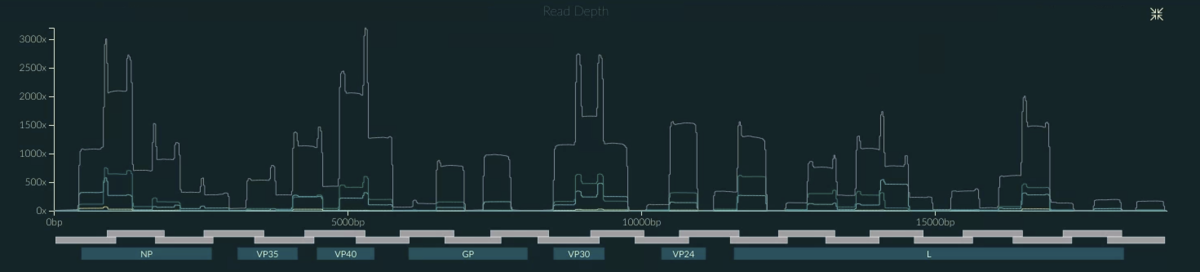
累積

バーコードにより複数データをmultiplexing runしている場合、 バーコードごとに出力を確認できる。
JSONファイルで遺伝子ポジションを指定している場合、遺伝子ごとのシーケンス率を表示できる。






実際のセットアップ例はmanualを読んでください。jsonファイルを編集して使用します。
https://github.com/artic-network/rampart/blob/master/docs/setting-up.md
引用
https://github.com/artic-network/rampart