2018 10/26 追記, 説明追加
2019 パラメータ追記, パラメータ修正, パラメータ追記, condaインストール追記, 0.14.1のhelpに更新, コメント追記, コマンド追記
2020 1/17 追記, help更新, multiqcと連携する例を追記
2023/01/11 other adapter sequences追記、12/14 構成を修正
2024/02/14追記
ダウンストリームデータ解析において高品質で信頼性の高いバリアントを得るためには、シーケンシングデータのクオリティ管理と前処理が不可欠となっている。データは、アダプター配列の汚染、塩基含有量のバイアス、過度な配列を持つことがある。より重要なことに、ライブラリーの調製およびシーケンシングステップは常にエラーを伴い、元の核酸配列の誤った表現を引き起こす可能性がある。近年、シーケンシング技術、特に次世代シーケンシング(NGS)技術は、臨床応用、特にnoninvasive prenatal testing(NIPT)(wiki)(Bianchi et al、2015)および癌診断において広く使用されている。例えば、循環系における癌関連のバイオマーカーを探す液体生検技術(Esposito、Criscitiello、Trapani、&Curigliano、2017)は、癌の診断と個人のオーダーメイド治療の決定に役立つ。液体生検の主要技術として、血漿、尿および他の循環液から腫瘍由来のDNAを検出する無細胞腫瘍DNA(catena)シーケンスが行われている。 ctDNAシークエンシングデータは通常非常にノイズが多く、検出された変異は通常、非常に低い突然変異対立遺伝子頻度(MAF)を有する。このような低MAF変異を検出して偽陽性および偽陰性を排除するためには、クオリティ管理およびデータ前処理が特に重要である。
既存のツールが既に存在するため、FASTQデータのクオリティ管理と前処理は解決された問題と考えられている。たとえば、FASTQC(Andrews)は、Javaベースのクオリティ管理ツールで、ベースごとおよびリードごとのクオリティプロファイリング機能を提供している。 Cutadapt(Martin、2011)は広く使われているアダプタートリマーで、いくつかのリードフィルター機能も備えている。 Trimmomatic(Bolger、Lohse、&Usadel、2014)もアダプターをトリミングするために広く使用されており、さらに特定のウインドウサイズでずらしてスキャンするアルゴリズムを使用してクオリティプルーニングを実行することもできる。 SOAPnuke(Y. Chen et al、2018)は、HadoopシステムでMapReduceを実装してアダプターのトリミングとフィルタリングを行う最近publistされたツールである。
しかし、これまでの実践では、FASTQデータクオリティ管理と前処理のための複数の異なるツールが使用されていた。たとえば、クオリティ管理にはFASTQC、アダプタートリミングにはCutadapt、リードのプルーニングとフィルター処理にはTrimmomaticを使用するのが典型的な組み合わせである。データを複数回読み込む必要があるため、前処理が遅くなり、I / Oが非効率的になっている。これらを組み合わせて使用する理由は、これらの問題をすべて解決できる単一のツールが存在しないためである。著者らは、クオリティ管理、アダプタトリミング、データフィルタリング、およびその他の便利な機能を1つのツールで統合するAfterQC(S. Chen et al。、2017)を開発した。 AfterQCは、すべての必要な操作を実行し、FASTQファイルの1回のスキャンでHTMLベースのレポートを出力できる便利なツールである(紹介)。また、ペアエンドリードのオーバーラップを探すことによって塩基を訂正する新しいアルゴリズムを提供した。しかし、AfterQCはPythonで開発されているため、大規模なFASTQファイルの処理には比較的多くの時間がかかる。
本論文では、FASTQデータのクオリティ管理、リードフィルタリング、ベース補正を行うための超高速なツールfastpを紹介する。 本ツールはFASTQC + Cutadapt + Trimmomatic + AfterQCのほとんどの機能をカバーしており、いずれよりも2〜5倍高速である。これらのツールで利用可能な機能に加えて、fastpは独自のunique molecular identifier(UMI)preprocessing、リードごとのpolyGテールトリミング、出力分割などの追加機能を提供する。 fastpは、フィルタリング前とフィルタリング後の両方のデータに対してQCレポートを単一のHTMLページで提供する。これにより、前処理ステップで変更されたクオリティ統計を直接比較することができる。 fastpは、シングルエンドおよびペアエンドイルミナデータのアダプタシーケンスを自動的に検出できる。 JavaやPythonで開発された上記のツールとは対照的に、fastpはC / C ++で開発されており、強固なマルチスレッド実装により、はるかに高速になっている。さらに、シーケンスエラーを修正または除去する機能に基づいて、fastpは従来のツールよりさらにクリーンなデータを得ることができる。
特徴
- filter out bad reads (too low quality, too short, or too many N...)
- cut low quality bases for per read in its 5' and 3' by evaluating the mean quality from a sliding window (like Trimmomatic but faster).
- trim all reads in front and tail
- cut adapters. Adapter sequences can be automatically detected,which means you don't have to input the adapter sequences to trim them.
- correct mismatched base pairs in overlapped regions of paired end reads, if one base is with high quality while the other is with ultra low quality
- trim polyG in 3' ends, which is commonly seen in NovaSeq/NextSeq data. Trim polyX in 3' ends to remove unwanted polyX tailing (i.e. polyA tailing for mRNA-Seq data)
- preprocess unique molecular identifer (UMI) enabled data, shift UMI to sequence name.
- report JSON format result for further interpreting.
- visualize quality control and filtering results on a single HTML page (like FASTQC but faster and more informative).
- split the output to multiple files (0001.R1.gz, 0002.R1.gz...) to support parallel processing. Two modes can be used, limiting the total split file number, or limitting the lines of each split file.
- support long reads (data from PacBio / Nanopore devices).
2018 05現在preprintが投稿されています。publishに向けて今後も機能追加の可能性があるとの記載がありますが、preprecessingはすべての解析に関わる影響力が大きな基幹となるプロセスです。たとえわずかでもフィルタリング精度が改善され処理時間が短くなれば、解析に携わる人全てに積み重なって大きな恩恵をもたらします。早めにまとめておきます。
追記
2018年にBioinformaticsにアクセプトされています。
好評のようですね。
Very impressed with fastp. One stop shop for error correction (paired-end), adapter trimming, and quality filtering. Also, it flies. It has replaced musket and trimmomatic in my assembly pipeline. git repo: https://t.co/mZd8D5ls1F paper: https://t.co/InDTB4GEXx
— ᴍᴀʀᴄ ᴄʜᴇᴠʀᴇᴛᴛᴇ (@wildtypeMC) October 25, 2018
インストール
mac os10.12と10.13でテストした。
依存
本体 Github
git clone https://github.com/OpenGene/fastp.git
cd fastp
make
sudo make install
#bioconda
mamba install -c bioconda -y fastp
> fastp #v 0.20.0
$ fastp
fastp: an ultra-fast all-in-one FASTQ preprocessor
version 0.20.0
usage: fastp [options] ...
options:
-i, --in1 read1 input file name (string [=])
-o, --out1 read1 output file name (string [=])
-I, --in2 read2 input file name (string [=])
-O, --out2 read2 output file name (string [=])
--unpaired1 for PE input, if read1 passed QC but read2 not, it will be written to unpaired1. Default is to discard it. (string [=])
--unpaired2 for PE input, if read2 passed QC but read1 not, it will be written to unpaired2. If --unpaired2 is same as --umpaired1 (default mode), both unpaired reads will be written to this same file. (string [=])
--failed_out specify the file to store reads that cannot pass the filters. (string [=])
-m, --merge for paired-end input, merge each pair of reads into a single read if they are overlapped. The merged reads will be written to the file given by --merged_out, the unmerged reads will be written to the files specified by --out1 and --out2. The merging mode is disabled by default.
--merged_out in the merging mode, specify the file name to store merged output, or specify --stdout to stream the merged output (string [=])
--include_unmerged in the merging mode, write the unmerged or unpaired reads to the file specified by --merge. Disabled by default.
-6, --phred64 indicate the input is using phred64 scoring (it'll be converted to phred33, so the output will still be phred33)
-z, --compression compression level for gzip output (1 ~ 9). 1 is fastest, 9 is smallest, default is 4. (int [=4])
--stdin input from STDIN. If the STDIN is interleaved paired-end FASTQ, please also add --interleaved_in.
--stdout stream passing-filters reads to STDOUT. This option will result in interleaved FASTQ output for paired-end output. Disabled by default.
--interleaved_in indicate that <in1> is an interleaved FASTQ which contains both read1 and read2. Disabled by default.
--reads_to_process specify how many reads/pairs to be processed. Default 0 means process all reads. (int [=0])
--dont_overwrite don't overwrite existing files. Overwritting is allowed by default.
-V, --verbose output verbose log information (i.e. when every 1M reads are processed).
-A, --disable_adapter_trimming adapter trimming is enabled by default. If this option is specified, adapter trimming is disabled
-a, --adapter_sequence the adapter for read1. For SE data, if not specified, the adapter will be auto-detected. For PE data, this is used if R1/R2 are found not overlapped. (string [=auto])
--adapter_sequence_r2 the adapter for read2 (PE data only). This is used if R1/R2 are found not overlapped. If not specified, it will be the same as <adapter_sequence> (string [=auto])
--adapter_fasta specify a FASTA file to trim both read1 and read2 (if PE) by all the sequences in this FASTA file (string [=])
--detect_adapter_for_pe by default, the auto-detection for adapter is for SE data input only, turn on this option to enable it for PE data.
-f, --trim_front1 trimming how many bases in front for read1, default is 0 (int [=0])
-t, --trim_tail1 trimming how many bases in tail for read1, default is 0 (int [=0])
-b, --max_len1 if read1 is longer than max_len1, then trim read1 at its tail to make it as long as max_len1. Default 0 means no limitation (int [=0])
-F, --trim_front2 trimming how many bases in front for read2. If it's not specified, it will follow read1's settings (int [=0])
-T, --trim_tail2 trimming how many bases in tail for read2. If it's not specified, it will follow read1's settings (int [=0])
-B, --max_len2 if read2 is longer than max_len2, then trim read2 at its tail to make it as long as max_len2. Default 0 means no limitation. If it's not specified, it will follow read1's settings (int [=0])
-g, --trim_poly_g force polyG tail trimming, by default trimming is automatically enabled for Illumina NextSeq/NovaSeq data
--poly_g_min_len the minimum length to detect polyG in the read tail. 10 by default. (int [=10])
-G, --disable_trim_poly_g disable polyG tail trimming, by default trimming is automatically enabled for Illumina NextSeq/NovaSeq data
-x, --trim_poly_x enable polyX trimming in 3' ends.
--poly_x_min_len the minimum length to detect polyX in the read tail. 10 by default. (int [=10])
-5, --cut_front move a sliding window from front (5') to tail, drop the bases in the window if its mean quality < threshold, stop otherwise.
-3, --cut_tail move a sliding window from tail (3') to front, drop the bases in the window if its mean quality < threshold, stop otherwise.
-r, --cut_right move a sliding window from front to tail, if meet one window with mean quality < threshold, drop the bases in the window and the right part, and then stop.
-W, --cut_window_size the window size option shared by cut_front, cut_tail or cut_sliding. Range: 1~1000, default: 4 (int [=4])
-M, --cut_mean_quality the mean quality requirement option shared by cut_front, cut_tail or cut_sliding. Range: 1~36 default: 20 (Q20) (int [=20])
--cut_front_window_size the window size option of cut_front, default to cut_window_size if not specified (int [=4])
--cut_front_mean_quality the mean quality requirement option for cut_front, default to cut_mean_quality if not specified (int [=20])
--cut_tail_window_size the window size option of cut_tail, default to cut_window_size if not specified (int [=4])
--cut_tail_mean_quality the mean quality requirement option for cut_tail, default to cut_mean_quality if not specified (int [=20])
--cut_right_window_size the window size option of cut_right, default to cut_window_size if not specified (int [=4])
--cut_right_mean_quality the mean quality requirement option for cut_right, default to cut_mean_quality if not specified (int [=20])
-Q, --disable_quality_filtering quality filtering is enabled by default. If this option is specified, quality filtering is disabled
-q, --qualified_quality_phred the quality value that a base is qualified. Default 15 means phred quality >=Q15 is qualified. (int [=15])
-u, --unqualified_percent_limit how many percents of bases are allowed to be unqualified (0~100). Default 40 means 40% (int [=40])
-n, --n_base_limit if one read's number of N base is >n_base_limit, then this read/pair is discarded. Default is 5 (int [=5])
-e, --average_qual if one read's average quality score <avg_qual, then this read/pair is discarded. Default 0 means no requirement (int [=0])
-L, --disable_length_filtering length filtering is enabled by default. If this option is specified, length filtering is disabled
-l, --length_required reads shorter than length_required will be discarded, default is 15. (int [=15])
--length_limit reads longer than length_limit will be discarded, default 0 means no limitation. (int [=0])
-y, --low_complexity_filter enable low complexity filter. The complexity is defined as the percentage of base that is different from its next base (base[i] != base[i+1]).
-Y, --complexity_threshold the threshold for low complexity filter (0~100). Default is 30, which means 30% complexity is required. (int [=30])
--filter_by_index1 specify a file contains a list of barcodes of index1 to be filtered out, one barcode per line (string [=])
--filter_by_index2 specify a file contains a list of barcodes of index2 to be filtered out, one barcode per line (string [=])
--filter_by_index_threshold the allowed difference of index barcode for index filtering, default 0 means completely identical. (int [=0])
-c, --correction enable base correction in overlapped regions (only for PE data), default is disabled
--overlap_len_require the minimum length to detect overlapped region of PE reads. This will affect overlap analysis based PE merge, adapter trimming and correction. 30 by default. (int [=30])
--overlap_diff_limit the maximum number of mismatched bases to detect overlapped region of PE reads. This will affect overlap analysis based PE merge, adapter trimming and correction. 5 by default. (int [=5])
--overlap_diff_percent_limit the maximum percentage of mismatched bases to detect overlapped region of PE reads. This will affect overlap analysis based PE merge, adapter trimming and correction. Default 20 means 20%. (int [=20])
-U, --umi enable unique molecular identifier (UMI) preprocessing
--umi_loc specify the location of UMI, can be (index1/index2/read1/read2/per_index/per_read, default is none (string [=])
--umi_len if the UMI is in read1/read2, its length should be provided (int [=0])
--umi_prefix if specified, an underline will be used to connect prefix and UMI (i.e. prefix=UMI, UMI=AATTCG, final=UMI_AATTCG). No prefix by default (string [=])
--umi_skip if the UMI is in read1/read2, fastp can skip several bases following UMI, default is 0 (int [=0])
-p, --overrepresentation_analysis enable overrepresented sequence analysis.
-P, --overrepresentation_sampling one in (--overrepresentation_sampling) reads will be computed for overrepresentation analysis (1~10000), smaller is slower, default is 20. (int [=20])
-j, --json the json format report file name (string [=fastp.json])
-h, --html the html format report file name (string [=fastp.html])
-R, --report_title should be quoted with ' or ", default is "fastp report" (string [=fastp report])
-w, --thread worker thread number, default is 2 (int [=2])
-s, --split split output by limiting total split file number with this option (2~999), a sequential number prefix will be added to output name ( 0001.out.fq, 0002.out.fq...), disabled by default (int [=0])
-S, --split_by_lines split output by limiting lines of each file with this option(>=1000), a sequential number prefix will be added to output name ( 0001.out.fq, 0002.out.fq...), disabled by default (long [=0])
-d, --split_prefix_digits the digits for the sequential number padding (1~10), default is 4, so the filename will be padded as 0001.xxx, 0 to disable padding (int [=4])
--cut_by_quality5 DEPRECATED, use --cut_front instead.
--cut_by_quality3 DEPRECATED, use --cut_tail instead.
--cut_by_quality_aggressive DEPRECATED, use --cut_right instead.
--discard_unmerged DEPRECATED, no effect now, see the introduction for merging.
-?, --help print this message
ラン
デフォルト設定では、Quality filtering、Length filtering、Low complexity filter、Adapter trimming、が有効になっている。
1、シングルエンド。 gz形式の入力にも対応している。
fastp -i single.fq -o cleaned.fq.gz -w 3 -q 15 -n 10
- -i read1 input file name (string)
- -o read1 output file name (string [=])
- -w worker thread number, default is 3 (int [=3])
- -q the quality value that a base is qualified. Default 15 means phred quality >=Q15 is qualified. (int [=15])
-
-u how many percents of bases are allowed to be unqualified (0~100). Default 40 means 40% (int [=40])
-
-n if one read's number of N base is >n_base_limit, then this read/pair is discarded. Default is 5 (int [=5])
2, ペアエンド。出力はgz圧縮する。レポートファイルをhtmlとjsonでそれぞれ出す。tail末端の1塩基をトリミングする。 20bp以下は捨てる。最大の16スレッド使う。
fastp -i pair1.fq -I pair2.fq -3\
-o out_pair1.fq.gz -O out_pair2.fq.gz\
-h report.html -j report.json -q 15 -n 10 -t 1 -T 1 -l 20 -w 16
#上の条件に加えて、先頭1塩基もトリミングする
fastp -i pair1.fq -I pair2.fq -3\
-o out_pair1.fq.gz -O out_pair2.fq.gz\
-h report.html -j report.json -q 15 -n 10 -t 1 -T 1 -l 20 -w 16 -f 1 -F 1
- -i read1 input file name (string)
- -I read2 input file name (string [=])
- -o read1 output file name (string [=])
- -O read2 output file name (string [=])
- -3 enable per read cutting by quality in tail (3'), default is disabled (WARNING: this will interfere deduplication for SE data)
- -w worker thread number, default is 3 (int [=3])
- -n if one read's number of N base is >n_base_limit, then this read/pair is discarded. Default is 5 (int [=5])
- -t trimming how many bases in tail for read1, default is 0 (int [=0])
- -T trimming how many bases in tail for read2, default is 0 (int [=0])
- -A adapter trimming is enabled by default. If this option is specified, adapter trimming is disabled
- -f trimming how many bases in front for read1, default is 0 (int [=0])
- -F trimming how many bases in front for read2. If it's not specified, it will follow read1's settings (int [=0])
- -l reads shorter than length_required will be discarded, default is 15. (int [=15])
クオリティトリミングを無効化するには"-Q"をつける。アダプターを自動検出してトリミングする機能は無効化するなら"-A"をつける。
3,レポートのみ作成。リードのトリミングやフィルタリングを実行しないならインプットのfastqのみ指定する。
fastp -i pair1.fq.gz -I pair2.fq.gz
ランが終わるとhtml形式のレポートが出力される。
レポートファイル

クオリティ分布。グラフは拡大縮小したり他のwebツールに渡すことができる。

塩基含量。
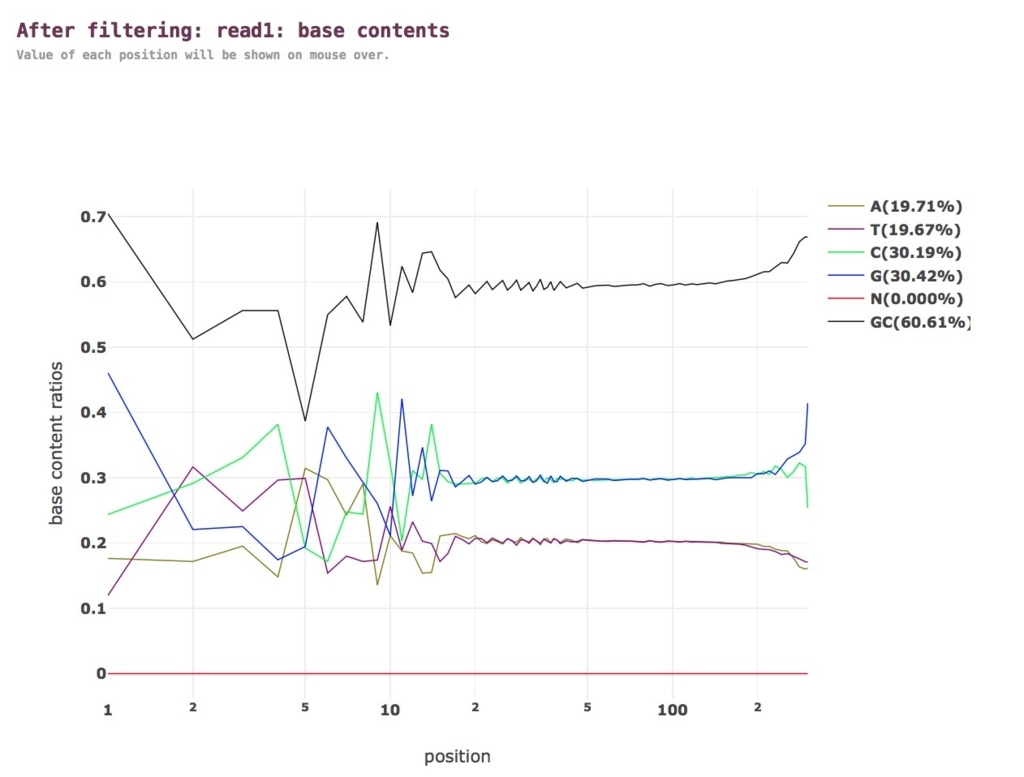
以下省略。
4,カスタムアダプタ−配列の指定。
fastp -i pair1.fq -I pair2.fq -3 -o out_pair1.fq.gz -O out_pair2.fq.gz --adapter_sequence TATATA --adapter_sequence_r2 AGAGAG
- -a, --adapter_sequence the adapter for read1. For SE data, if not specified, the adapter will be auto-detected. For PE data, this is used if R1/R2 are found not overlapped. (string [=auto])
- --adapter_sequence_r2 the adapter for read2 (PE data only). This is used if R1/R2 are found not overlapped. If not specified, it will be the same as <adapter_sequence> (string [=auto])
- --adapter_fasta specify a FASTA file to trim both read1 and read2 (if PE) by all the sequences in this FASTA file (string [=])
5,tips。出力ファイルの分割には-s(--split)フラグを使う。
fastp -i pair1.fq -I pair2.fq -3 -o out_pair1.fq.gz -O out_pair2.fq.gz --split 3
出力ファイルが均等に3分割される。

6,ロングリードのフィルタリング。ロングリードの品質管理にも使える。しかしトリミングをONにすると、ホモポリマーを読んだ領域などエラーが多い領域でトリミングされる恐れがある(より下流が高品質に読めていたとしても)。回避するために、スライディングウィンドウのサイズをデフォルトの"-W 4 "から増やす。ここでは-W 50とした(要検討)。
#raw ONT
fastp -i raw_long-reads.fq.gz -o QT_long-reads.fq.gz \
-W 50 -M 10
- -W the window size option shared by cut_front, cut_tail or cut_sliding. Range: 1~1000, default: 4 (int [=4])
- -M the mean quality requirement option shared by cut_front, cut_tail or cut_sliding. Range: 1~36 default: 20 (Q20) (int [=20])
(* ノイジーなロングリードのフィルタリングにはFiltlongが有用です(紹介)。)
7,強いクオリティフィルタリング。エラーを含むリードがあると、浸透率の低いバリアントをコールする際に低品質リードが多い領域からのコールがたくさん出てしまう。raw VCFを作ったあとでフィルタリングする事も出来るが、生のfastqの時点で強いフィルタリングをかけて生物学的な価値がない不要なコールを減らすと下流解析がシンプルになる。品質が低いリードの強いフィルタリングには、よく使うオプションに追加して”-e”オプションを追加するのが効果的。例えば"-e 35"を加えて平均クオリティ35以下のリードは除外する。
#先頭5塩基と末端1塩基の強制トリミング & sliding windowsによるトリミング、長さフィルタリング(20以下)、平均クオリティ35以下のリードのフィルタリング
fastp -i raw_R1.fq.gz -I raw_R2.fq.gz -o QT_R1.fq.gz -O QT_R2.fq.gz \
-q 30 -t 1 -T 1 -f 5 -F 5 -l 20 -w 16 -e 35
- -e if one read's average quality score <avg_qual, then this read/pair is discarded. Default 0 means no requirement (int [=0])
平均カバレッジが潤沢なら強いフィルタリングをかけても下の画像のように十分なリードが残る。

画像は、上のコマンドを実行前のIGVのアラインメントと(上半分)、実行後(下半分)のIGVのアラインメント。pileupのコーラーだけでなく、freebayesのようなハプロタイプベースのバリアントコーラーでも、上半分の部位は位相が揃っているためにraw VCFではコールすることが多い。fastpで贅沢なフィルタリングを行い(下半分の状態)、それからバリアントコールすればでこのような部位はraw コールから除外できる。ただしシークエンスしにくい領域のカバレッジがゼロ近くになるような弊害が発生する恐れがある。
8,MultiQCによる統合レポート
fastpはMultiQC(紹介)でサポートされています。fastpラン後にMultiQCを走らせるだけで統合レポート(html、PDFなど)を簡単に作成できます。
#fastpとmultiqcを連携させて使う例。illuminaはハイクオリティなのでQ30で切る。
#sample1
fastp -i sample1_1.fq.gz -I sample1_2.fq.gz -3 -o sample1_QT1.fq.gz -O sample1_QT2.fq.gz\
-h sample1_fastp.html -j sample1_fastp.json -q 30 -n 10 -t 1 -T 1 -l 20 -w 16
#sample2
fastp -i sample2_1.fq.gz -I sample2_2.fq.gz -3 -o sample2_QT1.fq.gz -O sample2_QT2.fq.gz\
-h sample2_fastp.html -j sample2_fastp.json -q 30 -n 10 -t 1 -T 1 -l 20 -w 16
#sample3
fastp -i sample3_1.fq.gz -I sample3_2.fq.gz -3 -o sample3_QT1.fq.gz -O sample3_QT2.fq.gz\
-h sample3_fastp.html -j sample3_fastp.json -q 30 -n 10 -t 1 -T 1 -l 20 -w 16
#multiqc "fastp"が認識され、jsonが分析、統合レポートが出力される。
multiqc .
その他
- 実験的にoptions.cppを変更して最大CPUを増やしてビルドしてみたが、ランタイムに顕著な差はなかった。
- 前処理がバクテリアのSNPコールに与える影響を調べたところ、リードのトリミングによるSNPコール精度の向上は、統計的には重要ではないと示した論文が出ている。fastpが最も性能の高いプリプロセッサと結論付けられている。https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8116680/
- カスタムのアダプター配列が高度に残存している場合、レポートのAdaptersのセクションのother adapter sequencesの行が見つかる。カスタムアダプターは、fastpを実行してもトリミングされない(-Aの有無にかかわらず)。lessで実際にリードを少し眺めてみて、例えばR2リードの末端に高頻度にその配列8-bpがあることがわかったら、-T 8をつけて強制トリミングを行う。もう一度fastpを実行し、レポートのother adapter sequencesの行が消えていることを確認する。
引用
fastp: an ultra-fast all-in-one FASTQ preprocessor
Shifu Chen, Yanqing Zhou, Yaru Chen, Jia Gu
Bioinformatics, Volume 34, Issue 17, 1 September 2018, Pages i884–i890,
fastp: an ultra-fast all-in-one FASTQ preprocessor
Shifu Chen1,2,*, Yanqing Zhou1, Yaru Chen1, Jia Gu
bioRxiv preprint first posted online Mar. 1, 2018;
doi: http://dx.doi.org/10.1101/274100.
https://www.biorxiv.org/content/biorxiv/early/2018/03/01/274100.full.pdf
RNA seq
ロングリードにも対応。fastpより高速。
ロングリード