2019 3/18 インストールの流れ修正
2019 3/26 誤字修正
2019 11/8 タイトル修正
ヒトゲノムは二倍体であり、すなわち、その常染色体の各々は2コピーである。これらの親のコピーは、異なる一塩基多型(SNPs)の影響を受ける。変異がどちらの染色体由来かアサインすることは進化遺伝学の助けになり、例えばpopulation研究(論文より The 1000 Genomes Project Consortium、2010; Francioli et al、2014)における選択的な圧力と亜集団の特定に関係し、おそらく病気の原因となるSNPsを相互に結びつける(Hartl and Clark、2007)。アサインするプロセスはphasing(以後フェージング)と呼ばれ、結果のSNPsグループはハプロタイプと呼ばれる。国際的に協調した取り組みにより、対応する下流分析に役立つ多様な集団のハプロタイプの参照パネルが作成された(International HapMap Consortium、2007、2010)。
バリアントのフェージングには2つの主要なアプローチがある。第1のアプローチは、SNPsのアレルのそれらの接合体のステータスつきリストとなる遺伝子型に依存する。ホモ接合の対立遺伝子は相同染色体両コピー上に現れ、明らかに両方のハプロタイプに適用されるが、ヘテロ接合対立遺伝子はコピーのうちの1つにのみ現れ、したがって2つのグループに分けられなければならない。 よってm個のヘテロ接合のSNPsアレルがあると、2のm乗通りのハプロタイプの可能性が生じる。対応する手法は、通常は統計手法になり、既存の参照パネルを統合する。根底にある仮定は、計算されるハプロタイプは、減数分裂中に組換えから生じる基準ハプロタイプブロックのモザイクであるということである。観察された遺伝子型が与えられると、統計的に最も可能性の高いモザイクが出力される。最も一般的なアプローチは、潜在変数モデリング(Howie et al、2009; Li et al、2010; Scheet and Stephens、2006)に基づいている。他のアプローチでは、マルコフ連鎖モンテカルロ法を用いる(Menelaou and Marchini、2013)。
他のアプローチは、シーケンシングデータを直接読み込む。このようなアプローチは、実質的に同一の染色体コピーからのリードを集め、 haplotype assembly approachesと呼ばれる。原則に従えば、目標は、2つのハプロタイプを計算して、最小限の量の配列エラーを訂正してすべてのリードを割り当てることである。このような処方の中で、最近の注目の大半は最小誤差訂正(MEC)問題が生じている。セクション2で正式に定義するMECの問題は、リードを競合のない2つのハプロタイプに配置するために、配列決定されたヌクレオチドに対して行われる補正の最小数を見つけることにある。 MECの主な利点は、phredスケールのエラー確率に対処するために、重み付きバージョン(wMEC)に容易に適合させることができることである。このようなphredベースのエラースキームは、特に将来の技術によって生成されたロングリードを処理するためには不可欠である。 wMEC問題の最適解は、修正されるべきエラーと比較した最尤シナリオに変換される。
ますます増大するリード長および減少するシーケンス決定コストのテラスケールシーケンスプロジェクト(例えば、Boomsma et al、2013; The 1000 Genomes Project Consortium、2010)は、明らかにリードデータから直接フェーズを合わせることが望ましい。しかし、(i)大部分のNGSリードは、いわゆるvariant desertsをブリッジするにはまだ短すぎるため、統計的アプローチが依然として選択される。しかし、連続的なリードベースのフェージングには、隣接するヘテロ接合のSNPsアレルが全てカバーされないといけない。 (ii)MECの問題はNP困難であり、他のすべての同様の問題の定式化もそうである。
MEC(Chen et al、2013; He et al。、2010)の最も進んだ既存のアルゴリズムソリューションは、皮肉なことにvariant desertsから正確に利益を得ていることが多い。しかし、リードベースのアプローチの主な動機は、可能な限り多くのバリアントをカバーするロングリードを処理することで、すべてのバリアントをブリッジすることである。したがって、現在のハプロタイプアセンブリの認識は、それが克服するのが難しい理論上の限界の根底にあることが多い。
この論文では、カバレッジ、つまりSNPs位置をカバーするフラグメントの数が唯一のパラメータである、wMECに対する固定パラメータの扱いやすい(FPT)アプローチであるWhatsHapを紹介する。よって著者らのアプローチの実行時間は、SNPs数の多項式(実際には線形)である。 wMECのための線形ランタイムソリューションは、将来のシークエンシング技術が数万塩基対(bp)のリードを生成し、それらが高い配列決定エラー率を被る可能性が高いことの両方に対処する。慎重に設計されたアルゴリズムを実装することで、標準的なワークステーション上で最大20時間のオーダーで最大カバレッジの全ゲノムデータセットを処理することができる。カバレッジの高いデータセットでは、合理的な選択肢を選択するテクニックを提供する。 WhatsHapはさまざまな面でさまざまな長さのシミュレートされたリードに基づいて比較され、評価された。これらのすべてが真のゲノムに由来する(Levy et al、2007)。そしてハプロタイプアセンブリでのフェージングのエラー率を低くするには10〜15のカバレッジで十分であることを示す。最先端のハプロタイプアセンブリアプローチと比較して、WhatsHapは、特に10〜15カバレッジのデータセット、すなわち重要なカバレッジレートに優れたランタイムを持つことを実証する。 WhatsHapによって計算されたハプロタイプはわずかな誤差しかないことが証明されている。統計的なフェージングアプローチと比較してもエラー率がかなり有利であることは注目に値するかもしれないが、依然として現実的な最先端技術を構成している。
マニュアル
https://whatshap.readthedocs.io/en/latest/guide.html
イルミナ、Nanopore、pacbioいずれのデータセットでも機能する。
インストール
Bitbucket
https://bitbucket.org/whatshap/whatshap
condaによるインストールと、pipによるインストールがサポートされている。
#Anacondaを使っているなら
conda install -c bioconda -c conda-forge whatshap
>whatshap -h
$ whatshap -h
usage: whatshap [-h] [--version] [--debug]
{phase,stats,compare,hapcut2vcf,unphase,haplotag,genotype} ...
positional arguments:
{phase,stats,compare,hapcut2vcf,unphase,haplotag,genotype}
phase Phase variants in a VCF with the WhatsHap algorithm
stats Print phasing statistics of a single VCF file
compare Compare two or more phasings
hapcut2vcf Convert hapCUT output format to VCF
unphase Remove phasing information from a VCF file
haplotag Tag reads by haplotype
genotype Genotype variants
optional arguments:
-h, --help show this help message and exit
--version show program's version number and exit
--debug Print debug messages
> whatshap phase
$ whatshap phase
usage: whatshap phase [-h] [-o OUTPUT] [--reference FASTA] [--tag {PS,HP}]
[--output-read-list FILE]
[--algorithm {whatshap,hapchat}] [--merge-reads]
[--max-coverage MAXCOV] [--mapping-quality QUAL]
[--indels] [--ignore-read-groups] [--sample SAMPLE]
[--chromosome CHROMOSOME]
[--error-rate READ_MERGING_ERROR_RATE]
[--maximum-error-rate READ_MERGING_MAX_ERROR_RATE]
[--threshold READ_MERGING_POSITIVE_THRESHOLD]
[--negative-threshold READ_MERGING_NEGATIVE_THRESHOLD]
[--full-genotyping] [--distrust-genotypes]
[--include-homozygous] [--default-gq DEFAULT_GQ]
[--gl-regularizer GL_REGULARIZER]
[--changed-genotype-list FILE] [--ped PED/FAM]
[--recombination-list FILE] [--recombrate RECOMBRATE]
[--genmap FILE] [--no-genetic-haplotyping]
[--use-ped-samples]
VCF [PHASEINPUT [PHASEINPUT ...]]
Phase variants in a VCF with the WhatsHap algorithm
Read a VCF and one or more files with phase information (BAM/CRAM or VCF phased
blocks) and phase the variants. The phased VCF is written to standard output.
positional arguments:
VCF VCF file with variants to be phased (can be gzip-
compressed)
PHASEINPUT BAM, CRAM or VCF file(s) with phase information,
either through sequencing reads (BAM/CRAM) or through
phased blocks (VCF)
optional arguments:
-h, --help show this help message and exit
-o OUTPUT, --output OUTPUT
Output VCF file. Add .gz to the file name to get
compressed output. If omitted, use standard output.
Reference file. Provide this to detect alleles through
re-alignment. If no index (.fai) exists, it will be
created
--tag {PS,HP} Store phasing information with PS tag (standardized)
or HP tag (used by GATK ReadBackedPhasing) (default:
PS)
--output-read-list FILE
Write reads that have been used for phasing to FILE.
--algorithm {whatshap,hapchat}
Choose an algorithm from whatshap or hapchat (default:
whatshap)
Input pre-processing, selection and filtering:
--merge-reads Merge reads which are likely to come from the same
haplotype (default: do not merge reads)
--max-coverage MAXCOV, -H MAXCOV
Coverage reduction parameter in the core phasing
algorithm. Phasing quality may improve slightly if
this is increased, but runtime increases
exponentially! Do not increase beyond 20. (default:
15)
--mapping-quality QUAL, --mapq QUAL
Minimum mapping quality (default: 20)
--indels Also phase indels (default: do not phase indels)
--ignore-read-groups Ignore read groups in BAM/CRAM header and assume all
reads come from the same sample.
--sample SAMPLE Name of a sample to phase. If not given, all samples
in the input VCF are phased. Can be used multiple
times.
--chromosome CHROMOSOME
Name of chromosome to phase. If not given, all
chromosomes in the input VCF are phased. Can be used
multiple times.
Read merging:
The options in this section are only active when --merge-reads is used
--error-rate READ_MERGING_ERROR_RATE
The probability that a nucleotide is wrong in read
merging model (default: 0.15).
--maximum-error-rate READ_MERGING_MAX_ERROR_RATE
The maximum error rate of any edge of the read merging
graph before discarding it (default: 0.25).
--threshold READ_MERGING_POSITIVE_THRESHOLD
The threshold of the ratio between the probabilities
that a pair of reads come from the same haplotype and
different haplotypes in the read merging model
(default: 1000000).
--negative-threshold READ_MERGING_NEGATIVE_THRESHOLD
The threshold of the ratio between the probabilities
that a pair of reads come from different haplotypes
and the same haplotype in the read merging model
(default: 1000).
Genotyping:
The options in this section require that either --distrust-genotypes or --full-genotyping is used
--full-genotyping Completely re-genotype all variants based on read
data, ignores all genotype data that might be present
in the VCF (EXPERIMENTAL FEATURE).
--distrust-genotypes Allow switching variants from hetero- to homozygous in
an optimal solution (see documentation).
--include-homozygous Also work on homozygous variants, which might be
turned to heterozygous
--default-gq DEFAULT_GQ
Default genotype quality used as cost of changing a
genotype when no genotype likelihoods are available
(default 30)
--gl-regularizer GL_REGULARIZER
Constant (float) to be used to regularize genotype
likelihoods read from input VCF (default None).
--changed-genotype-list FILE
Write list of changed genotypes to FILE.
Pedigree phasing:
--ped PED/FAM Use pedigree information in PED file to improve
phasing (switches to PedMEC algorithm). Columns 2, 3,
4 must refer to child, mother, and father sample names
as used in the VCF and BAM/CRAM. Other columns are
ignored.
--recombination-list FILE
Write putative recombination events to FILE.
--recombrate RECOMBRATE
Recombination rate in cM/Mb (used with --ped). If
given, a constant recombination rate is assumed
(default: 1.26cM/Mb).
--genmap FILE File with genetic map (used with --ped) to be used
instead of constant recombination rate, i.e. overrides
option --recombrate.
--no-genetic-haplotyping
Do not merge blocks that are not connected by reads
(i.e. solely based on genotype status). Default: when
in --ped mode, merge all blocks that contain at least
one homozygous genotype in at least one individual
into one block.
--use-ped-samples Only work on samples mentioned in the provided PED
file.
whatshap phase: error: the following arguments are required: VCF, PHASEINPUT
ラン
whatshapのphaseコマンドは以下のような処理を行う。
これはSample1とSample2のマージしたvcfファイルだが、samle1だけ、GT(genotype)フィールドが5つのアレルすべてでヘテロ(0/1)になっている。
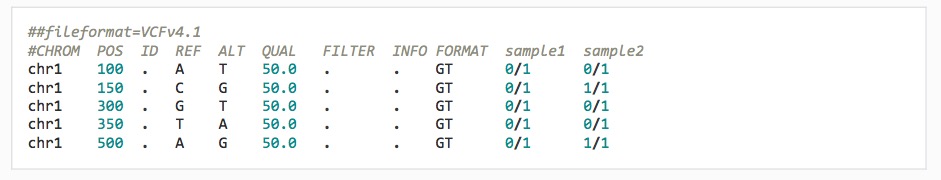
公式より転載
phaseコマンドはGTフィールドが0/1のものをフェーズがそろうよう並べ替えて、/ のかわりにパイプ( | )で区切る。Sample1の1つのハプロタイプはA, G, T, T, A、もう片方のハプロタイプはT, C, G, A, Gと推定した(0/1ではヘテロとしかわからない。|で区切ることで、REFかALTか明確にしている)。

公式より転載
phasingを行うには、バリアントコーラーが出力したVCFと、マッピングしたbamファイルが必要になる。 phaseコマンドを使う。
whatshap phase -o phased.vcf input.vcf mapped.bam
1000genomesのデータを3つ解析してみる。サンプル名は通し番号とする。
シェルスクリプト variant_call.sh
#!/bin/sh
#複数のbamを順に処理する。
#依存ツール
#BBtools - interleave fastqの作成
#sickle - quality trimming
#SAMTools - sam=>bam、ソート、index
#BWA MEM - mapping
#vcftools - VCFのマージ
#samblaster
#パラメータ
thread=20 #スレッド数
#保存場所
#全サンプルのfastq: ~/fastq/ #ペアエンド名はstrain通し番号_pair1/2.fq.gz (e.g., strain1_pair1.fq.gz & strain1_pair2.fq.gz)
#interleave_fastq: ~/interleave_fastq/
#reference.fasta: ~/ref/
#bam: ~/bam/
#VCF: ~/vcf/
#indexを作る。
ref=~/ref/Homo_sapiens.GRCh38.dna.chromosome.fa
bwa index $ref #isはたしか2GBまで
samtools faidx $ref
#3サンプルのループ処理
sample=(1 2 3)
for name in ${sample[@]}; do
echo sample$name
r1=strain${name}_pair1.fq.gz
r2=strain${name}_pair2.fq.gz
#interleaveのfastq.gz作成#interleaveのfastq.gz作成
reformat.sh in1=$r1 in2=$r2 out=~/interleave_fastq/${name}_interleave.fq.gz
fastq=~/interleave_fastq/${name}_interleave.fq.gzfastq=~/interleave_fastq/${name}_interleave.fq.gz
sleep 1s
#quality trimming
sickle pe -c $fastq -g -t sanger -l 20 -q 20 -m ~/interleave_fastq/${name}_interleave_qc.fq.gz -s single.fq.gz && rm single.fq.gz
qcfastq=~/interleave_fastq/${name}_interleave_qc.fq.gz
#mapping samblasrにパイプしてduplicationは捨てる
bwa mem -R "@RG\tID:1a\tLB:library1\tSM:Sample${name}\tPL:ILLUMINA" -t $thread $ref $qcfastq | samblaster -r -e -d samp.disc.sam -s samp.split.sam |samtools view -@ $thread -bS - > ${name}.bam
samtools sort -@ $thread ${name}.bam > ~/bam/${name}_sorted.bam && rm ${name}.bam #ソート
samtools index ~/bam/${name}_sorted.bam #bamのindex
freebayes -f $ref -C 5 -p 2 ~/bam/${name}_sorted.bam > ~/vcf/${name}_output.vcf && #バリアントのコール
bgzip -c ~/vcf/${name}_output.vcf > ~/vcf/${name}_output.vcf.gz && rm ~/vcf/${name}_output.vcf #vcfの圧縮
tabix -p vcf ~/vcf/${name}_output.vcf.gz #vcfのindex
done
GATKは遅すぎるのでfreebayesを使った。1_sorted.bam、2_sorted.bam、3_sorted.bam、とそのvcfファイルである1_output.vcf.gz、2_output.vcf.gz、3_output.vcf.gzが得られた。
VCFのマージ。
vcf-merge 1_output.vcf.gz 2_output.vcf.gz 3_output.vcf.gz | bgzip -c > merge.vcf.gz
merge.vcf.gzが出力される。
Sample1のphasingを行う。 WhatsHapのphaseコマンドを使う。ロングリードならリファンレスを与えて精度をあげる必要がある(see manual)。Sample1のchromosome1のみに限定する。
whatshap phase --indels --sample SampleA --chromosome 1 -o phased.vcf merge.vcf.gz 1_sorted.bam
- --max-coverage Reduce coverage (default: 15).
- --mapping-quality Minimum mapping quality (default: 20)
- --indels Also phase indels (default: do not phase indels)
- --ignore-read-groups Ignore read groups in BAM header and assume all reads come from the same sample.
- --sample Name of a sample to phase. If not given, all samples in the input VCF are phased. Can be used multiple
- --chromosome Name of chromosome to phase. If not given, all chromosomes in the input VCF are phased. Can be used multiple times.
phased.vcfが出力される。
gz圧縮してindexをつける。
bgzip phased.vcf
tabix phased.vcf.gz
フェーズ分析結果をもとにハプロタイプの配列を出力。
bcftools consensus -H 1 -s Sample1 -f Homo_sapiens.GRCh38.dna.chromosome.fa phased.vcf.gz > haplotype1.fa
bcftools consensus -H 2 -s Sample1 -f Homo_sapiens.GRCh38.dna.chromosome.fa phased.vcf.gz > haplotype2.fa
フェーズ分析結果をもとにGTFを出力。phasingされた数などのstatisticsも標準出力される。
whatshap stats --sample SampleA --chromosome 1 --indels --gtf=phased.gtf phased.vcf.gz
- --gtf Write phased blocks to GTF file.
- --sample Name of the sample to process. If not given, use first sample found in VCF.
- --only-snvs Only process SNVs and ignore all other variants.
- --chromosome Name of chromosome to process. If not given, all chromosomes in the input VCF are considered.
IGVに読み込む。
igv -g Homo_sapiens.GRCh38.dna.chromosome.fa phased.vcf.gz,phased.gtf,1_sorted.bam

他にも2つ以上のphasing解析結果を比較するcompareなどのコマンドがある。
引用
WhatsHap: Weighted Haplotype Assembly for Future-Generation Sequencing Reads.
Patterson M, Marschall T, Pisanti N, van Iersel L, Stougie L, Klau GW, Schönhuth A
J Comput Biol. 2015 Jun;22(6):498-509.