次世代シークエンシングは疾患関連遺伝子変異体の発見を容易にし、ルーチンの臨床診療における遺伝子診断に広く使用されている。Variant call format(VCF)は、医療遺伝学の研究および診断からの遺伝子変異データを報告するためのコミュニティ標準となっている(論文より ref.1)。 VCFファイルは、示されたリファレンスゲノムアセンブリからの逸脱として各変異体を記述する。標準列には、リファレンスゲノムにおける変異の位置、参照と異なる塩基、および変異がコールされた統計的信頼度が含まれる。追加の注釈は、dbSNP(ref.2)、ClinVar(ref.3)、ExAC(ref.4)などのパブリックデータベースに基づいて、またはSnpEffなどのバリアント注釈ツールを使用して計算された、INFO列のキーと値のペアとして(ref.5)、ANNOVAR(ref.6)、およびMutationTaster(ref.7)が含まれる。頻繁に使用されるカスタム注釈には、健常な個体および罹患した個体における変異の対立遺伝子頻度ならびに変異の有害な影響の予測が含まれ、これは所与の変異が病原性であるか否かを決定するのに役立ち得る(ref.8)。
医学の遺伝学者は、患者で観察された遺伝的変異をフィルタリングし、優先順位を付けるために他の家族のメンバーおよび関連コホート研究からの追加データを考慮に入れるため追加注釈を使用することが多い。 VCFファイルは、原則としてテキストエディタやスプレッドシートソフトウェアで表示できるテキストファイルだが、VCF形式では複雑なヘッダーと、正確で効率的なバリアントフィルタリングのための専用ツールが必要な内部データ構造が定義されている。さらに、VCFファイルは、Microsoft Excelを含む現行のスプレッドシートソフトウェアの限界を超えて非常に大きい可能性がある(ゲノムシーケンシングデータ全体で作業する場合、1サンプルあたり数百万行と数百MB)。したがって、理想的にはユーザフレンドリーで効率的なグラフィカルインターフェースの下で、VCFフィルタリングおよび注釈を有する医学遺伝学者を支援する専用のソフトウェアツールが必要とされている。
現在、VCF-Miner(ref.9)とBrowseVCF (ref.10)といったカスタムアノテーションを使用した遺伝子変異のフィルタリングと優先順位付けに特化した2つの非商用ソフトウェアツールがあるが、両方のツールには、多数のサンプルを含むファミリーまたはコホートで作業する場合に制限がある。 GEMINI(ref.11)、gNOME(ref.12)、BiERapp(ref.13)など、あらかじめ定義されたアノテーションセットに基づいてVCFファイルをフィルタリングするための追加ツールがあるが、これらのソフトウェアパッケージはフィルタリングルールや優先順位付けワークフローの柔軟性が低くなる。最後に、複数のコマンドラインツールがVCFバリアントフィルタリング(ref.1,11,14-16)を提供している。自動化されたワークフローには役立つが、インタラクティブな分析には適していない(論文 Supplementary1. VCFデータフィルタリングのための無料の学術ソフトウェアツールのリスト)。
著者らは、VCF.Filterを、GNU GPL v3のオープンソースライセンスの下で自由かつオープンに利用できる使いやすいスタンドアロンのグラフィカルソフトウェアツールとして開発した。 VCF.Filterは、VCF形式(最近リリースされたVCFバージョン4.2を含む)、カスタム注釈、大きなVCFファイル、および柔軟な分析タイプを幅広くサポートしている。 VCF.Filterは、インデックス付きのVCFファイルを入力として受け取り、デフォルトおよびカスタムバリアントアノテーションを使用して、複雑なフィルタチェーンをインタラクティブに定義、実行、保存することができる。出力は、タブで区切られたフィールドのカスタマイズ可能なリストで、表示、コピー(たとえばスプレッドシートへの)、またはテキストファイルとしての保存が可能になっている。結果は、新しいVCFファイルに直接書き込むこともできる。 VCF.Filterは、医学遺伝学者と緊密に連携して開発され、広範にテストされている。これは、広く使用されているGATKソフトウェア(ref.17)(フロー紹介、ただしGATKには、ガン専用のもっと高感度なやり方もある。GATK自体も最近version4になりbest practiceも更新された)上に構築されたバイオインフォマティクス処理パイプラインによって得られたVCFファイルをフィルタリングするために日常的に使用されている。

公式サイトより転載
https://biomedical-sequencing.at/VCFFilter/downloads/Tutorial.pdf
インストール
依存
- Java version 1.8
本体
https://biomedical-sequencing.at/VCFFilter/
zipファイルをダウンロードして解凍する。
ラン
以下のコマンドを打つと起動する。
java -Xmx1024m -Duser.language=US -jar VCFFilter.jar

Openをクリックし、exampleのindex付きVCFのどれかを指定すると、右のStart VCFFilterがクリックできるようになる。
次の画面

VCFのFilterling、ファミリー解析、バリアントの解析の3つが行える。まず左端のタブのVCFのFilterlingを行なってみる。上の画面のInput VCFからexampleのVCFを入力する。
右端下のRemoveボタンをクリックすれば、最初から表示されているカラムを消すことができる。Removeボタンの真上のAddボタンを押し、新しくカラムを追加する。

条件が追加された。

使用したVCFはこんな内容になっている。

chr1と3だけ抽出するには1と3を指定する。

緑のRunボタンを押す。
下のウィンドウ内に1と3だけ出力された。

右上の端のtotalボタンをクリックすると、heteroかhomoだけ選択もできる。
上のInclusion listsは、出力する領域を指定したbedファイルを指定する。

付属のlist files/のchr1とchr2を指定してRun(Ctrl+click)。

出力が指定のbedファイル領域に限定された。

その隣のExclusionでは、指定したbedファイルの領域を出力から除外できる。一般的には既知変異、変異のホットスポット、既知アセンブリエラー領域などを指定する。

右端は、コホート研究(wiki)のtsvファイルを指定する(VCFFilter_with_sample_data/cohort_frequency_files/)。


ファミリー解析では、病気に関連する recessive variants、dominant variants,、compound heterozygous variants、 X-linked variants,de novo mutationsを解析できる。詳細はチュートリアルで確認してください。

出力

Filterling条件も追加することができる。
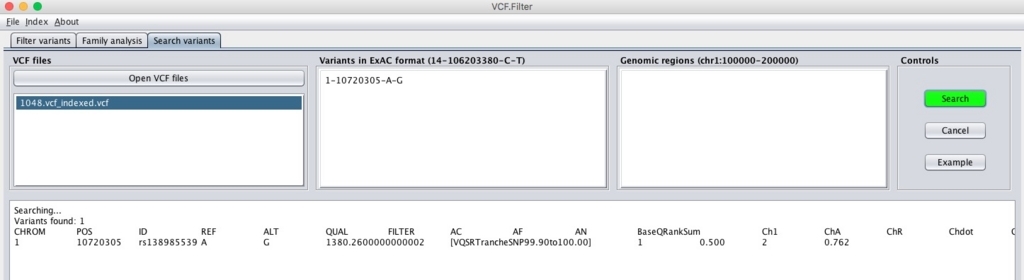
Search variantsのタブでは、コール部位を検索できる。 e.g. 1-10720305-A-G
引用
VCF.Filter: interactive prioritization of disease-linked genetic variants from sequencing data
Müller H, Jimenez-Heredia R, Krolo A, Hirschmugl T, Dmytrus J, Boztug K1, Bock C
Nucleic Acids Res. 2017 Jul 3;45(W1):W567-W572.