2019 3/8 タイトル修正 図追加
2021 6/5 追記
16q rRNAアンプリコンとショットガンメタゲノムシーケンシングは、健康や病気に関するヒトマイクロバイオーム研究に広範に使われている[prepirntより ref.1, 2] 。私たちはその後、天然に存在する微生物群集は、生物多様性の広範な範囲をカバーしていることを学んだ。おそらくhalf a dozenのレベルから1万を超える微生物のpopulationsレベルの微生物多様性を含むことができ、代表的な分類群は大きく異なる可能性がある[ref.9-12]。これらの多様なコミュニティを分析することは困難である。この問題は、データ生成における幅広い実験設定の使用と、短期および長時間のシークエンシング技術の急速な進化によって悪化する[ref.13,14]。生成されるデータが非常に多様であるため、特定の実験設定に対する現実的なベンチマークデータセットを生成する可能性は、計算メタゲノミクスソフトウェアを評価するために不可欠である。
メタゲノム解釈のクリティカルアセスメントのinitiative であるCAMIは、コンピュテーションメタゲノムソフトウェアの幅広く客観的なパフォーマンス概要を生成することを目的としたコミュニティの取り組みである[rwef.15]。 CAMIはベンチマーキングの課題を編成し、データの生成、ソフトウェアの適用、結果の解釈[ref.16]など、すべての面で標準の開発と再現性を促進する。
ここでは、最初にCAMIの最初のチャレンジで使用されたシミュレートされたメタゲノムデータセットを生成するために書かれたCAMISIMについて説明する。 CAMISIMの有用性をいくつかのアプリケーションで実証する。著者らは、ヒトおよびマウスの腸内微生物のtaxonomyプロファイルから、複雑で多重反復のベンチマークデータセットを生成した[ref.1, 17]。数千もの小さな“minimally challenging metagenomes” をシミュレートし、ポピュラーなMEGAHIT [ref.18](紹介)やmetaSPAdes [ref.19]アセンブラ(紹介)で、シーケンシングカバレッジ、ゲノムの進化的な相違、シーケンシングエラープロファイルなどの変化に伴う影響を特徴付けた。
CAMISIMのワークフロー。 Preprintより転載。
マニュアル
https://github.com/CAMI-challenge/CAMISIM/wiki/User-manual
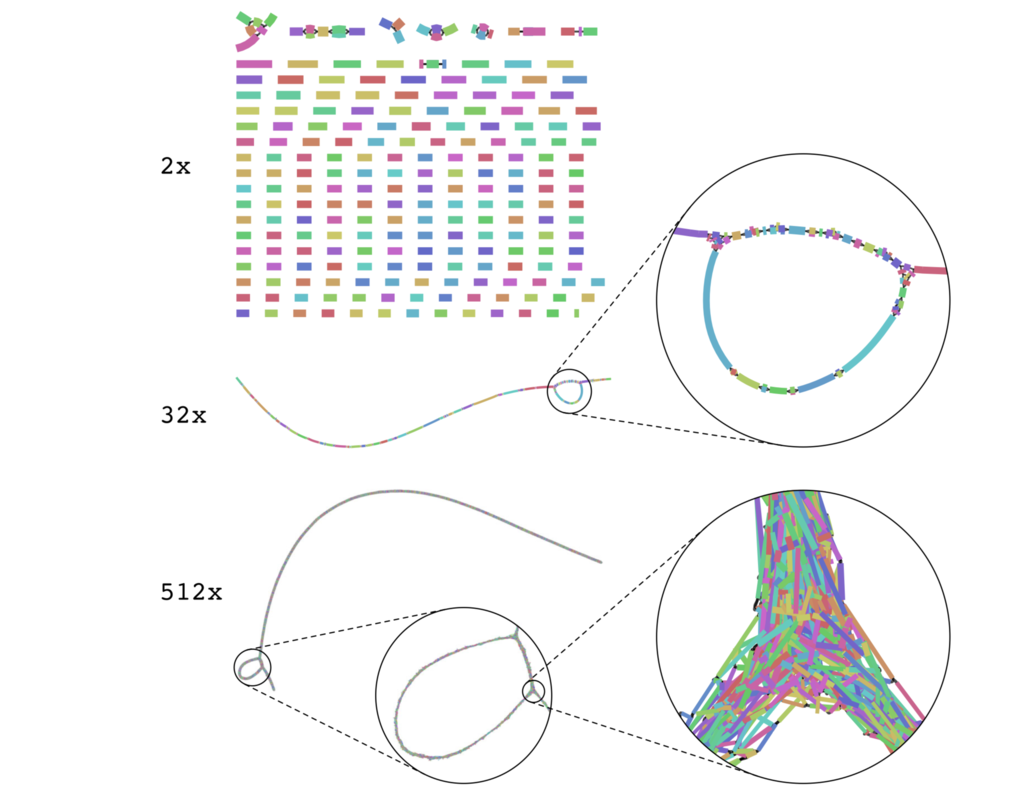
Assembly graphs become more complex as coverage increases.
論文より転載
インストール
依存
python 2.7.10
- Biopython
- BIOM
- NumPy
- Matplotlib
Genome annotation
- Hmmer3 or RNAmmer 1.2(RNAmmer is a wrapper of Hmmer2. Hmmer uses hidden markov profiles to search marker genes in sequences.)
- Mothur(A multi tool program. Alignment of sequences and clustering.)
- MUMmer(A genome alignment software)
Perl 5
- XML::Simple
Simulation
- ART(ART is a set of simulation tools to generate synthetic next-generation sequencing reads.)
- wgsim(Read simulator which offers error-free and uniform error rates.)
- NanoSim(Read simulator for the generation of Oxford Nanopore Technologies (ONT) reads. )
- PBsim(Read simulator for generating Pacific Biosciences (PacBio) reads.)
- SAMtools 1.0
本体 Github
依存が多いのでここではdockerコンテナを使う。
docker pull cami/camisim:latest
> python metagenomesimulation.py -h
docker run --rm -it -v /Users/user/Documents/docker_share/:/home/ cami/camisim metagenomesimulation.py -h
usage: python metagenomesimulation.py configuration_file_path
#######################################
# MetagenomeSimulationPipeline #
# Version 0.0.6 #
#######################################
Pipeline for the simulation of a metagenome
optional arguments:
-h, --help show this help message and exit
-v, --version show program's version number and exit
-silent, --silent Hide unimportant Progress Messages.
-debug, --debug_mode more information, also temporary data will not be deleted
-log LOGFILE, --logfile LOGFILE
output will also be written to this log file
optional config arguments:
-seed SEED seed for random number generators
-s {0,1,2}, --phase {0,1,2}
available options: 0,1,2. Default: 0
0 -> Full run,
1 -> Only Comunity creation,
2 -> Only Readsimulator
-id DATA_SET_ID, --data_set_id DATA_SET_ID
id of the dataset, part of prefix of read/contig sequence ids
-p MAX_PROCESSORS, --max_processors MAX_PROCESSORS
number of available processors
required:
config_file path to the configuration file
ERROR: 0
——
> python genomeannotation.py
$ docker run --rm -it -v /Users/user/Documents/docker_share/:/home/ cami/camisim genomeannotation.py -h
usage: python genomeannotation.py configuration_file_path
#######################################
# GenomeAnnotationPipeline #
# Version 0.0.6 #
#######################################
Pipeline for the extraction of marker genes, clustering and taxonomic classification
optional arguments:
-h, --help show this help message and exit
-v, --version show program's version number and exit
-verbose, --verbose display more information!
-debug, --debug_mode tmp folders will not be deleted!
-log LOGFILE, --logfile LOGFILE
pipeline output will written to this log file
optional config arguments:
-p MAX_PROCESSORS, --max_processors MAX_PROCESSORS
number of available processors
-s {0,1,2,3}, --phase {0,1,2,3}
0 -> Full run (Default)
1 -> Marker gene extraction
2 -> Gene alignment and clustering
3 -> Annotation of Genomes
required:
config_file path to the configuration file of the pipeline
——
> python metagenome_from_profile.py
ヘルプなし。
ラン
ホストの指定パスをinputとoutputとして認識してランする。初回はNCBI database("nodes.dmp", "merged.dmp", "names.dmp") をダウンロードするので時間がかかる。
docker run -it -v "/Volumes/test/CAMISIM/defaults:/input:rw" \
-v "/Volumes/test/CAMISIM/out:/output:rw" \
cami/camisim metagenome_from_profile.py \
-p /input/mini.biom -o /output
引用
CAMISIM: Simulating metagenomes and microbial communities.
Adrian Fritz, Peter Hofmann, Stephan Majda, Eik Dahms, Johannes Droege, Jessika Fiedler, Till R. Lesker, Peter Belmann, Matthew Z. DeMaere, Aaron E. Darling, Alexander Sczyrba, Andreas Bremges, Alice C. McHardy
bioRxiv, 300970. doi:10.1101/300970
参考
Critical Assessment of Metagenome Interpretation—a benchmark of metagenomics software
Alexander Sczyrb, Peter Hofmann, Alice C McHardy
Nature Methods volume 14, pages 1063–1071 (2017)
