2019 1/10 追記
Cyberinfrastructureは、直訳するとサイバー空間のインフラとなる。計算科学の分野では大規模な計算化学の課題に対する解決策を提供するもの、というような意味で使われている(wiki)。CyVerseはこのCyberinfrastructureを提供する、様々なインフォマティクスツールを実行可能なクラウド環境である。元々、米国植物科学コミュニティに貢献するために、iPlant Collaborativeという名前で2008年にNational Science Foundationによって作成されたのが始まりと記載されている。 2015年、iPlantは、ライフサイエンスへのより普遍的なサポートを行うため、CyVerseに改名された。 スーパーコンピューティング機能へのアクセスを民主化することで、科学者が将来のソリューションを見つけるための重要なリソースを提供している。
CyVerseについて
CyVerse wiki
https://pods.iplantcollaborative.org/wiki/dashboard.action
Webinar(50分)
Discovery Environmentにアクセスする。
LAUNCHをクリック。

log inして立ち上げる。初回はユーザー登録(Register now)をクリックしてIDを手に入れる。

ブラウザの中にデスクトップが出てきたような感じの環境が出現する。Data(macのFInderに相当)をクリックすると、ウィンドウが出現する。初回はUploadからローカルのファイルをアップロードする。

ログアウントしても再度ログインすれば、ウィンドウの配置も含め全て復元される。
Simple uploadでも2GBのファイルをuploadできる。例えばgz圧縮したfastqデータをuploadしてみる。

fastq.gzを指定してuploadをクリック。

左上のボタンAppsをクリックし、Apps選択ウィンドウを表示する。ウィンドウはクリックして移動したり拡大/縮小できる。知らなければ、もはやクラウドかローカルか分からない。gzと検索。

4つAppsが見つかった。並列化に対応したコマンドラインのgz解凍ツールのunpigzをクリック(圧縮ならpigz)。(コマンドラインのツールをGUIで操作できるので、これを”Apps"と呼んでいる)
unpigz解析ウィンドウが出てくるので、Inputをクリック=> アップロードしたgz圧縮ファイルを選択。必要ならOutputのパスも指定する。

Runをクリック。

進捗状況はAnalysisボタンから確認する。

終わるとcompletedになる。
指定した出力ディレクトリに解凍したfastqが出力されている。

ダブルクリックすると中身も表示できる。

de novo assemblyを行ってみる。Appsウィンドウでassemblyと検索。大量のツールがヒットした(まだ下にかなりスクロールできる)。

spadesと検索。複数バージョンが登録されている。ありがたい。
spades3.8を選択。inputにペアエンドのfastq.gzを指定した。

パラメータを指定する。コマンドラインと同じようなパラメータが指定できる。?をクリックすると簡単な説明も表示される。coverage cutoffを10とした。
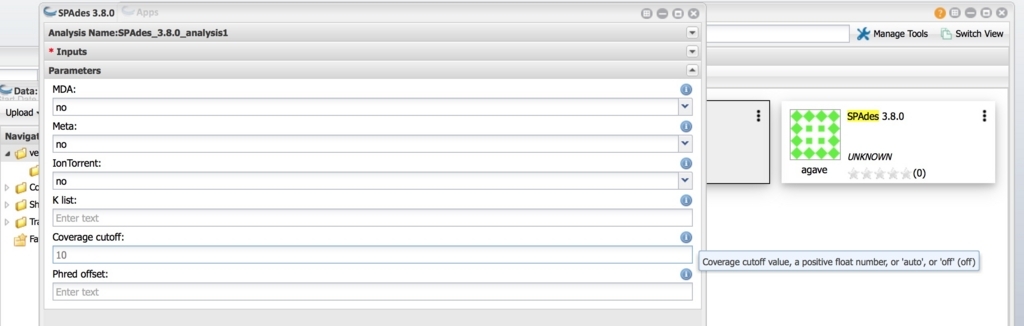
アセンブリなどの計算負荷の高いジョブはHigh performance computing(HPC)として扱われる。詳細はこちらに記載されている(リンク)が、HPCに属するジョブはアメリカの複数の学術機関のクラスタを使い分けてランされているらしい。HPCの並列処理を実行したりして1人でキューをたくさん発生させると(または混雑時など)、ラン開始まで数日かかる可能性がある。要はスパコンの利用時と同じである。
 HPCジョブは開始まで時間がかかるため、エラーと勘違いしてラン前に何度も同じジョブを発生させてはいけない(当然ますます遅くなる)。
HPCジョブは開始まで時間がかかるため、エラーと勘違いしてラン前に何度も同じジョブを発生させてはいけない(当然ますます遅くなる)。
終わると該当フォルダにアセンブリされた配列が出力される。出力されるファイルはコマンドラインでspadesを実行したときと変わらない。
大量のツールが登録されている。
mappingで検索。

indelで検索。
SNPで検索。
metagenomeで検索。

long readで検索。

genomeで検索。

RNAで検索。

bedで検索。

taxonで検索。

statisticsで検索。

bacteriaで検索。

filterで検索。

gwasで検索。

chipで検索。
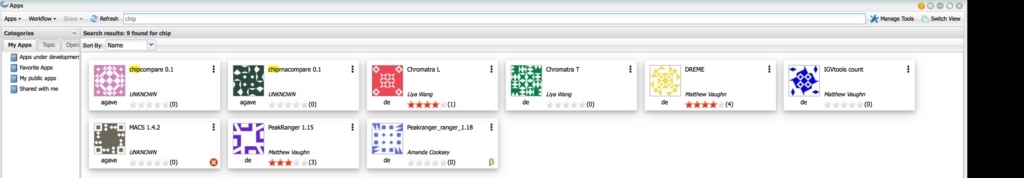
errorで検索。

trimmingで検索。

分野によって数に大きな偏りはあるが、このようにNGSの主流の解析ツールなら非常に充実している。TopicからもAppsは探索できる。

joveで、CyVerseを使いtranscriptome解析を行う論文が発表されている。ビデオ学術誌なので動画があります。5分で概要をつかめます。
Appsを組み合わせて自動で進めるワークフローも構築できるようです。詳細はWebinarやwikiでチェックしてください。
https://pods.iplantcollaborative.org/wiki/dashboard.action
追記1
GB以上の実験データをいきなり使うと時間がかかるので、最初はコントロールとなるようなファイルの小さいテストデータで試してみてください。
追記2
- RNA-seq Tutorial- STAR, StringTie and DESeq2
https://pods.iplantcollaborative.org/wiki/display/TUT/RNA-seq+Tutorial-+STAR%2C+StringTie+and+DESeq2
- RNA-Seq with Kallisto and Sleuth
https://cyverse-kallisto-tutorial.readthedocs-hosted.com/en/latest/
追記3
データベースや巨大な配列をダウンロードする。
Apps => Curlを使う。
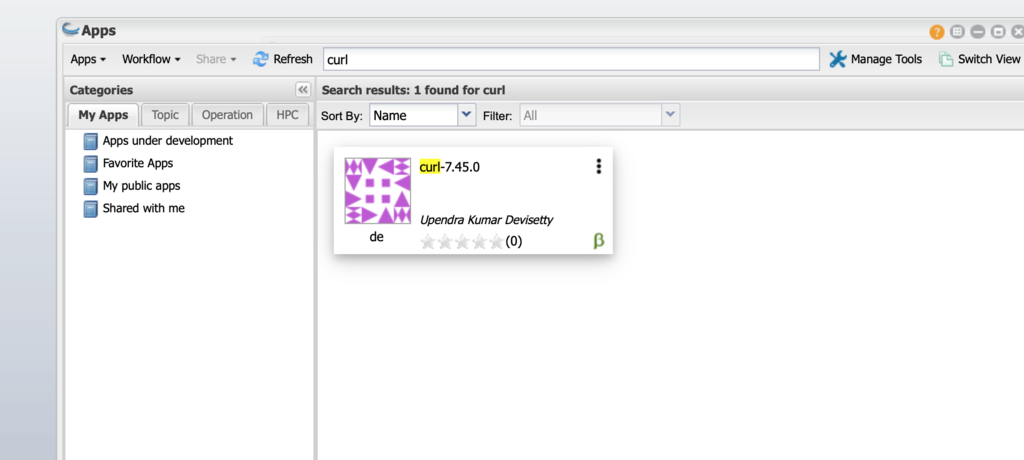
URLを指定、実行する。

ジョブがスタートしたかどうかは右上のベルでチェック

ダウンロードが終わったら解凍する。Apps => uncompress files with gunzipを選択

引用
Bringing numerous methods for expression and promoter analysis to a public cloud computing service.
Polanski K, Gao B, Mason SA, Brown P, Ott S, Denby KJ, Wild DL
Bioinformatics. 2018 Mar 1;34(5):884-886.
Leveraging CyVerse Resources for De Novo Comparative Transcriptomics of Underserved (Non-model) Organisms
Blake L. Joyce, Asher K. Haug-Baltzel, Jonathan P. Hulvey, Fiona McCarthy, Upendra Kumar Devisetty, Eric Lyons
J Vis Exp. 2017 May 9;(123). doi: 10.3791/55009.
関連
