Genome 10K Project(論文より ref.1)、 Bird 10,000 Genomes (B10K) Project(ref. 2)、i5k: Sequencing Five Thousand Arthropod Genomes Project(ref.3)など、様々な大規模ゲノムプロジェクトの成果とともに、様々な種から大量のゲノム配列が蓄積している。これらのゲノム配列は、通常、NCBI refseqデータベース(4)、JGI GOLDデータベース(ref.5)、UCSCゲノムブラウザデータベース(ref.6)およびEnsembl Genomes(ref.7)などの公的データベースに保存される。それらは、異なる種間のゲノム類似性、ならびにそのような類似性の機能的帰結を明らかにするために使用されてきた。比較ゲノミクス研究は、分子多様性とゲノム進化の分子レベルのメカニズムのより良い理解に貢献している(ref.8-12)。
複数種間で保存されているゲノム領域であるシンテニーブロックは、比較ゲノミクスにおいて中心的な役割を果たしている。 CYNTENATOR(ref.13)、DRIMM-Synteny(ref.14)、i-ADHoRe 3.0(ref.15)、inferCars(ref.16)、OSfinder(ref.17)、Sibelia(ref.18)などのシンメニーブロックの構築と利用のためおのたくさんのツールが報告されてきた。これらはスタンドアロンでコマンドラインのツールであり、ユーザーはローカルマシンにダウンロードしてインストールする必要がある。したがって、このようなツールを使用したシンテニーブロックの構築には、コンピュータリソース、高度なバイオインフォマティクススキルが必要であり、入力データを適切に準備し、コマンドラインプログラムを実行する必要がある。これらの困難を軽減するために、シンテニーブロックを視覚化するより使いやすいツールが開発されている。たとえば、GenomeMatcher(ref.19)、Mauve(ref.20)(紹介)、MizBee(ref.21)、およびSyMap(ref.22)は、グラフィカルユーザーインターフェイスをサポートするスタンドアロンのツールである。 Gbrowse_syn(ref.23)とSybil(ref.24)は、ユーザーのマシンにインストールする必要のある比較ゲノミクス用のWebベースのソフトウェアパッケージである。 GSV(ref.25)およびmGSV(ref.26)は、ユーザーが提供するシンテシー情報を視覚化するのに役立つ、Webベースのシンテシービューアである。 Cinteny(ref.27)(紹介)は、シンテニーブロックを同定し、ゲノム再編成を分析するためのWebサーバーである。 CoGe(ref.28)、VISTA(ref.29)およびEnsembl SyntenyView(ref.30)は、比較ゲノム研究のためのさまざまな分析を実行するWebベースのプラットフォームである。 Genomicus(ref.31)は、異なる種の相同なゲノムコンテクストを表示するウェブブラウザである。 C-Sibelia(ref.32)は、変異の注釈とともに、参照ゲノムとアセンブリを比較するためのWebサーバーである。 GRIMM-Synteny(ref.33)は、数字で表されるゲノムマーカーのリストを使用してゲノム再編成を分析するためのウェブサーバーである。しかし、それぞれのツールに異なる欠点があり、例えば、ペアワイズ比較のみのサポート、ユーザによるシンテシー情報の生成ステップ、視覚化のための限定された機能、高品質画像の保存またはダウンロード(ツール比較のための補足データ)をサポートしない、などの問題が依然として存在する。
著者らは、既存のツールの限界を補うことで、シンジニーブロックの構築、可視化、ブラウジングを可能にする多用途のWebベースのアプリケーションポータルであるSynteny Portalを報告した。 Synteny Portalを使用すると、ユーザーは簡単に、(i)UCSCゲノムブラウザデータベース(ref.6)内の予め作成されたアラインメントを使用して複数の種間でシンテニーブロックを構築し、(ii)シンテニーリレーションシップを高品質画像として視覚化してダウンロード、(iii) 遺伝子情報とともにシンテニーブロックをブラウズ、 (iv)下流のシンテニーベースの解析のための入力として使用できるシンテニーブロックの詳細をダウンロード、の解析を直観的で使いやすいWebインターフェイスですべて行うことができるようになっている。
--SynCircos--
マニュアル
http://bioinfo.konkuk.ac.kr/synteny_portal/img/documentation/SynCircos/SynCircos_doc.pdf
生物とクロモソームを選択してSubmitを押す。3生物以上の同時比較も可能だが、染色体数が20を越えると時間がかかる。
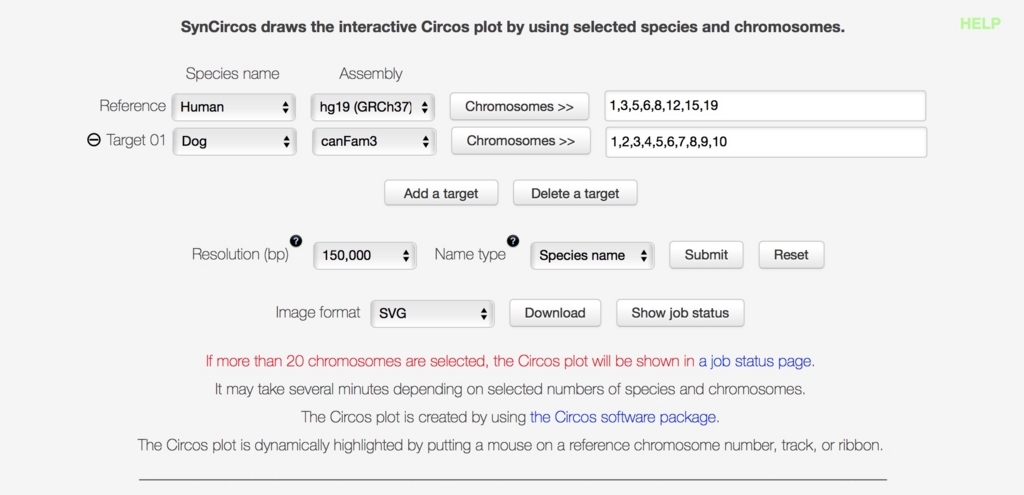
ヒト、イヌ、ネコのゲノムを比較してみる。
結果。

--SynBrowser--
マニュアル
http://bioinfo.konkuk.ac.kr/synteny_portal/img/documentation/SynBrowser/SynBrowser_doc.pdf
SynCircosと機能は似ている。クロモソームを比較し、submitする。

結果が表示される。

シンテシーブロック部分にカーソルを合わせると該当領域の遺伝子が表示される。

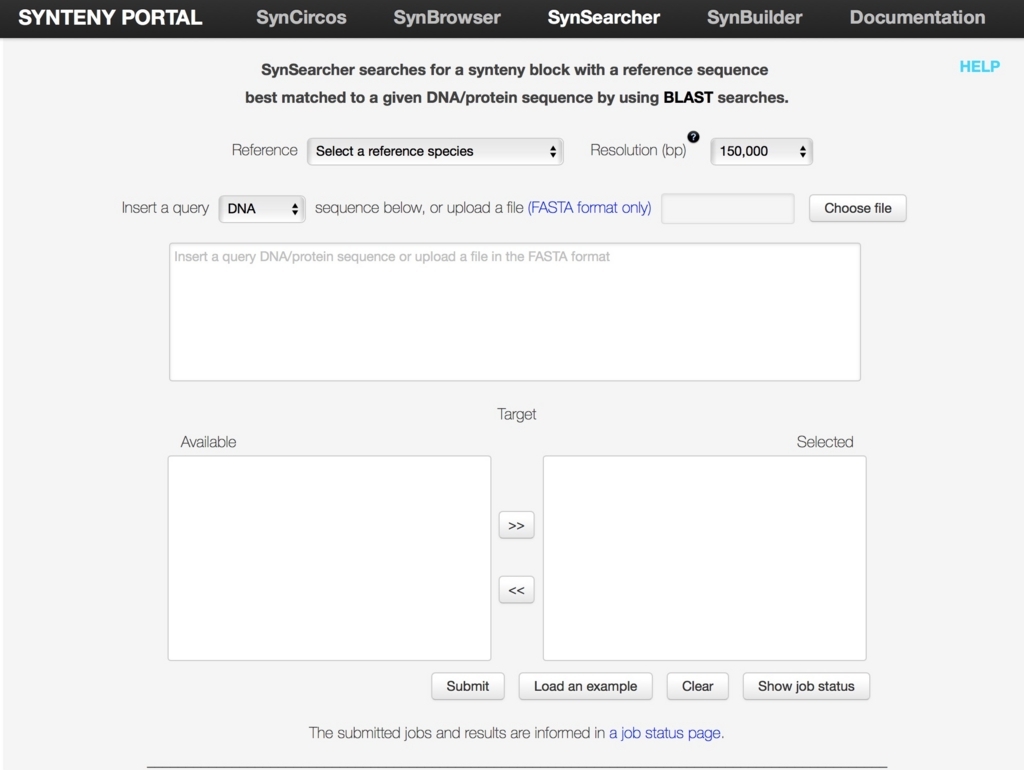
--SynSearcher--
マニュアル
http://bioinfo.konkuk.ac.kr/synteny_portal/img/documentation/SynSearcher/SynSearcher_doc.pdf
ユーザーが選択した複数種のシンテシーブロックを構築するSynBuilderもある。
マニュアル
http://bioinfo.konkuk.ac.kr/synteny_portal/img/documentation/SynBuilder/SynBuilder_doc.pdf
わかりやすいマニュアルが用意されているので、読みながらテストしてみてください。
引用
Synteny Portal: a web-based application portal for synteny block analysis.
Lee J, Hong WY, Cho M, Sim M, Lee D, Ko Y, Kim J
Nucleic Acids Res. 2016 Jul 8;44(W1):W35-40.
