bamsurgeonはガンの原因となる体細胞突然変異をシミュレートするために構築されたbamに対する変異導入ツール。ユーザーが用意したリストを元にして、bamに不完全な変異や構造変化を引き起こす大きな変異を導入することができる。2015年にnature methodsに発表された。
マニュアル
https://hpc.nih.gov/apps/bamsurgeon/Manual.pdf
インストール
https://github.com/adamewing/bamsurgeon
git clone https://github.com/adamewing/bamsurgeon.git
python setup.py build
sudo python setup.py install
ラン
塩基置換の導入
SNV変異をbamに導入する (test/)。
addsnv.py -v random_snvs.txt -f testregion_bwamem_realign.bam -r Homo_sapiens_chr22_assembly19.fasta -o change.bam
#リストについてはもう少し下の方に記載。
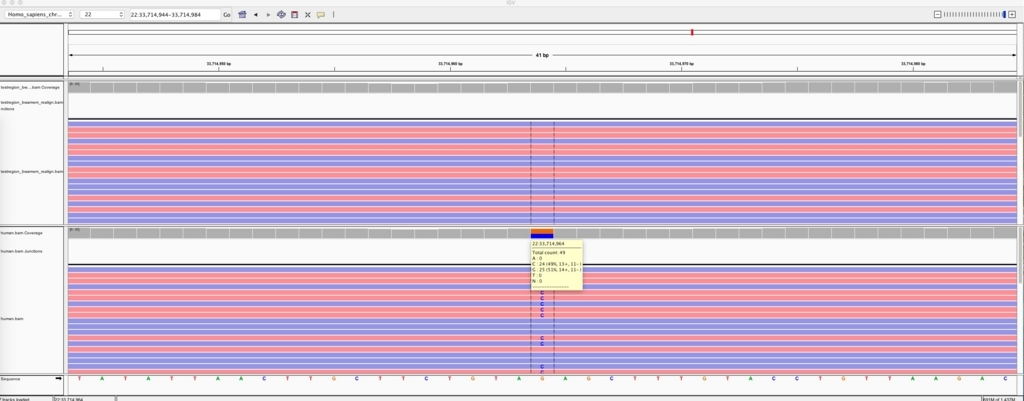
手術したbam出力を確認する。
igv -g Homo_sapiens_chr22_assembly19.fasta testregion_bwamem_realign.bam,change.bam
22 33714964 33714965 0.5 50%変異のはず(下が改変後)。
- -v Target regions to try and add a SNV, as BED
- --mindepth minimum read depth to make mutation (default = 10)
- --maxdepth maximum read depth to make mutation (default = 2000)
- -m allelic fraction at which to make SNVs (default = 0.5)
- -f sam/bam file from which to obtain reads
- -r reference genome, fasta indexed with bwa index -a stdsw _and_ samtools faidx
- -o .bam file name for output
- -s maximum allowable linked SNP MAF (for avoiding haplotypes) (default = 1)
- --ignoresnp make mutations even if there are non-reference alleles sharing the relevant reads
- --ignoreref make mutations even if the mutation is back to the reference allele
- --force force mutation to happen regardless of nearby SNP or low coverage
- --single input BAM is simgle-ended (default is paired-end)
- --maxopen maximum number of open files during merge (default 1000)
- --requirepaired skip mutations if unpaired reads are present
- -d allow difference in input and output coverage (default=0.9)
- -n maximum number of mutations to try (default: entire input)
#リストについて
SNVのリストは以下のようなフォーマットで準備する。1-3列目はBEDフォーマットと同じである。4列目はなくても動くが、0-1の数値を入れることで変異の割合を指定できる。4列目がなければ50%となる。
user$ head random_snvs.txt
22 34166720 34166721 1.0
22 33908770 33908771 0.15
22 33714964 33714965 0.5
22 33769483 33769484 0.75
22 33958087 33958088 0.01
22 34264774 34264775 0.25
22 33702084 33702085 0.2
22 34141184 34141185
22 33926586 33926587
BEDOPSを使えば、VCFから作ることができる。
フォーマット変換 VCF, GTF, GFF => BED - macでインフォマティクス
vcf2bed --snvs < input.vcf > SNV.bed
cut -f 1,2,3,4,6 SNV.bed |sed -e 's/\./0\.5/g' > SNV.txt
出力
user$ head -5 SNV.txt
Chromosome 198597 198598 0.5 C
Chromosome 387005 387006 0.5 C
Chromosome 528665 528666 0.5 G
Chromosome 609078 609079 0.5 T
Chromosome 609083 609084 0.5 A
あとは0.5を必要な変異率に変えればリストに使うことができる。
small indelの導入
small indel変異をbamに導入する(test/)。
addindel.py -v test_indels.txt -f testregion_bwamem_realign.bam -r Homo_sapiens_chr22_assembly19.fasta -o human_indel.bam
SNVのリストは以下のようなフォーマットで準備する。indel変異では4列目以降も必ず必要である。
user$ cat test_data/test_indels.txt
22 33694303 33694304 0.5 INS ATGGC
22 33815273 33815274 0.25 INS CG
22 33920490 33920491 0.33 INS ATTTGCTTAGCTGAGGGCTTAGGCTTAGCATGCGTA
22 33944542 33944543 0.50 DEL
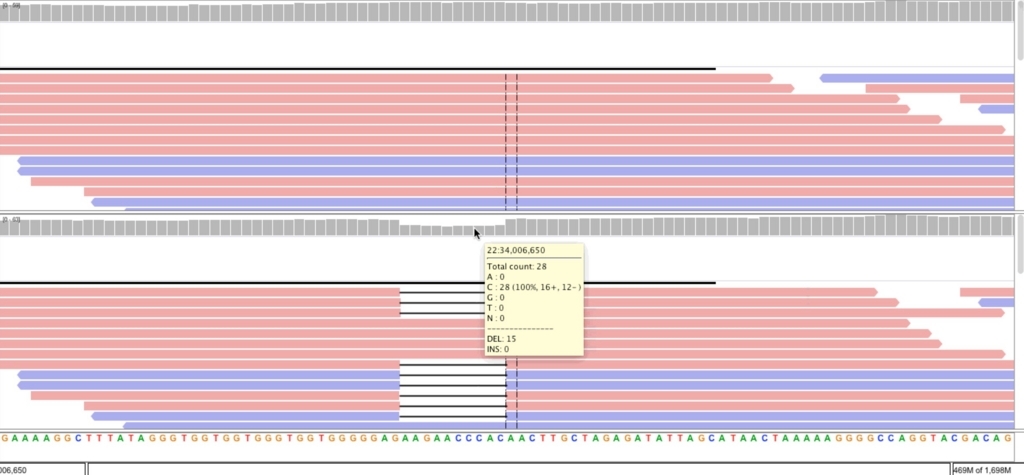
22 34006643 34006653 0.50 DEL
22 33958087 33958090 0.40 DEL
5列目はINSかDELのみ許されている。挿入は6列目に挿入される配列を記載する。
やはりVCFからリストを作ることができる。
#insertion
vcf2bed --insertions < input.vcf > ins.bed #insertionのみbedに変換
cut -f 1,2,3,4 ins.bed |sed -e 's/\./0\.5/g' > temp
cut -f 4,6 ins.bed |sed -e 's/\./INS/g' |paste temp - > insertion.txt
#deletion
vcf2bed --deletions < input.vcf > del.bed
cut -f 1,2,3,4 del.bed |sed -e 's/\./0\.5/g' > temp
cut -f 4,6 del.bed |sed -e 's/\./DEL/g' |paste temp - > deletion.txt
#マージしてソートする。
cat insertion.txt >> deletion.txt
sort -k 1,1 -k 2n deletion.txt > indel.txt
del
22 33944542 33944543 0.50 DEL
In

igvで見るには、ソートしてindexをつけておく必要がある(e.g., samtools sort i.bam > o.bam && samtools index o.bam)
SVの導入
構造変化(SV)を引き起こす大きな変異も導入できる(insertions, deletions, duplications, inversions, and compound rearrangements)。
addsv.py -p 18 -v test_sv2.txt -f testregion_bwamem_realign.bam -r Homo_sapiens_chr22_assembly19.fasta -o human_sv.bam
SVのリストは以下のようなフォーマットで準備する。
user$ cat test_data/test_sv.txt
22 34171055 34184700 INS ACTAACCTGCACAATGTGCACATGTACCCTAAAACTTAGAGTATAATAAAAAAAA 13 AA^TTTT
22 33607508 33607508 TRN 22 34314981 34314981
22 33871043 33884754 DEL 0.9
22 34071043 34084754 INV
22 34271043 34284754 DUP 3
細部は異なるが、small indelと同じような方法でVCFからリストは準備できる。ただしSV検出ツールによっては、VCFに変異後の配列を記載しないものがある。大きな挿入でも塩基を記載するようなVCF(例えばpindelならO.K)を変換元にしてください。
large deletion。
22 33871043 33884754 DEL 0.9

指定領域をアセンブルして、できた最長のcontigの9割(0.9)の領域が欠損となる。
duplication。
22 34271043 34284754 DUP 3
3回複製される。アセンブルしているので、duplicationが起きる部位は、指定領域より短くなる(上の図)。
逆位
22 34071043 34084754 INV
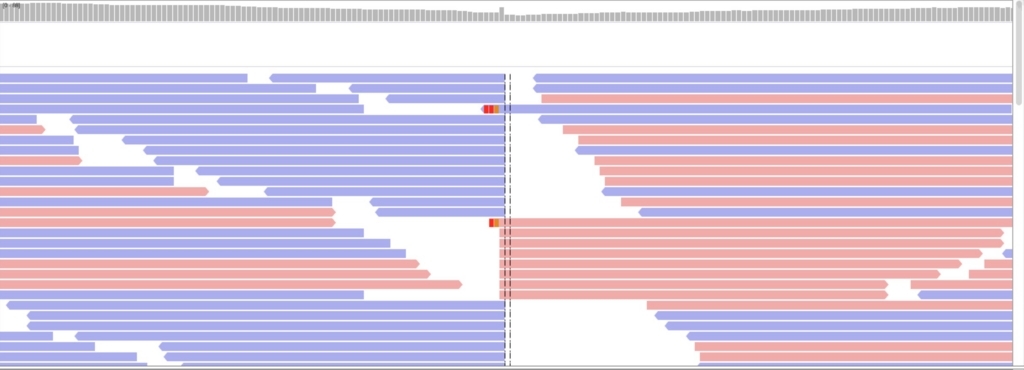
上流側境界。
ACTAACCTGCACAATGTGCACATGTACCCTAAAACTTAGAGTATAATAAAAAAAAを挿入。ただし13-bp末端を複製して挿入する。また、AA^TTTTが領域内になれば、そこをターゲットとする(AAとTTTTの間に導入)
22 34171055 34184700 INS ACTAACCTGCACAATGTGCACATGTACCCTAAAACTTAGAGTATAATAAAAAAAA 13 AA^TTTT
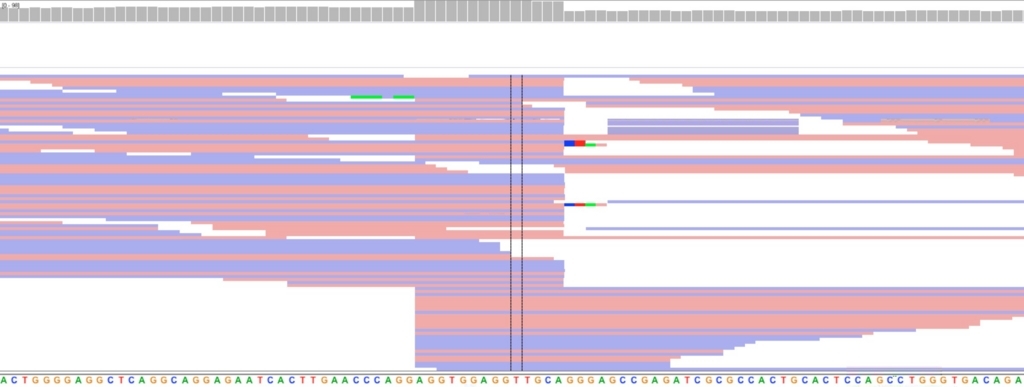
13-bp末端の複製があるので、13-bpズレて挿入部位が見えている。ターゲット配列を書かなければ、アセンブルされた最長のcontigの真ん中に導入される。
同時に複数の変異を導入、例えば欠損を起こしてそこに挿入を起こすような変異も導入できます。詳しくはマニュアルを参照してください。
https://hpc.nih.gov/apps/bamsurgeon/Manual.pdf
引用
Combining tumor genome simulation with crowdsourcing to benchmark somatic single-nucleotide-variant detection
Adam D Ewing, Kathleen E Houlahan, Yin Hu, Kyle Ellrott, Cristian Caloian, Takafumi N Yamaguchi, J Christopher Bare, Christine P'ng, Daryl Waggott, Veronica Y Sabelnykova, ICGC-TCGA DREAM Somatic Mutation Calling Challenge participants, Michael R Kellen, Thea C Norman, David Haussler, Stephen H Friend, Gustavo Stolovitzky, Adam A Margolin, Joshua M Stuart & Paul C Boutros
Nature Methods 12, 623–630 (2015) doi:10.1038/nmeth.3407