原核生物および真核生物において、上流のオープンリーディングフレーム(uORF、いわゆるリーダーペプチドをコードすることが多い)は、下流のメインORF(mORF)の翻訳と転写を制御することができる。しかし、新規機能性uORFのアノテーションは、通常100コドン未満という短いサイズであるため、困難である。転写および翻訳レベルのNGS技術は、ゲノムワイドな機能性uORFの同定に利用できるが、ゲノムが解読された大多数の生物種では、このデータはまだ利用できない。同時に、ゲノムアセンブリの量が飛躍的に増加していることから、機能性ORFの予測において進化的保存性を利用する機会が与えられている。
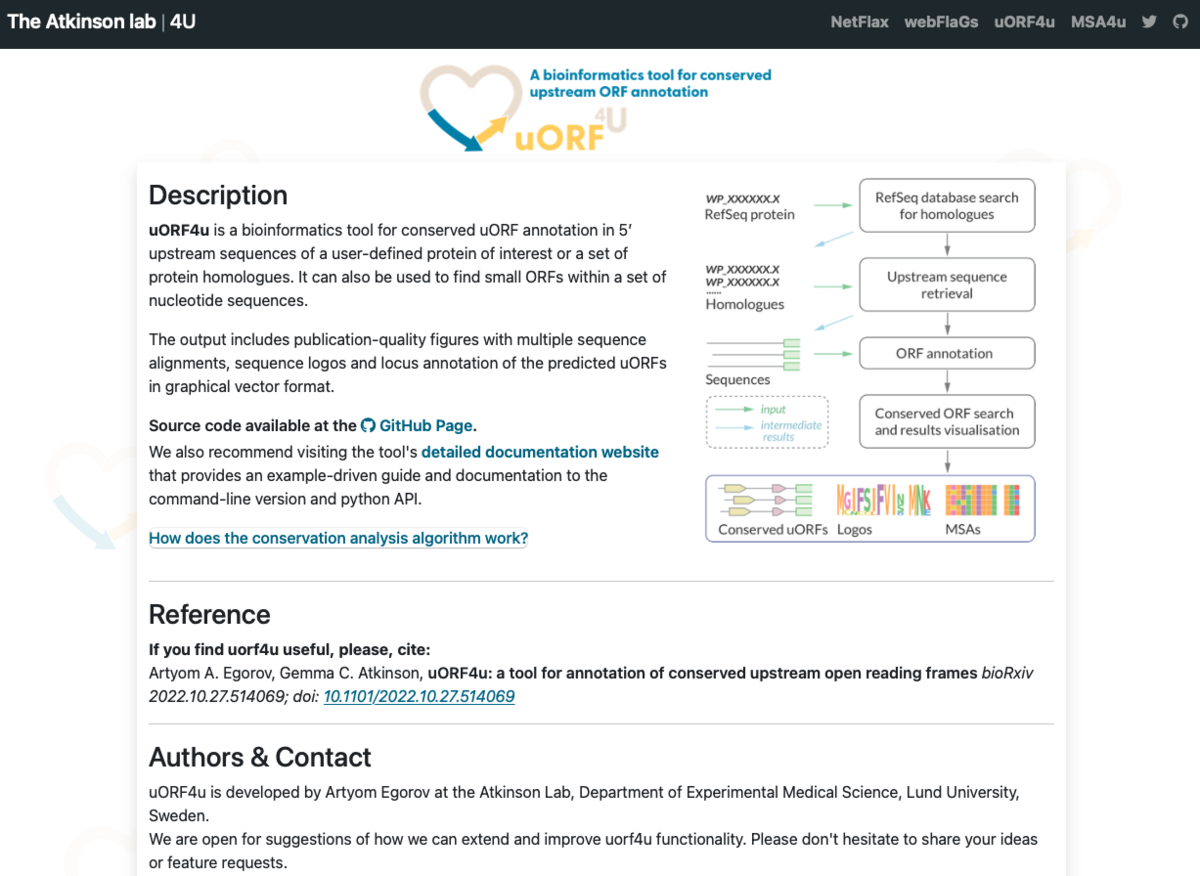
ここでは、ユーザが定義した目的のタンパク質やタンパク質のホモログのセットの5ʹ上流配列に保存されたuORFをアノテーションするためのツールを紹介する。また、塩基配列のセットから小さな保存ORFを見つけるのにも使用できる。出力には、予測された保存されているuORFの複数配列アラインメント、配列ロゴ、遺伝子座アノテーションを含む出版物品質の図がグラフィカルベクター形式で含まれている。uORF4uはPython3で書かれており、LinuxとMacOSで動作する。コマンドラインインターフェースはほとんどの実用的なユースケースをカバーし、提供されるPython APIはPythonプログラム内での使用と追加のカスタマイズを可能にする。ソースコードはGitHubページ(github.com/GCA-VH-lab/uorf4u)より入手できる。サンプルガイドを含む詳細なドキュメントは、ソフトウェアのホームページ: gca-vh-lab.github.io/uorf4u で入手できる。uORF4uのウェブバージョンはserver.atkinson-lab.com/uorf4uでできる。
Documentation
https://gca-vh-lab.github.io/uorf4u/
Happy that our uORF4u small ORF prediction tool is published! Awesome work from PhD student @art_egorov! uORF4u: a tool for annotation of conserved upstream open reading frames https://t.co/Zdp5FecSru
— Gemma C Atkinson (@gem__atkinson) 2023年5月16日
ここではウェブバージョンを紹介します。
https://server.atkinson-lab.com/uorf4uにアクセスする。
メニュー右から、微生物およびウイルスゲノムのタンパク質の機能的関連性の予測を行うwebFlaGs、MSAを可視化するMSA4Uなどにもアクセスできる。
入力はRefSeqアクセッションID、もしくはタンパク質アクセッションIDのリスト(最大200個)、または上流のヌクレオチド配列(最大1000 nt、200配列以内)となる。以下はexample入力時(ErmC homologues)。

他のパラメータを指定してサブミットする。

調整可能なパラメータについてはそれぞれの項目で説明されている。検索する上流配列の長さやデータベース、配列同一性の閾値、代替開始コドンであるか、ORF保存のカットオフ値などとなる。
ジョブが終わると結果をダウンロードできるページにジャンプする。

出力例
出力には、予測されたuORFの複数の配列アライメント、配列ロゴ、遺伝子座アノテーションを含む出版物品質の図がグラフィカルベクター形式で含まれている。
https://gca-vh-lab.github.io/uorf4u/ExampleDrivenGuide/cmd_guide/#output-folder-structureで説明されています。
その他
uorf4uの機能の拡張・改善に関する提案を受け付けているそうです。アイデアや機能要望であったり技術的な問題が発生した場合、メール(レポジトリ参照)もしくはGitHub Issuesを利用して連絡をして下さいと書かれています。
引用
uORF4u: a tool for annotation of conserved upstream open reading frames
Artyom A Egorov, Gemma C Atkinson
Bioinformatics, Volume 39, Issue 5, May 2023
参考
This tool really works. We have already used it to map out the uORFs regulating the expression of antibiotic resistance ARE ABCFs https://t.co/y9WEx8RvTx https://t.co/O4tFmGYRNJ
— Vasili Hauryliuk (@hauryliuk) 2023年5月16日