挿入および欠失(indel)は、フレームシフトの導入による遺伝子機能の根本的な変更を介してたくさんのヒト疾患に関与している。しかし、次世代シークエンシングデータからのこれらのindelを正確に検出する事は現在でもチャレンジングである。これは、DNAシーケンシングリードが短いため、中間サイズのindels(50 bp以上)に特に当てはまる。著者らは、BWAのソフトクリッピングされたフラグメント(マッピングされたリードの中で部分的にマッチしない領域)とマッピングされていないリードを使用して、中間サイズのindelを予測する新しい方法を開発した。我々(著者ら)は、同じサンプルからのwholeエクソンシークエンシングデータを使用して、本手法、GATK、PINDELおよびScanIndelの性能比較を報告する。これらの4つの方法において予測されたすべてのindelsのサンガーシーケンシングにより、偽陽性および偽陰性の数が決定された。Recallとprecisionの調和平均(F-measure)を用いて、各方法の性能を測定した。GATK 0.56、PINDEL 0.52、ScanIndel 0.46と比較して、我々(著者ら)の方法は1つのサンプルで0.84の最高F値を達成した。追加の試料でも同様の結果が得られ、本方法は他の中程度の指標を検出する方法より優れていることが示された。、我々(著者ら)は、この方法論がヒト疾患に関連した中間サイズのindels発見に貢献すると考えている。
東京医科歯科大学 プレスリリース
http://www.tmd.ac.jp/archive-tmdu/kouhou/20180411_1.pdf
インストール
mac10.12のdokcerコンテナ上でテストした。
依存
本体 Github
dokcerコンテナとして導入する。
git clone https://github.com/NCGG-MGC/IMSindel.git
cd IMSindel/
docker build -t imsindel .
> docker run --rm -v /Users/user/Documents/docker_share:/data imsindel
$ docker run --rm -v /Users/user/Documents/docker_share:/data imsindel
Usage: imsindel [options]
--bam /path/to/foo.bam
--chr chromosome
--outd /path/to/outoput-dir
--indelsize maximal indel-size
--reffa /path/to/ref.fa
--baseq [20]
--mapq [20]
--within [3]
--pair-within [5]
--alt-read-depth [5]
--support-reads [3]
--clip-length [5]
--support-clip-length [5]
--glsearch [glsearch36]
--glsearch-mat [data/mydna.mat]
--samtools [samtools]
--temp [/temp]
--thread [1]
--output-consensus-seq /path/to/output-dir
ラン
実行するには、リードをマッピングして得たbamとbam.bai、リファレンスのfastaが必要になる。マッパーとして、論文ではBWA-MEMが使われている。
#コンテナの/home/との共有ディレクトリ/Users/user/Documents/docker_share/をホスト側(mac os10.12側)に準備し、そこにbamとbamのindex、リファレンスfastaを配置した。
#共有ディレクトリを指定してランする。"--rm"で終了後コンテナ破棄。ファイルパスは仮装イメージ側の絶対パスを記載する。
docker run --rm -v /Users/user/Documents/docker_share:/data imsindel --bam /data/input.bam --chr chr10 --outd /data --indelsize 10000 --reffa /data/hg19.fa
結果は載せられないが、出力は以下のフォーマットに乗っ取っている。
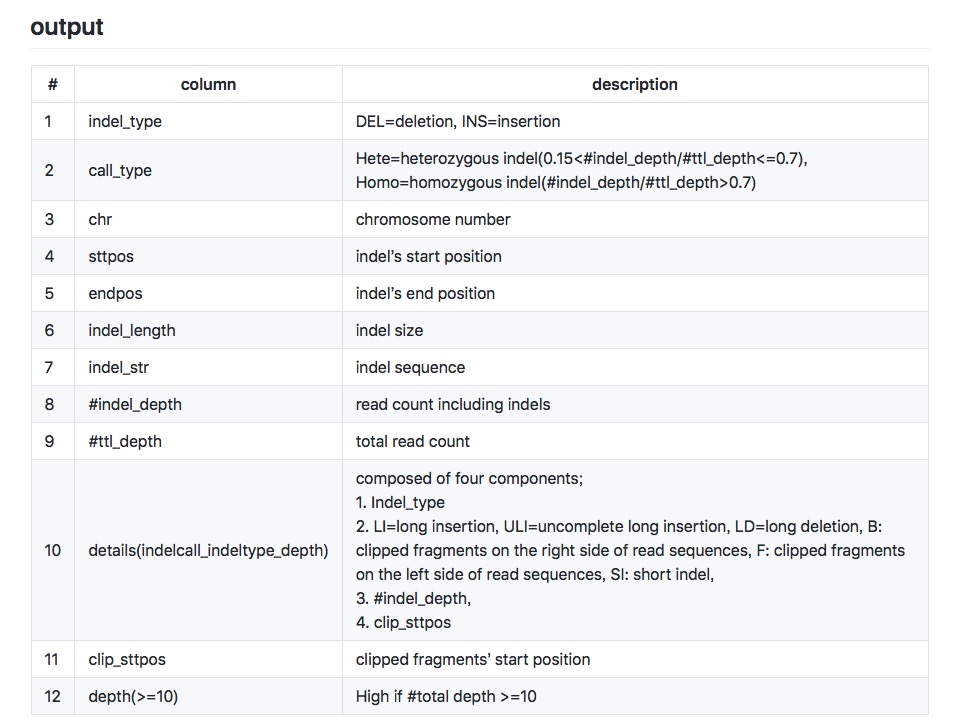
クロモソーム1つ分なら、早ければ数分で解析は終わります(WES 2GBデータ)。
引用
IMSindel: An accurate intermediate-size indel detection tool incorporating de novo assembly and gapped global-local alignment with split read analysis
Shigemizu D, Miya F, Akiyama S, Okuda S, Boroevich KA, Fujimoto A, Nakagawa H, Ozaki K, Niida S, Kanemura Y, Okamoto N, Saitoh S, Kato M, Yamasaki M, Matsunaga T, Mutai H, Kosaki K, Tsunoda T
Sci Rep. 2018 Apr 4;8(1):5608.