PlasMapper 3.0は、出版品質のプラスミドマップを生成、編集、注釈、対話的に可視化できるウェブサーバーである。プラスミドマップは、遺伝子クローニング実験に関する重要な情報を計画、設計、共有、公開するために使用される。PlasMapper 3.0は、PlasMapper 2.0の後継で、市販のプラスミドマップ作成/編集パッケージでしか見られない多くの機能を提供する。PlasMapper 3.0では、入力としてプラスミド配列を貼り付けたり、アップロードしたり、2000以上の注釈済みプラスミドからなる大規模データベース(PlasMapDB)から既存のプラスミドマップをアップロードすることができる。このデータベースは、プラスミド名、配列特徴、制限部位、好ましい宿主生物、配列長で検索できる。PlasMapper 3.0は、ほとんどのクローニングプラスミドに見られる共通のプロモーター、ターミネーター、制御配列、複製起点、選択可能マーカーなどの特徴を含む独自の特徴データベースを用いて、新しいプラスミドや見たこともないプラスミドのアノテーションもサポートしている。PlasMapper 3.0は、プラスミド領域の選択と表示、遺伝子の挿入、制限部位の変更、コドンの最適化を行うことができる複数の対話型配列エディター/ビューアーを備えている。PlasMapper 3.0では、グラフィックも大幅にアップグレードされた。インタラクティブなフルカラーのプラスミドビューア/エディタで、ズーム、回転、再カラー化、直線化、環状化、アノテーションの編集、プラスミド画像やラベルの修正などができ、プラスミドマップやテキスト表示の美観を向上させることができる。すべてのプラスミド画像とテキスト表示は、複数のフォーマットでダウンロードできる。PlasMapper 3.0は、https://plasmapper.ca、オンラインで利用できる。
PlasMapper 3.0は、高価なプラスミド編集/マッピングパッケージでしか見られない多くの機能を備えている。例えば、入力として任意のプラスミド配列を貼り付けたり、アップロードしたり、2000以上の注釈済みプラスミドからなるカスタムデータベース(PlasMapDB)から既存のプラスミドマップをアップロードすることができるようになっている。このデータベースは、プラスミド名、配列特徴、制限部位、好ましい宿主生物、配列長をブーリアン演算で組み合わせて検索することができる。PlasMapper 3.0は、独自のフィーチャーデータベース(FeatureDB)を用いて、新しいプラスミドや見たことのないプラスミドのアノテーションもサポートしている。フィーチャーデータベースは、プロモーター、ターミネーター、制御配列、複製起点、選択可能マーカー、その他ほとんどのクローニングプラスミドやベクターで見られるフィーチャーを網羅している。PlasMapper 3.0は、プラスミド領域の選択と表示、遺伝子の挿入、制限部位の変更、コドンの最適化を行うことができるインタラクティブな配列エディタを備えている。また、PlasMapper 3.0のグラフィカルな表示機能は、PlasMapper 2.0に比べ大幅にアップグレードされている。特に、PlasMapper 3.0では、インタラクティブでカラフルなプラスミドビューアー/エディターが搭載され、ズーム、回転、再カラー化、線状化、環状化、アノテーションのカスタマイズ、プラスミド画像やラベルの修正など、プラスミドマップの美観を向上させることが可能になった。また、PlasMapper 3.0では、カラフルなマップやテキスト表示を複数のフォーマットでダウンロードすることができるようになった。
help
https://plasmapper.caにアクセスする。
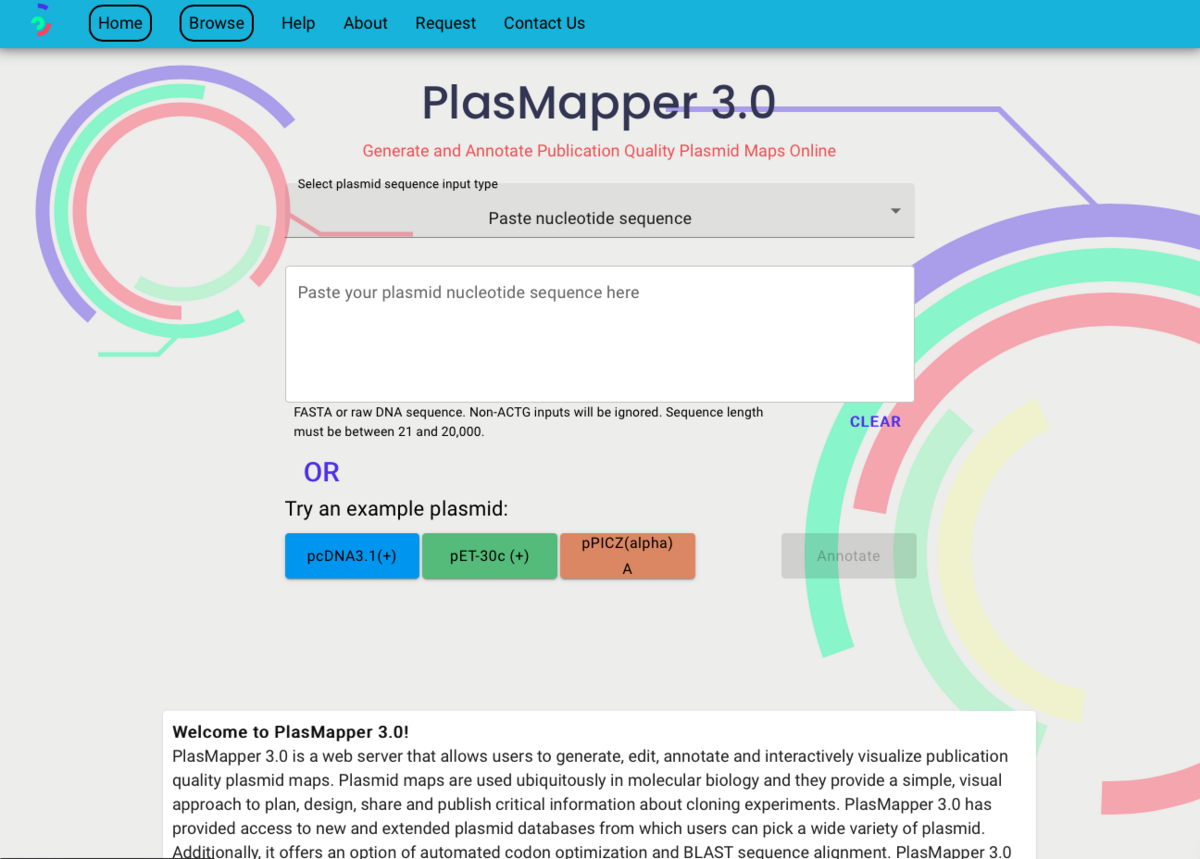
FASTA形式のプラスミド配列をペーストするかアップロードする。ここではexampleのpcDNA3.1を指定。
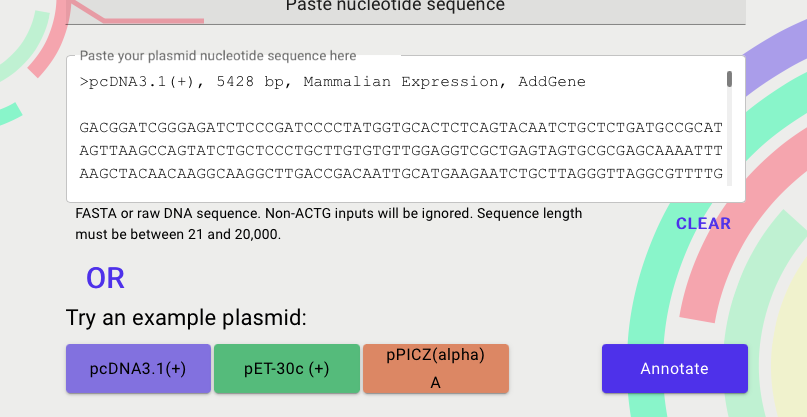
Annotateをクリックするとエディターの画面に移行する。
出力例
図は自由に拡大縮小、移動ができる。

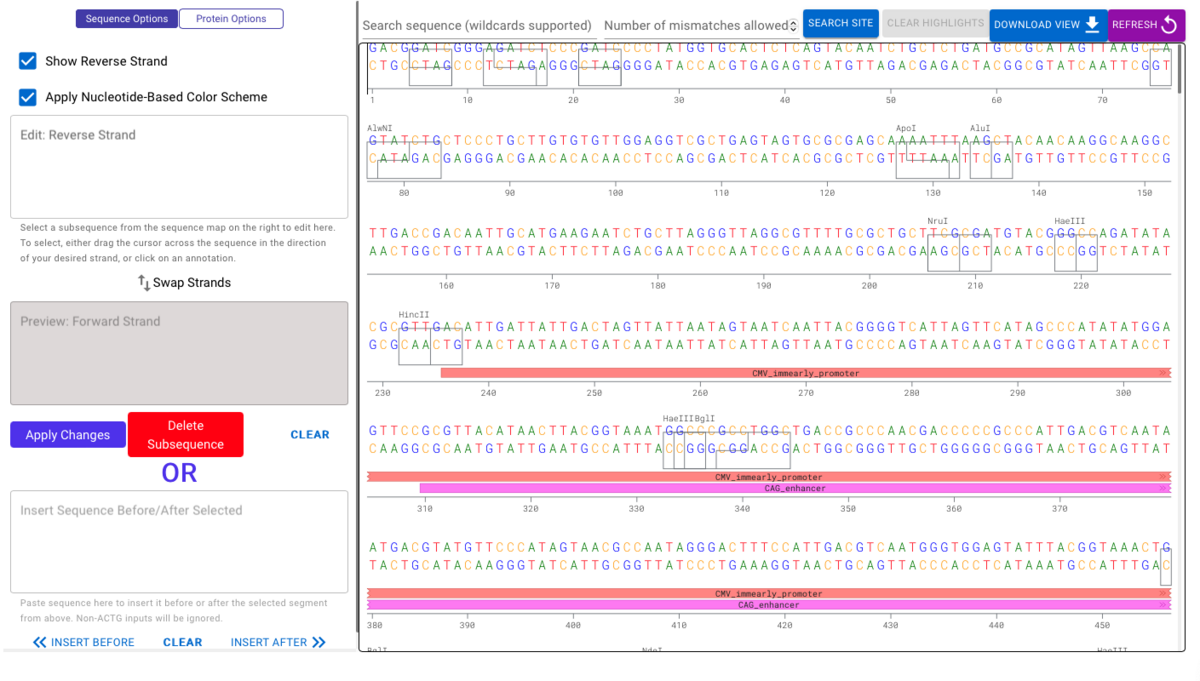
拡大を繰り返すことで最終的に塩基配列まで表示できる。

上の○/- ボタンをクリックすると線状表示に切り替わる。
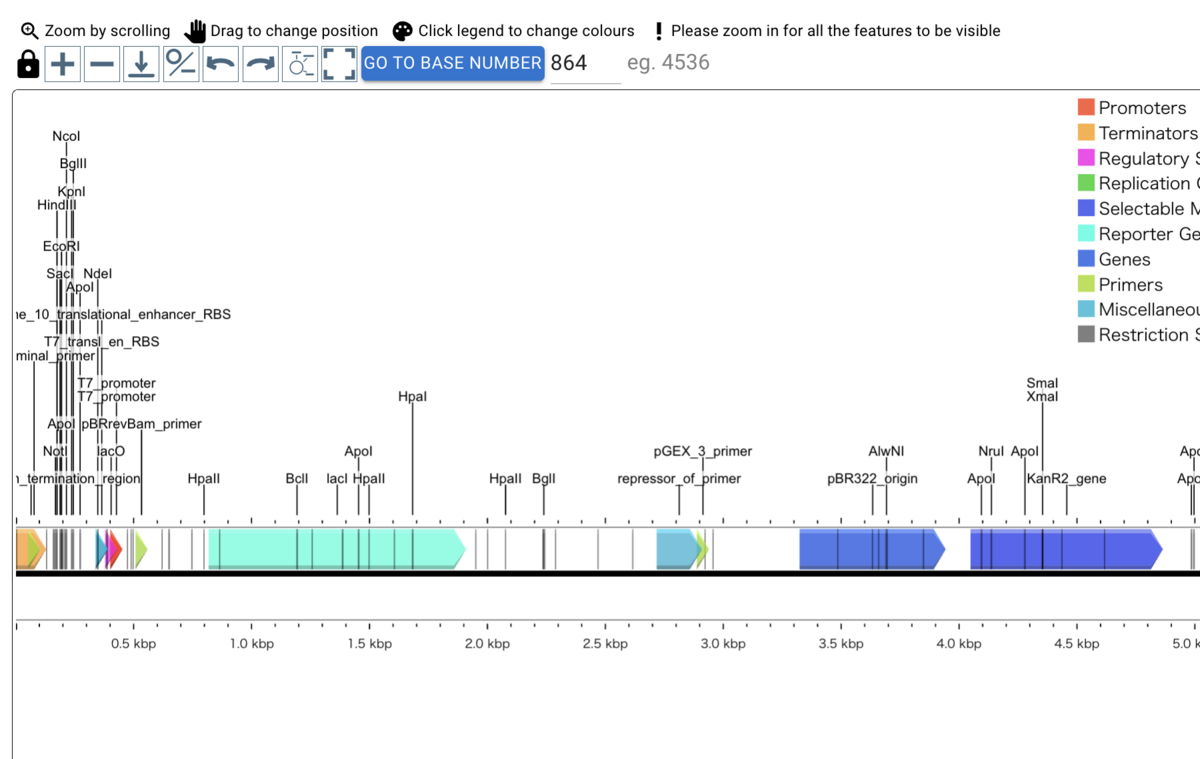
All featuresには全てのアノテーションが表示されている。図と連動しており、図のフィーチャーをクリックするとそのアノテーションが展開される。下の画像では図のlacIをクリックした。

図ではアノテーションの種類ごとに色分けして表示されている。種類は左側のメニューから手動で変更できる。lacIの種類を変えた。

制限酵素サイトを選択して表示
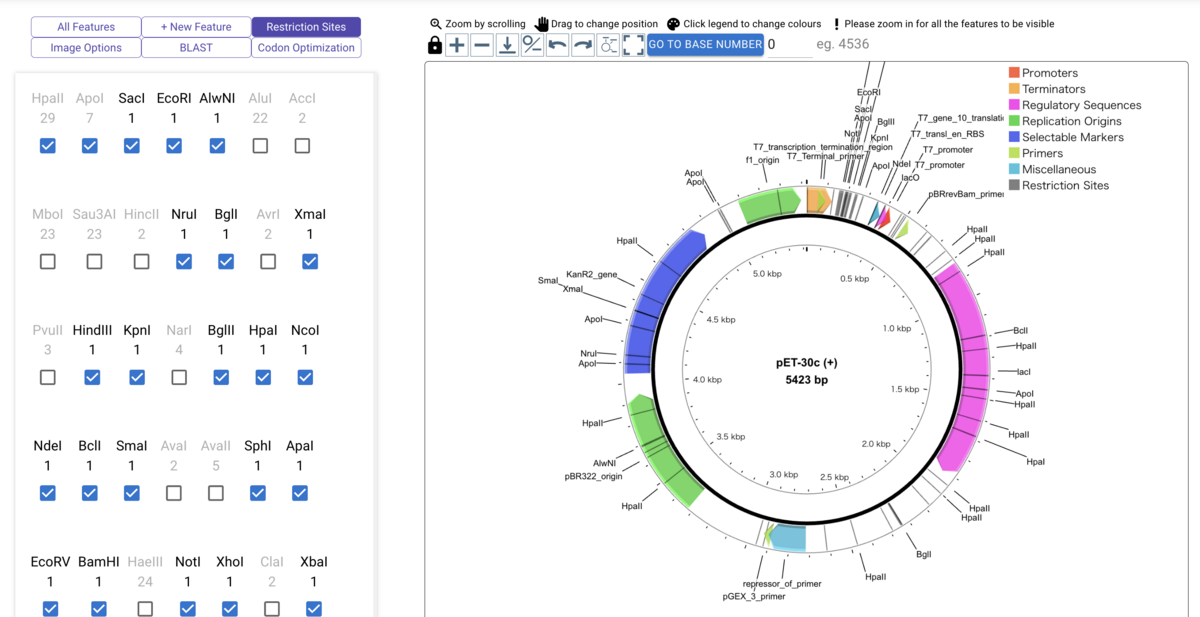
ユーザーが定義した新しいアノテーションフィーチャーを指定した座標に追加

追加する際はアノテーションの種類を選べる。種類ごとに色分けして表示される。

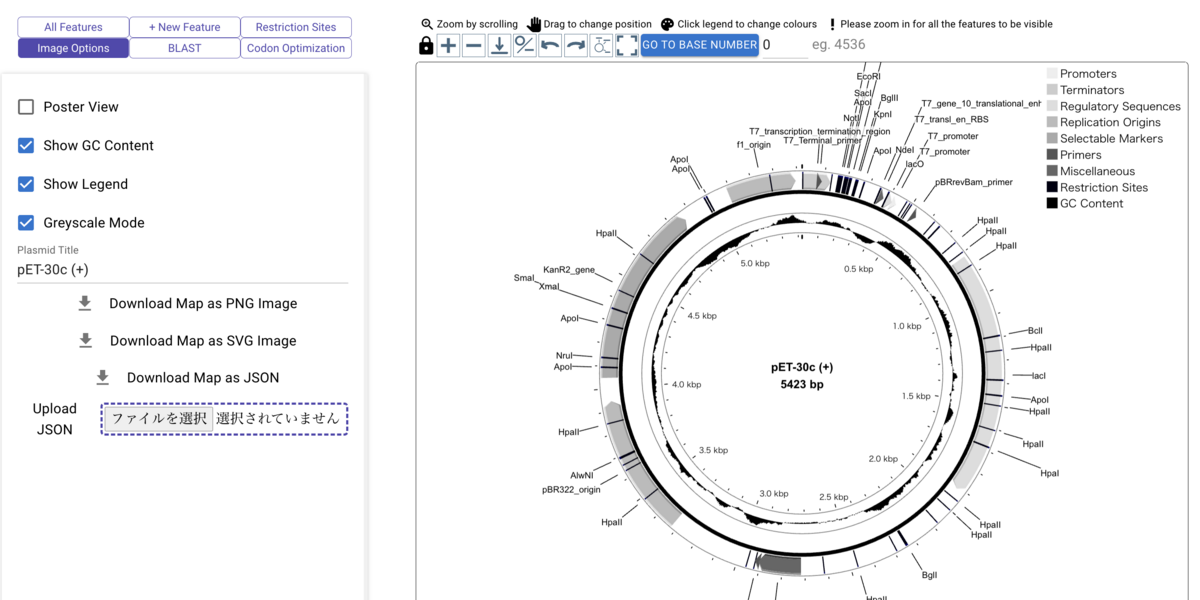
image optionからは視覚化される内容を指定できる。GC含量を表示してグレイスケールに変更(出版するジャーナルの関係で白黒にしたい時など)。
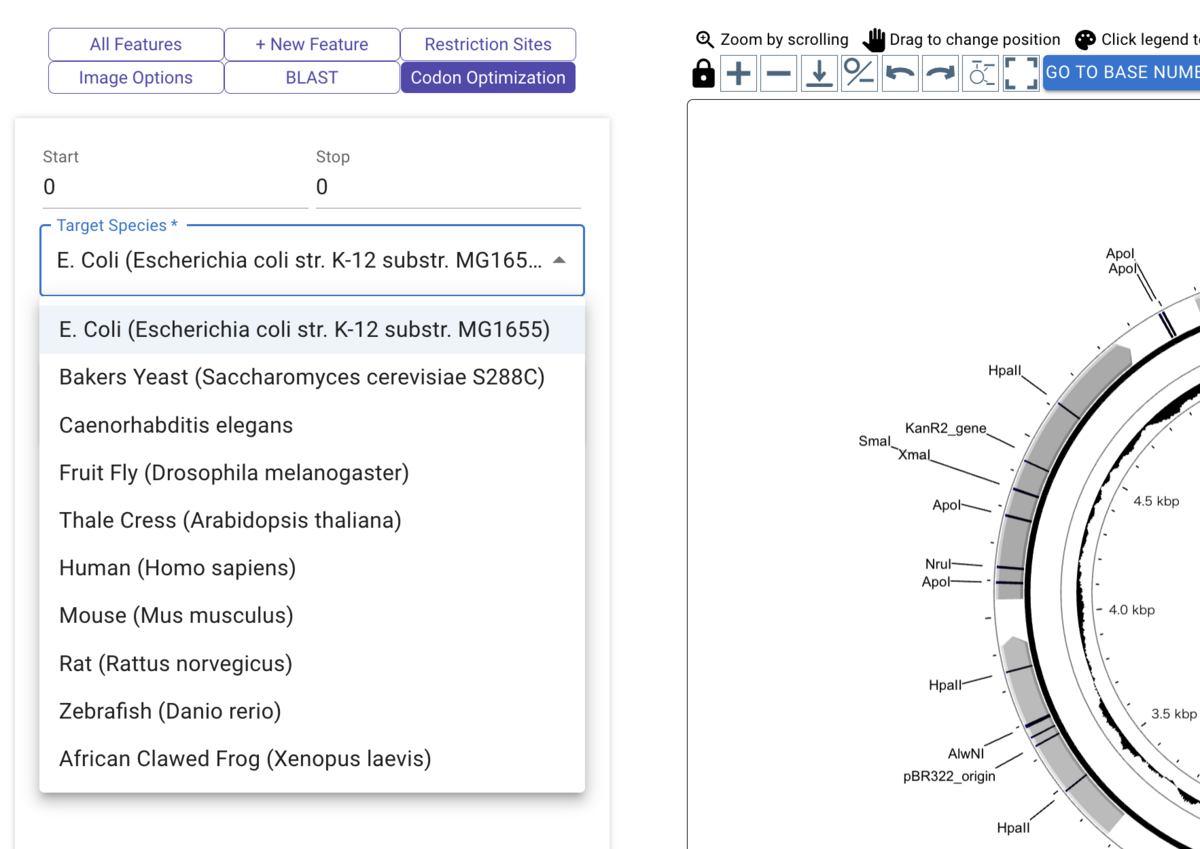
Codon optimizationからはコドンを指定できる。
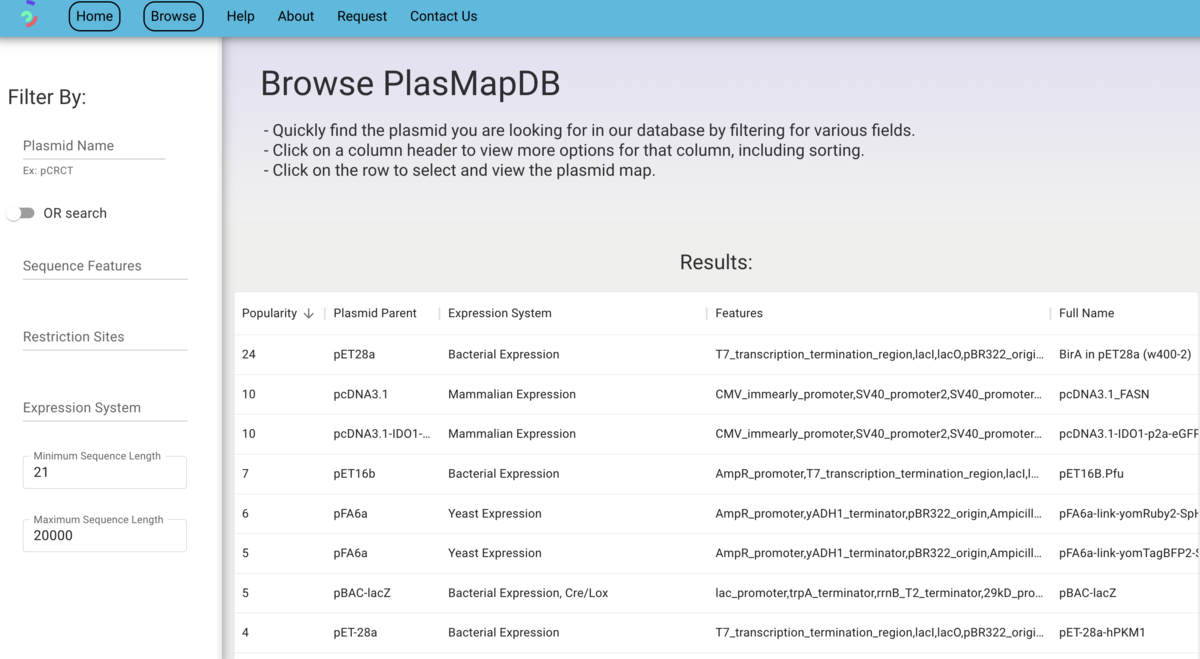
ブラウズタブからはPlasMapDBに登録されている2,000以上のプラスミド配列から、ユーザーが指定した条件で探索し、PlasMapperでアノテーションを確認し、さらに編集することが出来る。
紹介した以外にも、配列をNCBI BLAST ntデータベースに対しBLAST検索したり、出版品質の図をダウンロードする機能があります。アクセスしてみて下さい。
引用
PlasMapper 3.0—a web server for generating, editing, annotating and visualizing publication quality plasmid maps
David S Wishart, Leah Ren, Jacques Leong-Sit, Sukanta Saha, Jason R Grant, Paul Stothard, Upasana Singh, Abby Kropielnicki, Eponine Oler, Harrison Peters, Vasuk Gautam
Nucleic Acids Research, Published: 26 April 2023