2022/09/08 論文引用
ここ数十年、微生物叢、特にヒトの腸内細菌叢の研究と特性評価に大きな関心が寄せられ、常在微生物が人体の正常な解剖学的発達と生理的機能に極めて重要な役割を果たすことが明らかにされている。異なる環境を特徴づける複雑な細菌の動態をよりよく理解するためには、バクテリオファージの捕食や遺伝子導入も、細菌群集の密度、多様性、ネットワーク相互作用の制御に寄与する重要な要因であるため、考慮する必要がある。現在までに、様々なバクテリオファージ同定ツールが開発されており、ファージマイニング戦略、要求される入力ファイル、生成される結果が異なっている。しかし、バクテリオファージ解析に初めて取り組むユーザーは、様々な手法を理解し、生成される異なる結果を比較することに苦労するかもしれない。ここでは、複数のファージマイナーの使用を合理化し、重要な結果を要約して視覚化する包括的なレポートを生成し、さらに特定の結果をインタラクティブなフィルタリング可能な表で探索できるようにする包括的なreads-to-reportパイプラインであるMetaPhageを紹介する。このパイプラインは、広く採用されているワークフローマネージャであるNextflowで実装されており、ローカルサーバからクラウドまで、異なる前提でのタスクの最適な並列化を可能にし、コンテナ化パッケージを使用して再現可能な結果を保証している。MetaPhageは、スケーラビリティ、再現性、そして新しいマイニングや手法を容易に拡張できるように設計されており、この分野は常に拡大し続けている。MetaPhageはGPL-3.0ライセンスのもと、https://github.com/MattiaPandolfoVR/MetaPhage、自由に利用することができる。
#MetaPhage is a reads-to-report pipeline for metavirome analysis, which includes an exploration of the diversity and taxonomy of the prokaryotic #virome https://t.co/wWYQnMowtz
— andrea telatin (@telatin) April 18, 2022
When bioinformaticians and a phage scientist get together, magic happens and #MetaPhage was born.
— Evelien Adriaenssens (she/her) (@EvelienAdri) April 19, 2022
The #Nextflow pipeline includes all the steps to understand the #phages in the #microbiome #virome, reads to assembly, phage mining, network-based classification and much more! https://t.co/YoW5JR1Kgx
Documentation
https://mattiapandolfovr.github.io/MetaPhage/
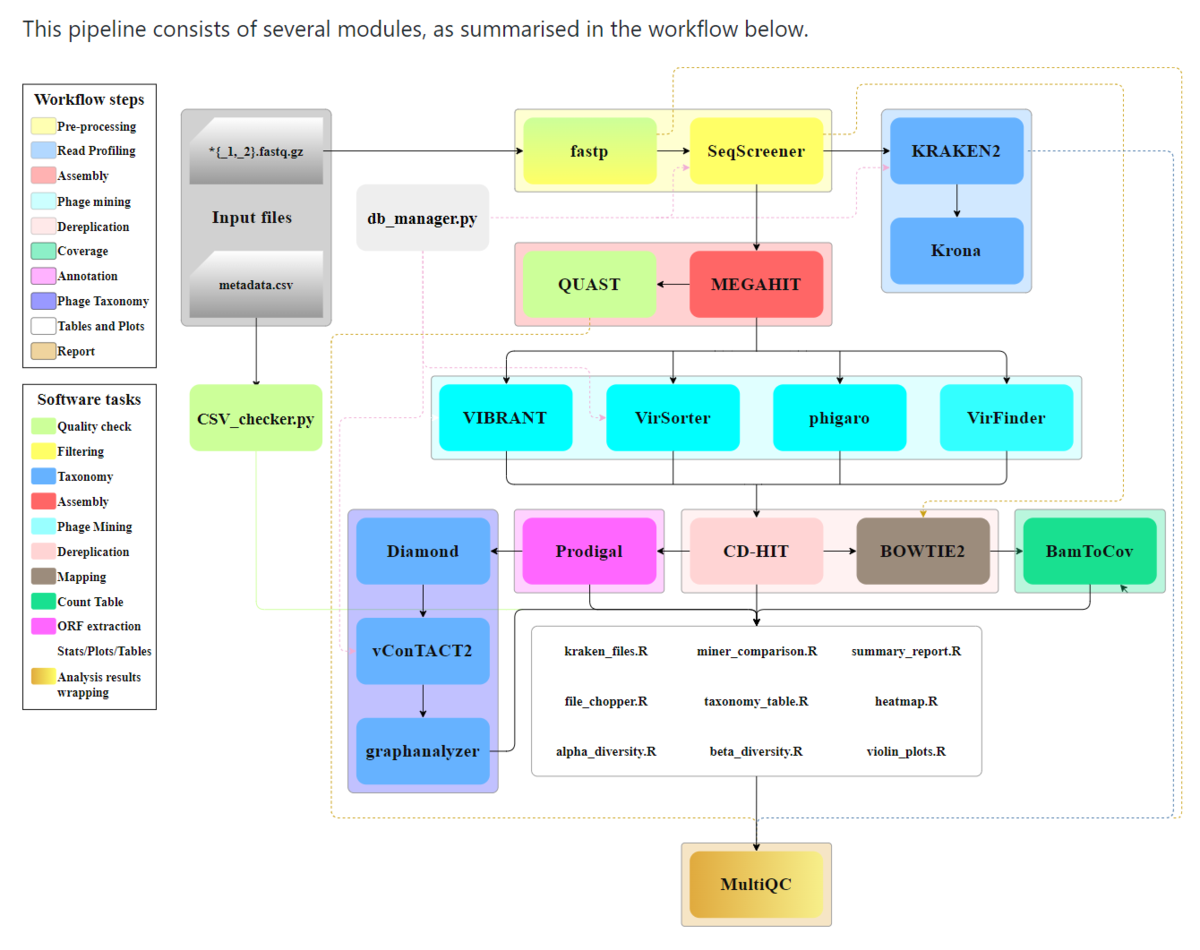
Pipelineオーバービュー。Githubより転載。
インストール
condaで環境を作って依存を導入した (ubuntu18)。
git clone https://github.com/MattiaPandolfoVR/MetaPhage.git
cd MetaPhage
#データベースのダウンロード。./dbが作成される。wgetが必要
python bin/python/db_manager.py -o ./db
#環境作成
mamba env create -n metaphage --file deps/env.yaml
conda activate metaphage
#docker (dockerhub) 明示的にpullする必要はない
docker pull andreatelatin/metaphage:1.0
テストラン
1、fastqファイル(20サンプル)のダウンロード(こちらのデータが使われている)。-t INTで同時ダウンロード数を指定している。
cd MetaPhage/
./bin/getExample.py --verbose -t 8
出力
demo/

2、configファイルの作成
python MetaPhage/bin/newProject.py -i demo \
-m demo/infant-metadata.csv \
-v Infant_delivery_type \
-s demo.conf
demo/infant-metadata.csv

demo.confが出力される。
3、nextflowのパイプラインのラン。2で作ったdemo.confを指定する。
nextflow run main.nf -c demo.conf


ランが終わるまで9時間ほどかかった(TR3990x, 256GBメモリ環境)。
出力
MetaPhage/

assembly/megahit/

mining/

prodigal/

cd-hit/

taxonomy/

taxonomy/vcontact2/

report/

report/MetaPhage_report.html








引用
MetaPhage: an automated pipeline for analyzing, annotating, and classifying bacteriophages in metagenomics sequencing data
Mattia Pandolfo, Andrea Telatin, Gioele Lazzari, Evelien M. Adriaenssens, ProfileNicola Vitulo
bioRxiv, Posted April 17, 2022
2022/09
MetaPhage: an Automated Pipeline for Analyzing, Annotating, and Classifying Bacteriophages in Metagenomics Sequencing Data
DOI: https://doi.org/10.1128/msystems.00741-22