過去10年間で、大量のRNAシーケンス(RNA-seq)データが公開リポジトリに寄託され、さらに前例のない速度で生産されている。しかし、汎用性が高く、RNA-seqデータセットの包括的な解析を合理的に行うことができる、ポイント&クリックのインターフェースを持つオープンソースツールはほとんど存在しない。これらの膨大な公的リソースを最大限に活用し、生物学者によるRNA-seqデータの解析を促進するために、OneStopRNAseqというRNA-seqデータのワンストップ解析のためのウェブアプリケーションを開発した。OneStopRNAseqはユーザーフレンドリーなインターフェースを持ち、包括的なデータ品質管理、遺伝子発現のdifferential gene expression (DGE)、differential exon usage (DEU)、differential alternative splicing (DAS) 、differential transposable element expression (DTE) 、 allele-specific gene expression (ASE) quantification、GO termおよびKEGGパスウェイの過剰発現解析、MSigDB-based gene-set enrichment analysis (GSEA) など、一般的なRNA-seqデータ解析のワークフローを提供する。ユーザーは、希望する解析とゲノムを選択し、Gene Expression Omnibus (GEO) アクセッション番号または配列ファイル、アラインメントファイル、遺伝子発現行列、ランクファイルへのリンクと対応するメタデータをDropboxに提供するだけで、簡単に利用することができる。このパイプラインは、プライベートおよびパブリックなRNA-seqデータの包括的かつ効率的な解析を促進する。
OneStopRNAseqは、生のリード品質をチェックするために広く使用されているFastQC を採用し、統合レポートを生成するためにMultiQCを採用している。また、ワークフローはリードアライメントにSTARを採用している。現在、ヒト、マウス、酵母、ショウジョウバエ、ゼブラフィッシュ、線虫ゲノムのRNA-seqデータ解析がサポートされているが、ユーザーの要望に応じて他のゲノムを容易に追加することができる。アライメント後のRNA-seq品質管理はQoRTを用いて行われ、RNA-seqデータの品質メトリクスを最も包括的に可視化して出力する。ワークフローは、BAMファイルから遺伝子レベルのカウントテーブルを取得するfeatureCounts、DASを検出するrMATS、DEU解析にDEXseq、TE発現定量にSalmonTE、DGEおよびDTE解析にDESeq2、ASE(対立遺伝子特異発現定量)にGATK ASEReadCounter、遺伝子セットエンリッチメント解析にGSEAが使用されている。OneStopRNAseqは、https://mccb.umassmed.edu/OneStopRNAseq、アカデミックユーザーが自由にアクセスできる。Snakemake ワークフローは、https://github.com/radio1988/OneStopRNAseq からダウンロードできる。
Users guide
https://mccb.umassmed.edu/OneStopRNAseq/documents/users_guide.pdf
Description of outputs
https://mccb.umassmed.edu/OneStopRNAseq/documents/description_of_output_files.pdf
OneStopRNAseqにアクセスする。
初回はアカウントを作成する必要がある。1分で作れる。
黒枠のFASTQなどをクリックするとジョブを開始できる。FASTQからは全解析が行えるが、Count tableから実行できるのはDGE analysisなど一部の解析のみとなる。

New studyの画面
ファイルをアップロードするか、GEOのIDを指定する。FASTQファイルから解析を開始した場合、すべての解析が行われる。ただし、ASEの定量には、遺伝子型情報を含むVCFファイルを追加で提供する必要がある。DGE解析やGSEAを行うだけなら、遺伝子発現数テーブルから始める。GSEAを実行するには、ランク付けされた遺伝子リストをアップロードするだけで良い。ここではGEOを選択した。

(論文より プライベートRNA-seqデータはローカルに保存されるか、より一般的にはDropbox、OneDrive、Google Drive、Box、pCloudなどの商用クラウドストレージ空間に保存される。その中でも、Dropboxはデータ共有のためのクラウドストレージスペースとして人気が出てきている(https://www.pcmag.com/picks/the-best-cloud-storage-and-file-sharing-services)。OneStopRNAseqを使用してDropbox内のRNA-seqデータを解析するには、ユーザーはウェブインターフェースを通じてデータとサンプル情報(メタデータ)の共有Dropboxリンクを提供するか、必要なメタデータを含むExcelスプレッドシートをアップロードして、比較する条件を指定する。)
次の画面では、研究の名前、生物、解析内容、GEOのID等を指定していく。ここでは一番下のexampleボタンをクリックして以下の設定にした。


使用されているゲノムとアノテーションのバージョンはこちらを参照。
次の画面ではサンプルのグループやラベルを確認する。グループは最低2つないとランできない。
(論文より 既存のRNA-seq解析パイプラインの大部分とは異なり、OneStopRNAseqは2つ以上のグループを持つ複雑なデザインに対応できる。例えば、ランダム化完全ブロックデザインでは、GROUP_LABELに研究対象因子を入力し、BATCH_LABELで研究対象外の因子をブロックすることができる。因子のデザインによって、ユーザーは異なる要因のラベルを連結したGROUP_LABELを入力することができる。例えば、前述の事例で2つの処理(CKとUSP7797)と2つの時点(24時間と48時間)からなる2×2の因子デザインでは、ユーザーはGROUP_LABELをCK_24h、CK_48h、USP7797_24h、USP7797_48hと入力することが可能になっている。治療と時間の主効果、一対の比較、異なる時点での治療の差分効果など、任意の比較や対照を指定することができる。DGEおよびDAS解析のための様々な種類の対照を指定する方法に関する詳細な説明は、補足ファイルとしてユーザーガイドに含まれており、https://mccb.umassmed.edu/OneStopRNAseq のヘルプメニューから入手できる。)
表を修正したい時はセルをクリックする。特定のサンプルを除外するには右端のDELETEボタンをクリックする。
submitをクリック。
次に、どの条件をAとBとして比較するか聞かれる(log2(GROUP A / GROUP B)として計算される)。また、対応する解析パラメータを指定する。

submitをクリック。
最終の確認画面。OKならsubmitをクリック。

メニュー右上のView historyからは、自分のジョブの進捗を確認できる。下の画像だとQCの計算が進行中と分かる。

登録したメールに、計算が終わったプロセスの通知が来る。そのリンクから結果にアクセスできる。
fastqcの結果

Differential Gene Expression Analysis (DGE) Results from DESeq2/

R Markdownのファイルも含まれている。
DESeq2.html
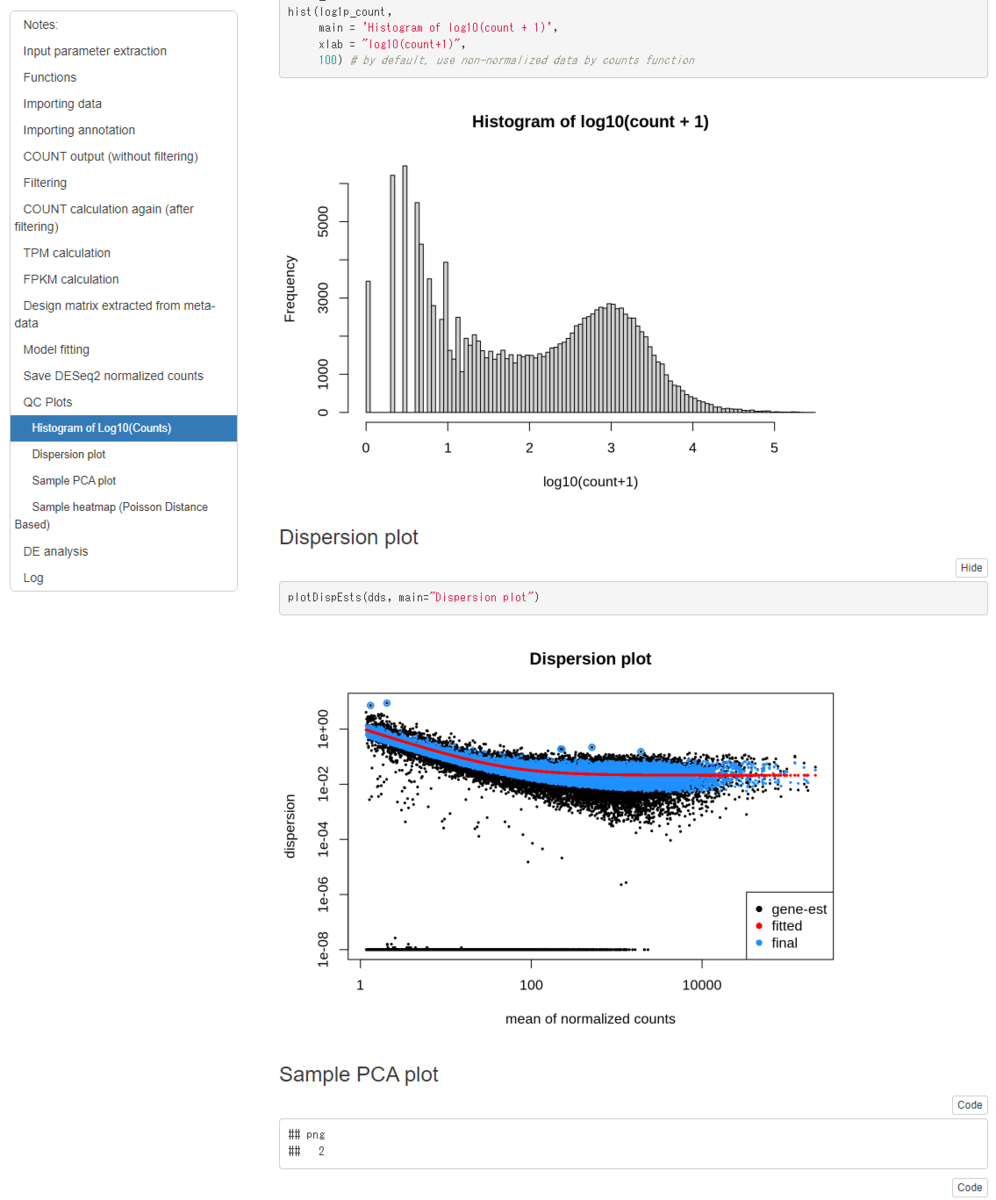
それぞれのコマンドも確認、再実行できるようになっている。
アクセスしてみて下さい。
引用
OneStopRNAseq: A Web Application for Comprehensive and Efficient Analyses of RNA-Seq Data
Rui Li, Kai Hu, Haibo Liu, Michael R Green, Lihua Julie Zhu
Genes (Basel). 2020 Oct 2;11(10):1165
関連
