シグナルペプチド(SP)は、すべての生物において、タンパク質の分泌や移動を制御する短いアミノ酸配列である。SPは配列データから予測することができるが、既存のアルゴリズムでは既知のSPの種類を全て検出することはできない。本稿では、5種類のSPをすべて検出し、メタゲノムデータに適用可能な機械学習モデル、SignalP 6.0を紹介する。
SPは、真核生物では分泌経路(Sec)、原核生物では細胞膜(内膜)を通過するタンパク質の標的となるN末端の短いアミノ酸配列である。実験的にSPを網羅的に同定することは困難であるため、計算機によるSPの予測は細胞生物学の研究にとって重要な意味を持つ。SP予測ツールは、一般的な分泌経路やtwin-arginine translocation (Tat) 経路をたどるタンパク質を同定し、シグナルペプチダーゼ (SPase) がSPを切断する配列上の位置を予測することができる。SignalP 5.0は、SPase I (Sec/SPI) またはSPase II (Sec/SPII, 原核生物のリポタンパク質) によって切断されるSec基質、およびSPase I (Tat/SPI) によって切断されるTat基質を予測することができる.。しかし、アノテーションデータがないため、SignalP 5.0はSPase IIによって切断されたTat基質やSPase III (prepilin peptidase, 時にはSPase IV2とも呼ばれる) によって処理されたSec基質を検出することができない。このようなSec/SPIII SPは、原核生物において接着、運動、DNAの取り込みに重要な役割を果たすIV型ピリン様タンパク質のトランスロケーションを制御していることが分かっている。さらに、SignalP 5.0は、SPの生物学的機能の根幹をなす部分領域(N末端のn領域、疎水性のh領域、C末端のc領域)を定義できないため、SP構造に関して不可知論的であった。
SignalP 6.0は、全生物領域にわたる数百万の未注釈タンパク質配列の情報を用いたタンパク質言語モデル(LM)6、7、8、9をベースにしている。LMは、タンパク質の生物学的特性や構造を理解するための意味的な表現を作成する。SignalP 6.0は、これらのタンパク質表現を用いて、従来のバージョンでは検出できなかった新たなタイプのSPを予測し、モデル作成に使用したタンパク質と遠縁のタンパク質や起源不明のメタゲノムデータをより適切に外挿することができる。さらに、SPのサブリージョンを特定することも可能である。
webサービス(オンライン)
https://services.healthtech.dtu.dk/service.php?SignalP-6.0
mirror;https://dtu.biolib.com/SignalP-6
(HPの説明より)SignalP 6.0サーバーは、古細菌、グラム陽性菌、グラム陰性菌、真核生物のタンパク質におけるシグナルペプチドの存在とその切断部位の位置を予測します。BacteriaとArchaeaでは、SignalP 6.0は以下の5種類のシグナルペプチドを識別することができます。
- Sec/SPI: Sec/SPI:Secトランスロコンによって輸送され、シグナルペプチダーゼI (Lep) によって切断される「標準的」な分泌シグナルペプチド
- Sec/SPII:Secトランスロコーンで輸送され、シグナルペプチダーゼII(Lsp)で切断されるリポ蛋白質シグナルペプタイド
- Tat/SPI。Tatトランスロコンにより輸送され、シグナルペプチダーゼI(Lep)により切断されたTatシグナルペプチド
- Tat/SPII: Tatリポ蛋白のシグナルペプチドがTatトランスロコンにより輸送され、シグナルペプチダーゼII(Lsp)により切断されたもの。
- Sec/SPIII:ピリンおよびピリン様シグナルペプチドがSecトランスロコンを介して輸送され、シグナルペプチダーゼIII(PilD/PibD)によって切断されたもの。
さらに、SignalP 6.0はシグナルペプチドの領域を予測します。シグナルペプチドの種類に応じて、n-、h-、c-領域の位置や、その他の特徴的な領域を予測することができます。
真核生物のタンパク質 シグナルペプチドの有無がタンパク質の局在の全てではないことをお忘れなく! 真核生物のタンパク質の局在についてもっと知りたい場合は、タンパク質細胞内局在予測ツールDeepLocをお試しください。また、シグナルペプチドを持つタンパク質がGPIアンカーを持ち、細胞膜の外側に付着しているかどうかをNetGPIという予測ツールでチェックすることもできます。
タンパク質配列を貼り付けるかアップロードする(10アミノ酸以上)。タンパク質の最大数は5000だが、ユーザーのエントリが100件を超えるとタイムアウトすることがある。
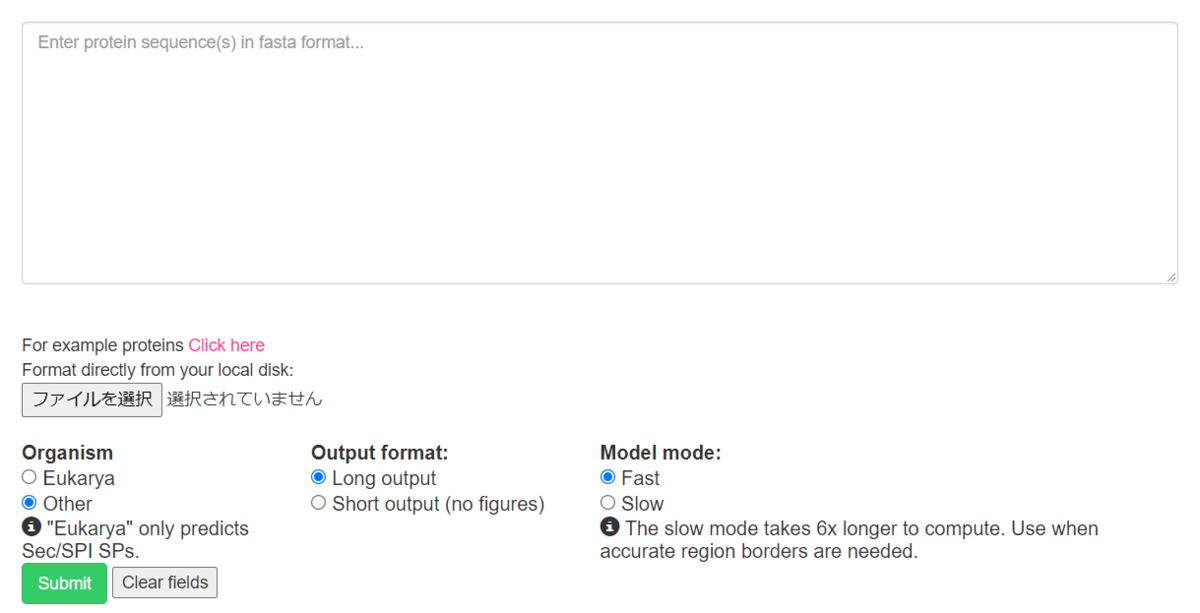
パラメータを確認してsubmitする。
exaamle outputファイルの出力


ローカルマシンへのインストール
fastモデルとslowモデル(HPによると、fastより精度が高いが6倍ほど遅い)がダウンロードできる。
https://services.healthtech.dtu.dk/software.php
感想
論文図2にはv5と比較した時の結果が提示されています。切断部位の予測精度と感度の増加と、配列同一性が低い時の感度と精度(マシューズ相関係数で評価)の増加が著しいようです。
引用
SignalP 6.0 predicts all five types of signal peptides using protein language models
Felix Teufel, José Juan Almagro Armenteros, Alexander Rosenberg Johansen, Magnús Halldór Gíslason, Silas Irby Pihl, Konstantinos D. Tsirigos, Ole Winther, Søren Brunak, Gunnar von Heijne & Henrik Nielsen
nature biotechnology, Brief Communication, Published: 03 January 2022
関連