2020 8/9 誤字修正
2020 10/5 説明追加
2021 8/9 追記
ggplot2' パッケージは R でのエレガントなデータ可視化のための優れた柔軟性を持っているが、デフォルトで生成されるプロットは、出版前にいくつかのformatingを必要とする。さらに、'ggplot'をカスタマイズするための構文は不透明であり、高度なRプログラミングスキルを持たない研究者にとっては難易度が高くなる。ggpubr' は、'ggplot2'ベースのpublication readyプロットを作成し、カスタマイズするための使いやすい関数を提供する。
インストール
Rstudio最新版を使ってテストした。
install.packages("ggpubr")
#最新版
if(!require(devtools)) install.packages("devtools")
devtools::install_github("kassambara/ggpubr")
実行方法
1、ライブラリのロード
library("ggpubr")
2、データの読み込み
次のような整然構造のフォーマットを読み込む(excelライクな形式は*1参照)。
| genome | GC | |
| 1 | genome1 | 77 |
| 2 | genome1 | 64 |
| 3 | genome1 | 70 |
| 4 | genome1 | 55 |
| 5 | genome1 | 77 |
| 6 | genome1 | 76 |
| 7 | genome1 | 76 |
| 8 | genome1 | 79 |
| 9 | genome1 | 71 |
| 10 | genome2 | 60 |
| 11 | genome2 | 61 |
| 12 | genome2 | 59 |
| 13 | genome2 | 59 |
| 14 | genome2 | 63 |
| 15 | genome2 | 64 |
| 16 | genome2 | 61 |
| 17 | genome2 | 57 |
| 18 | genome3 | 91 |
| 19 | genome3 | 90 |
| 20 | genome3 | 90 |
| 21 | genome3 | 97 |
| 22 | genome3 | 85 |
| 23 | genome3 | 91 |
| 24 | genome3 | 83 |
| 25 | genome3 | 88 |
excelライクなフォーマットと違って欠損値(欠損セル)問題が起きない。
コピペ読み込み。上の表をコピーして下のコマンドを実行する。
#mac
input <- read.table(pipe("pbpaste"), header = TRUE)
#windows
input <- read.table("clipboard"), header = TRUE)
またはファイルinput.tsvに保存し、データフレームに読み込む。
#カレント
input <- read.table("input.tsv", header=T, sep="\t")
#ファイルがカレントにないならフルパス指定か. セパレータがコンマならsep=","
input <- read.table("/home/kazu/data/input.csv", header=T, sep=",")
#tiny dataではない行列ファイルならtiny dataに変換する(上のデータでは不要)
reformat <- melt(data = input)
3、視覚化
violin plot - Violin plot manual
ggviolin(input, x = "genome", y = "GC", fill = "genome",
palette = c("#00AFBB", "#E7B800", "#FC4E07"),
add = "boxplot")

色を青系で統一する。
ggviolin(input, x = "genome", y = "GC", fill = "genome",
palette = "Blues", add = "boxplot")

palette = "rainbow"にすれば虹色で描画される。Bluesやrainbowのように定義済みのカラーパレットについてはこちらが詳しい(リンク)。
”DARK2”から3色指定。
library(RColorBrewer)
#色を確認、”DARK2”から3色指定。
display.brewer.pal(3, "Dark2")
#plot
ggviolin(input, x = "genome", y = "GC", fill = "genome",
palette = brewer.pal(3, "DARK2"),
add = "boxplot")

box plot - box plot manual
input <- read.table(pipe("pbpaste"), header = TRUE)
ggboxplot(input, x = "genome", y = "GC", color = "genome", palette =c("#00AFBB", "#E7B800", "#FC4E07"), add = "jitter", shape = "genome")

p-valueを追加
p <- ggboxplot(input, x = "genome", y = "GC", color = "genome", palette =c("#00AFBB", "#E7B800", "#FC4E07"), add = "jitter", shape = "genome")
#一度描画
p
#比較対象を定義
my_comparisons <- list( c("genome1", "genome2"), c("genome2", "genome3"), c("genome1", "genome3") )
#Piarwise比較してp-valueを追加
p + stat_compare_means(comparisons = my_comparisons)
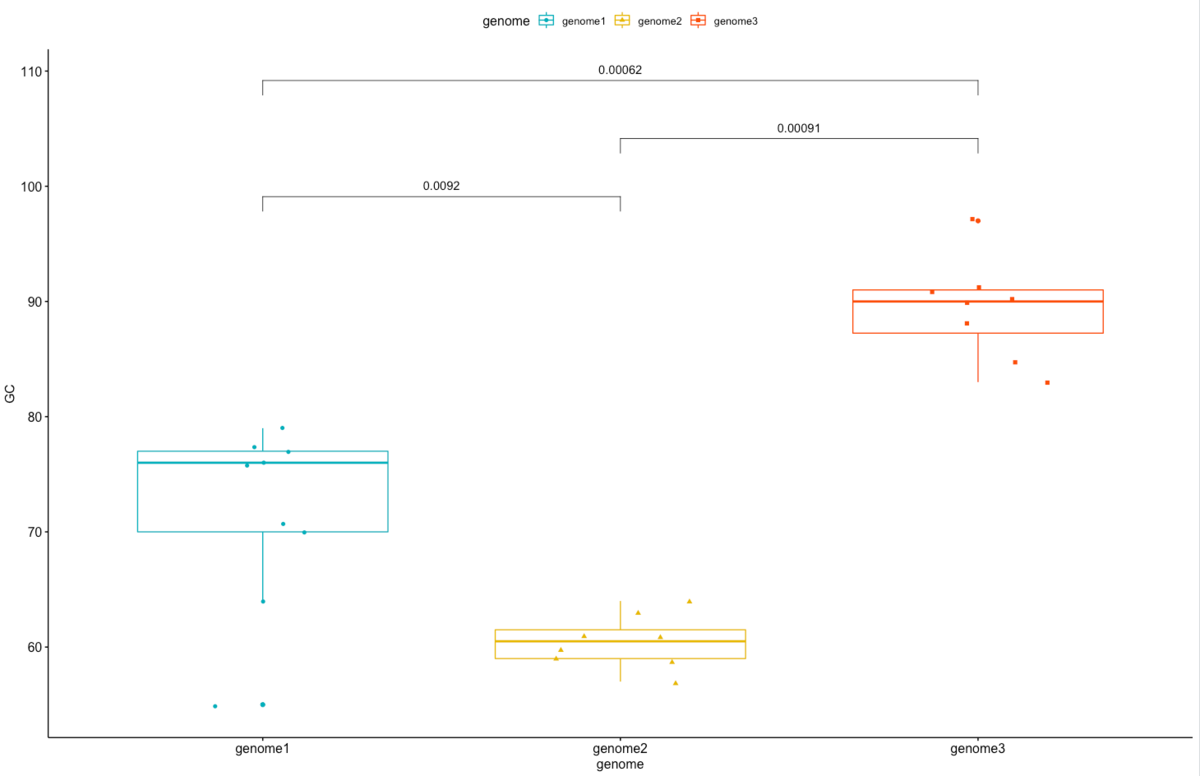
#比較対象を定義
my_comparisons <- list( c("genome1", "genome2"), c("genome2", "genome3"), c("genome1", "genome3") )
#global p-valueを求めた結果も追加.label.y = 120でy=120の位置に追加
p + stat_compare_means(comparisons = my_comparisons) + stat_compare_means(label.y = 120)
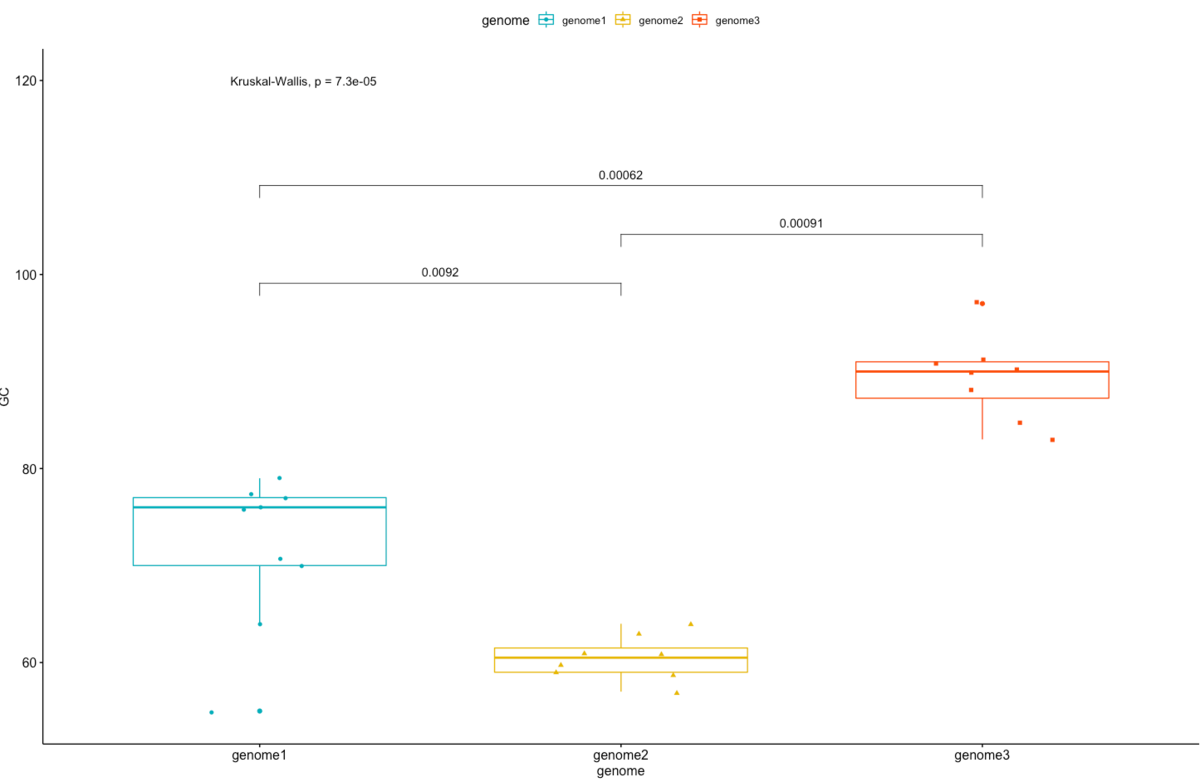
追記
7つ以上データ(ここではgenome)がある場合、手動で色を指定する必要がある。
Githubやマニュアルには他にも様々な作図例が掲載されています。確認して下さい。
引用
Ggpubr: ‘Ggplot2’ Based Publication Ready Plots
Alboukadel Kassambara
2018 R Package Version 0.2. Available online: https://CRAN.R-project.org/package=ggpubr
*1
下の様なexcelライクなフォーマットの方が見やすいが、全ての列で行数が同じなどの制約がある。さらに、excelライクなフォーマットだとRに読み込んでから一手間かかる。
| genome1 | genome2 |
| 77 | 67 |
| 64 | 54 |
| 70 | 60 |
| 55 | 45 |
| 77 |
67 |
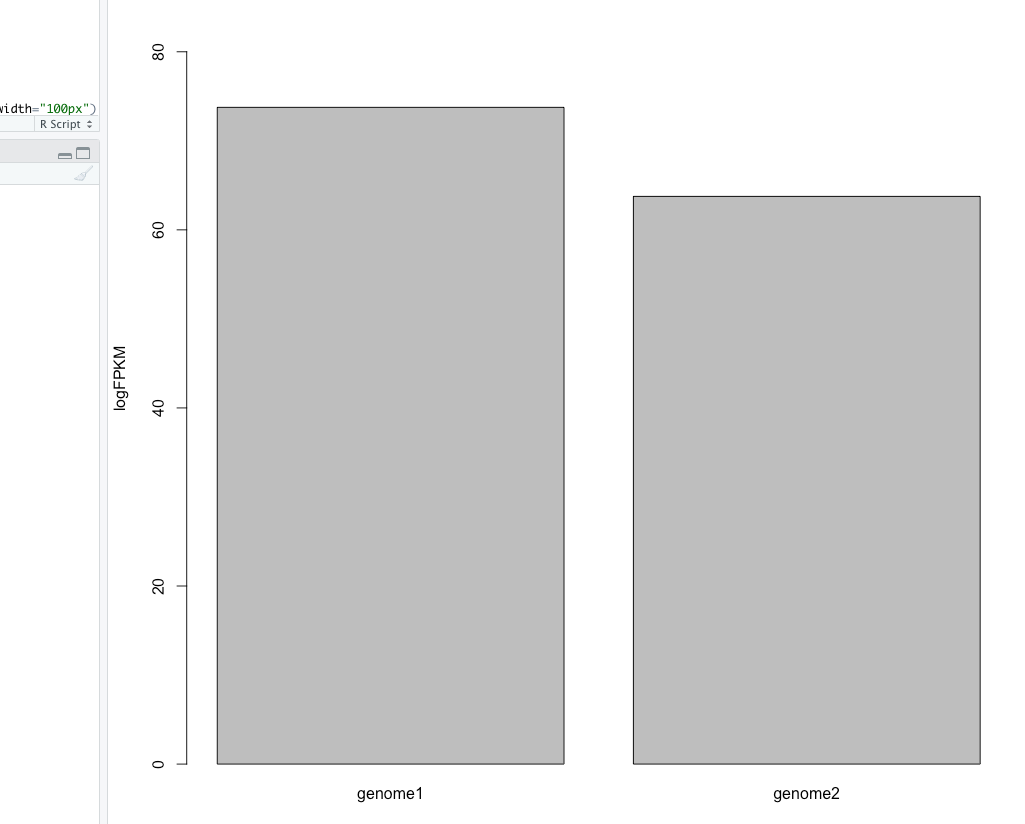
のようなデータのbar plotを出力するなら
#読み込み
x <- read.table(pipe("pbpaste"), header = TRUE)
#名前、平均、SD
colnames(x) <- c("genome1", "genome2") #カラム名を再アサイン(スキップ可)
xmean <- apply(x, 2, mean) #平均
xsd <- apply(x, 2, sd) #S.D
#プロット
plot <- barplot(bindmean, xlab = "name", ylab = "logFPKM", ylim = c(0, max(xmean + xsd)))
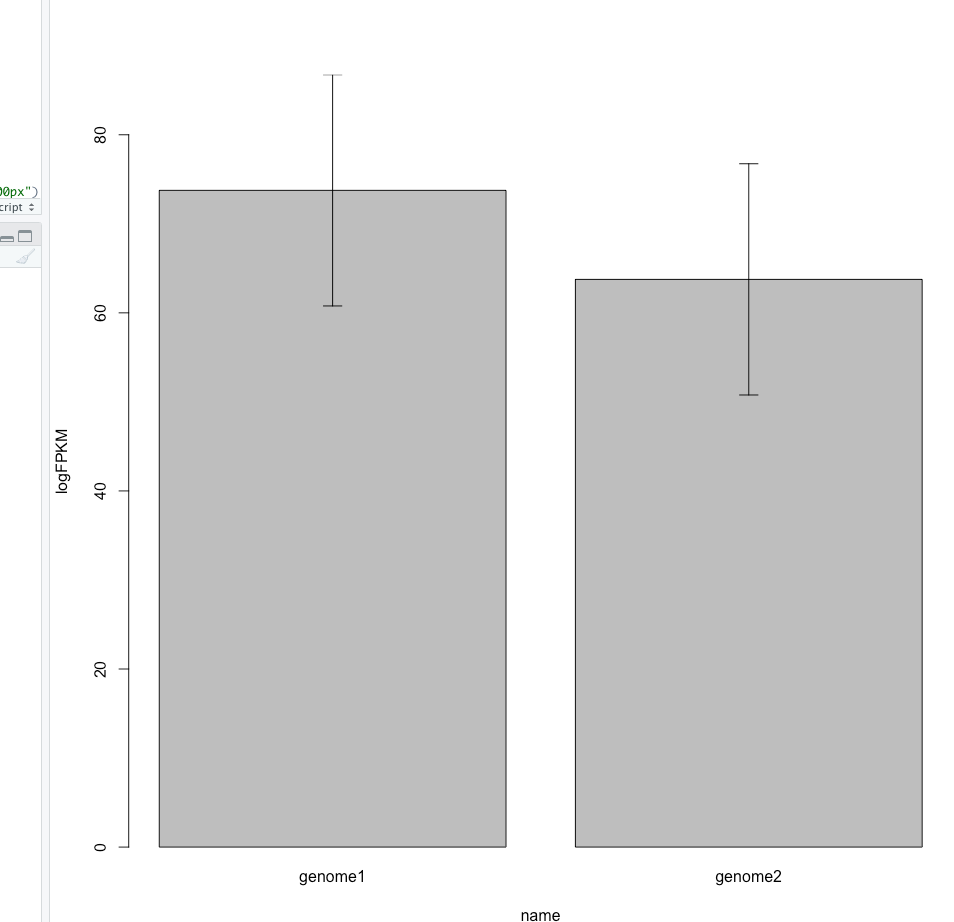
さらにエラーバーをつける。
arrows(plot, xmean - xsd, b, xmean + xsd, code = 3, lwd = 1, angle = 90, length = 0.1)
このように、視覚化まで時間がかかる。整然フォーマットのデータを準備し、さらにggpubrを使うことで煩雑なコードを削減できる。
参考
関連
2020 11/28