精密腫瘍学における現在の課題は、患者における腫瘍の進行を追跡する能力である(例えば治療への反応)。これに対する古典的なアプローチは、異なる時点で組織生検を実施することであろう。これは高価で時間がかかるプロセスであり、そしてこれは侵襲的処置であるので、患者にとって困難であり得る。さらに、腫瘍が転移した場合、生検はさらに困難または不可能になる。より魅力的な代替手段は、患者の血液や尿から循環する無細胞DNA(cfDNA)をシーケンスすることである。これは、これらの欠点を抱えていない。
cfDNAの存在は数十年前から知られており、1948年に発見された(ref.1)。このようなDNAは、主に細胞のアポトーシス、壊死、および能動的な放出によって血中に発生する(ref.2)。これらの細胞の特定の部分、ひいてはDNAは、腫瘍に由来する可能性があり、循環腫瘍DNA(ctDNA)として知られている(ref.3)。実際、cfDNAレベルは健康なコントロールと比較して癌患者では200倍も上昇することが示されている(ref.2)。 ctDNAの占める割合は患者間で大きく異なり( 0.003%〜95%)(ref.4、5)、そして初期の腫瘍では、進行した疾患または転移よりも低くなる傾向がある(redf.6)。ctDNAは任意の腫瘍部位(原発性または転移性であり得る)および任意の腫瘍subpopulation/クローンに由来し得ることに留意されたい。
定量的PCR(qPCR)またはドロップレットデジタルPCR(ddPCR)を用いた単一遺伝子座アッセイは、白血病、膵臓および結腸直腸ガンの変異を検出するために首尾よく使用されている(ref.7-9)。ごく最近では、NGSアプローチが肺ガンと前立腺ガンの変異を検出するために使用されており(ref.10,11)、感度の低下を伴うが1回の実行でより多くの遺伝子座をカバーできる(ref.12,13)。我々(著者ら)のアプローチはNGSデータに頼っているので、非常に少量のctDNAしか含まないサンプルのシーケンシングのリミットのために感度が制限される。このような場合、十分なリードカバレッジを確保するために、シーケンス処理は非常に深いデプスで行われる(通常は20 000倍、90 000倍以上になる可能性がある)。一般に、これを達成するために全エキソームまたはターゲットシーケンシングが使用される。
circulating fragmentはしばしば非常に短く、最頻値の長さ(167 bp)はヌクレオソームの周りを包むDNAの長さ(約147 bp)に関連している(ref.6, link)。多くの断片はさらに短く、50 bpから166 bpの間であり、一本鎖DNAライブラリー調製法を使用するとより効果的に検出できる(ref.14, link)。典型的なショートシーケンシングリードは75bpから150bpなので、ペアエンドシーケンシングは、両方のリードの末端が互いオーバーラップしており、そして多くのリードはターゲット長より短い。これらの短い断片が最も一般的だが、はるかに長い断片(> 1000 bp)も観察される(ref.6)。非常に深いカバレッジデプスと共に、DNAソースおよびフラグメント長の変動性は、汎用のゲノム分析ツールを混乱させノイズの多いデータをもたらす可能性がある。
この研究では、cfDNAを使用したゲノム構造変異(SV)の検出に焦点を当てている。ゲノム構造変異は、2bp以上(通常5bp以上)ゲノムバリアントである。主な種類のSVには、欠失、挿入、重複(tandem or interspersed)、および逆位が含まれる。これらのいずれかが非常に大きなゲノム距離にわたって起こるか、または異なる染色体からの配列を含む場合、それらは転座として知られている。 SVが異なる遺伝子からのエクソンを隣接させるとき、それらは遺伝子融合を形成する。エキソン領域で観察される融合およびSVは、異常なタンパク質産物をもたらし得るか、または翻訳を完全に妨げ、そして疾患、特に癌と関連している。よく知られた例は、前立腺ガンにおけるTMPRSS2とETS遺伝子ファミリーの融合である(ref.15)。
VariationHunter(ref.16)のようなゲノム構造変異検出のための最初の方法の導入以来、構造変異検出の分野は、多くのツールを用いて様々なアプローチを用いて成熟してきた。すべてのツールに共通しているのは、構造的な変異の指標としての不一致リードおよび/またはスプリットリードの使用である。(一部略)
たとえば、Breakdancer(ref.17)とVariationHunter(16)は不一致マッピングのみを使用するが、Socrates(ref.18)などはほとんど分割またはソフトクリップリードを使用する。これらの戦略の組み合わせは、Lumpy 2(ref.19)、GRIDSS(ref.20)、Pindel(ref.21)、Delly 2(ref.22)などの他のツールによって採用されている。 cfDNAに対するこれらのツールの有効性は調査されておらず、著者らの知る限りでは、cfDNA用に調整されたSV callerは存在しませんない。具体的には、これらのcallerが(i)非常に深いカバレッジデプス、(ii)非常に低い希釈率、(iii)可変リード長、(iv)高い不均一性、および(v)高いシステマティックノイズを処理できるかどうかは不明である。
これらの課題により、cfDNAに合わせた最初のSV callerであるSViCT(Structural Variation detection in Circulating Tumor DNA)の開発が動機となった。 SViCTは、標準的なシーケンシングデータのBAMフォーマットから始まり、アセンブリ、k-merマッピング、およびグラフベースのアルゴリズムの組み合わせを通じて、すべての主要クラスの構造変異を予測する。予測は、ブレイクポイントのbase-pair resolution、SVタイプアノテーション、ゲノムコンテンツアノテーション(融合を含む)および供給源DNAの座標/配列を含む。ツールの性能は、10%、1%、0.5%および0.01%の希釈率を含む模擬cfDNAデータセットを用いて評価した。 SViCTは最低希釈度でも良好な感度を維持し、既存のSV callerよりも優れていた。 感度はリアルデータセットでさらに評価され、SViCTが既知のSVすべてを見つける唯一のツールであることが示された。最後に、SViCTを8つの転移性去勢抵抗性前立腺ガン(CRPC)患者サンプルのコホートに適用し、いくつかの高信頼性構造変異を発見した。
SViCTは、次の5つの段階でcfDNAを使用してゲノム、腫瘍特異的構造変異を予測する。(i)片端アンカー(OEA)およびソフトクリップ/スプリットリードの抽出およびクラスター化、(ii)各クラスターの局所集合すなわち、( iii) k-merインデックス付けおよびリファレンスへのマッピング、(iv)マッピングされた区間の連鎖および最適連鎖マッピングの計算、ならびに(v)構造的変形および融合識別。SViCTの要約を論文図1に示し、各段階の詳細は論文のサブセクションに記載されている。
Overview of the SViCT algorithm. 論文より転載
Our new SV and fusion detection for ctDNA method is out on @NAR_Open!
— Hach Lab (@HachLab) February 13, 2019
📄 paper: https://t.co/X2hXRStHQU
💻 code: https://t.co/4M9oQb79P3
💿 data: https://t.co/OPOiuPJ2kQ@VanProstateCtr #Bioinformatics #CompBio
インストール
依存
You will need at least g++ 4.9 to compile the source code.
本体 Github
git clone https://github.com/vpc-ccg/cfdna-sv.git
cd cfdna-sv && make
> ./svict
$ ./svict
SViCT does not accept the following parameter values:
Error: Reference Genome (specify by -r ) is required to predict SV
Check help message for more information
======================================================
| SViCT: Structural Variant in ctDNA Sequencing Data |
======================================================
-h|--help: Shows help message.
-v|--version: Shows current version.
Mandatory Parameters:
-i|--input: Input file. (SAM/BAM)
-r|--reference: Reference Genome. Required for SV detection.
Main Optional Parameters:
-o|--output: Prefix of output file
-g|--annotation: GTF file. Enables annotation of SV calls and fusion identification.
-s|--min_support: Min Read Support (default 2).
-S|--max_support: Max Read Support (default unlimited).
-m|--min_length: Min SV length (default 30).
-M|--max_length: Max SV length (default 20000).
Additional Parameters:
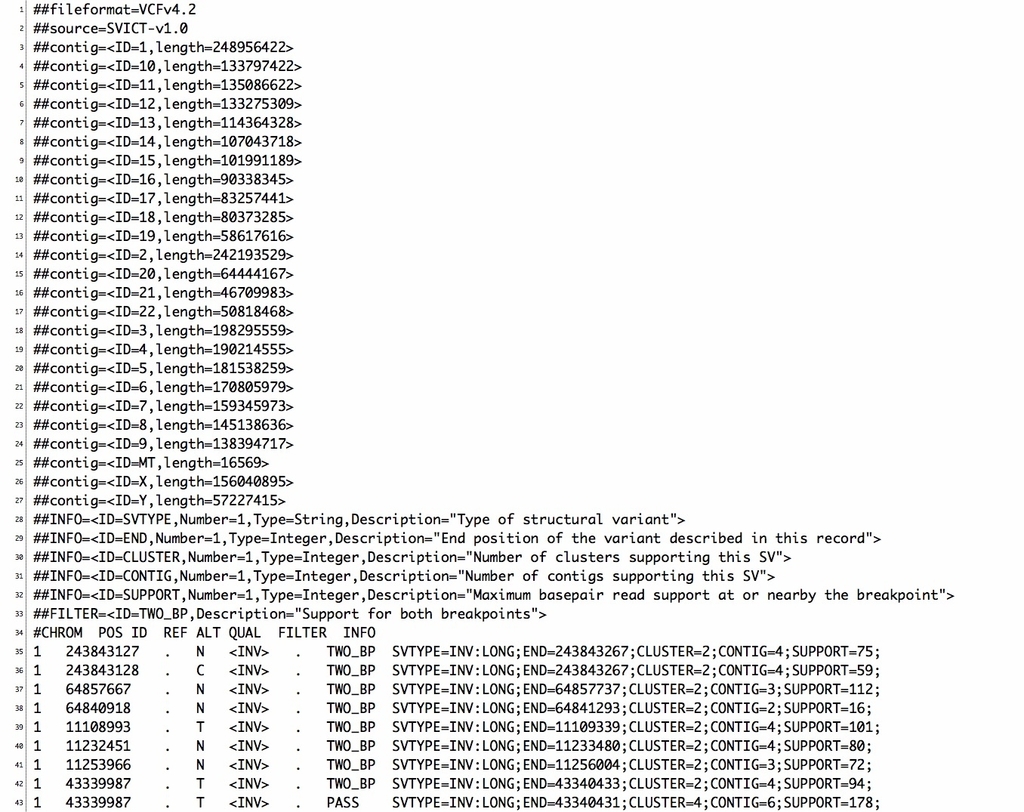
-p|--print_reads: Print all contigs and associated reads as additional output.
-P|--print_stats: Print statistics to stderr.
-w|--window_size: Clustering window (default 3).
-d|--min_sc: Minimum soft clip to consider (default 10).
-n|--no_indel: Disable indel parsing (I and D in cigar).
-O|--assembler_overlap: Required read overlap for assembly (default 50).
-a|--anchor: Anchor length (default 30).
-k|--kmer: k-mer length (default 14).
-u|--uncertainty: Uncertainty (default 8).
-c|--sub_optimal: Maximum difference from longest path (default 0 - co-optimals only, negative value disables).
-H|--heuristic: Use clustering heuristic (good for data with PCR duplicates).
-D|--dump_contigs: Dump contigs in fastq format for mapping.
-R|--resume: Resume at the interval chaining stage with mapped contigs.
Example Usage:
./svict -i input.bam -r human_genome.fa -o final
This command will generate prediction result final.vcf directly from input.sam.
kazu@edb2e2639563:~/cfdna-sv$
テストラン
ダウンロードリンク
https://figshare.com/articles/Simulation_Datasets_for_Evaluation/5758539
bamとリファレンスfastaを指定する。
svict -i sim.150.sorted.bam -r Homo_sapiens.GRCh38.87.dna.chromosomes.fa
- -i Input file. (SAM/BAM)
- -r Reference Genome. Required for SV detection
- -o Prefix of output file
引用
Structural variation and fusion detection using targeted sequencing data from circulating cell free DNA
Alexander R Gawroński, Yen-Yi Lin, Brian McConeghy, Stephane LeBihan, Hossein Asghari, Can Koçkan, Baraa Orabi, Nabil Adra, Roberto Pili, Colin C Collins, S Cenk Sahinalp, Faraz Hach
Nucleic Acids Research, Published: 13 February 2019
関連
