2020 10/9 リンク追加
複数の配列のblast解析を行う場合、ローカルでデータベースなどを構築して進めるのが一つの手である。しかしローカルだとデータベースの更新や、データサイズが問題になる(例えばnrのデータも2015年にダウンロードすると200GBを超えていた)。
ネットワーク越しのランで十分な速度が得られる限りは、専門のアノテーターが管理している最新のデータベースを使うのがベストである。そのためにコマンドラインからネットワーク越しにblast解析を行う方法も用意されているが、特定のポートを開ける必要があり、環境によってはやや敷居が高い。今回はNCBIにプラウザ越しにアクセスして、数百のcontigのblast解析を行う方法を書いていく。
後半では、全blast結果をテキストでダウンロードし、それからbest hitのIDを取り出し生物名を得る流れをまとめている。ダウンロードされたファイルにはヒットした生物のEntrez_IDしか書かれていないので、生物名を得るためにUNIXのコマンドとNCBIのツールを組み合わせている。
生物系研究者の方はSafariなどのブラウザを使ってNCBIにアクセスされている人が多いと思います。その時、全配列を同時にblastにかけて結果をダウンロードする方法を知っていると便利です。一例ですが参考にしてみてください。
8/20 追記
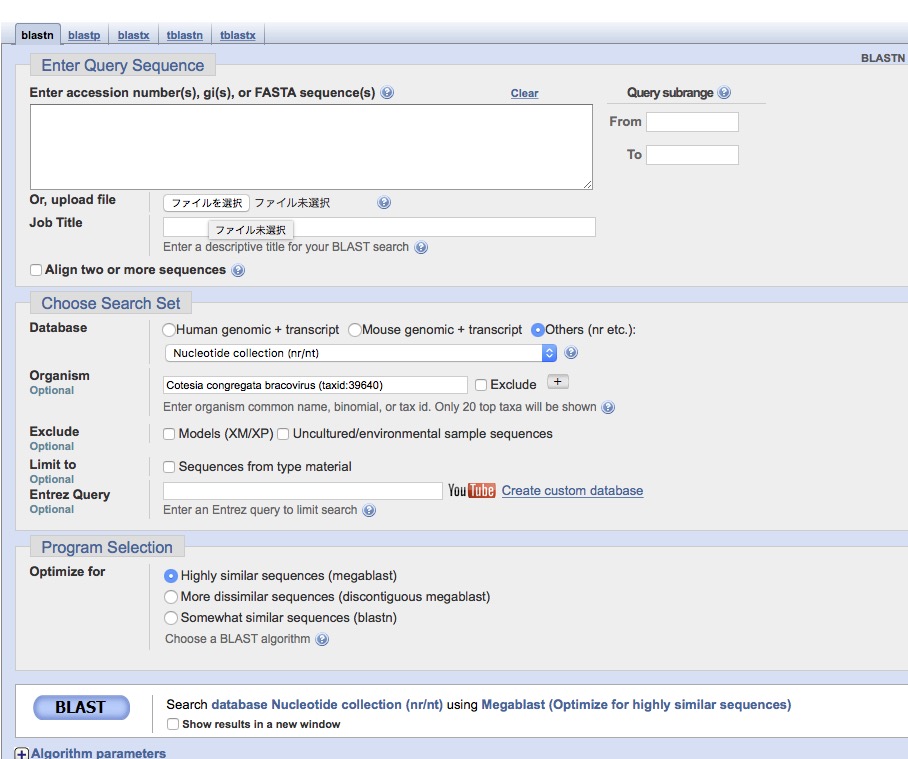
NCBIのNucleotide blastに移動する。
Nucleotide blastを選択。
ウィンドウ下のファイルを選択、をクリック。
生物を限定するならorganismの項目に学名を書く。
de novoアセンブルして得たcontigファイルを選択。
BLAST解析を実行する。
途中でページ更新に失敗した場合、1ページ戻り右上のRecent resultsを選択(recent resultsリンク)。
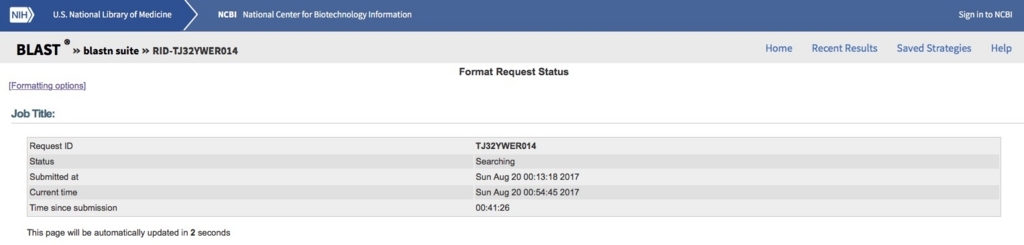
IDをクリックすれば再開できる。
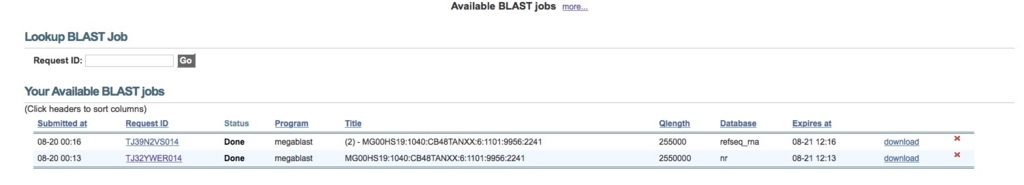
ジョブが終わっている場合、右端の downloadでダウンロード可能(xmlファイルのみ)。
数分で結果が得られた。
上の方のダウンロードをクリック。
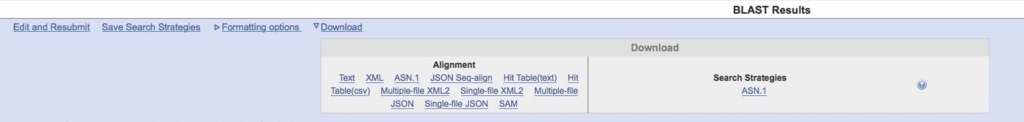
Hit Table(text) を選択してダウンロード。
ダウンロードされたファイルにはhitした全ての部位の情報がタブ形式で保存されている。
usr $ head alignment.txt
# blastn
# Iteration: 0
# Query: NODE_1_length_40202_cov_39.6477
# RID: SS3HY4K7014
# Database: nr
# Fields: query id, subject ids, query acc.ver, subject acc.ver, % identity, alignment length, mismatches, gap opens, q. start, q. end, s. start, s. end, evalue, bit score
# 264 hits found
NODE_1 gi|545742605|emb|FR873486.1| NODE_1 FR873486.1 83.535 2976 407 41 9041 11995 12939 15852 0.0 2704
NODE_1 gi|545742605|emb|FR873486.1| NODE_1 FR873486.1 84.876 2215 295 22 34599 36796 838 3029 0.0 2198
NODE_1 gi|545742605|emb|FR873486.1| NODE_1 FR873486.1 85.036 1517 197 19 12592 14100 17670 19164 0.0 1517
hitしたIDのところはEntrezのIDとembのIDだが、このままでは何がヒットしたのか分かりにくい。IDをコンバートしてわかりやすく修正する。
query_IDとsubject_IDだけ取り出す。
cut -f 1-2 Alignment.txt > subject_and_query_ID
head -15 subject_and_query_ID
# blastn
# Iteration: 0
# Query: NODE_1_length_40202_cov_39.6477
# RID: SS3HY4K7014
# Database: nr
# Fields: query id, subject ids, query acc.ver, subject acc.ver, % identity, alignment length, mismatches, gap opens, q. start, q. end, s. start, s. end, evalue, bit score
# 264 hits found
NODE_1 gi|545742605|emb|FR873486.1|
NODE_1 gi|545742605|emb|FR873486.1|
NODE_1 gi|545742605|emb|FR873486.1|
NODE_1 gi|545742605|emb|FR873486.1|
NODE_1 gi|545742605|emb|FR873486.1|
NODE_1 gi|545742605|emb|FR873486.1|
NODE_1 gi|545742605|emb|FR873486.1|
NODE_1 gi|545742605|emb|FR873486.1|
1つのcontigに複数当たっている。ここではbest hitだけで十分なので、uniqコマンドを使い1列目のNODE名が重複する行を消す。awkにパイプしてコメント行の削除もワンライナーで行う。
uniq -f 2 subject_and_query_ID | awk '! /^#/' > subject_and_query_ID_unique
user$ head -5 subject_and_query_ID_unique
NODE_1 gi|545742605|emb|FR873486.1|
NODE_2 gi|332139268|gb|HQ009547.1|
NODE_3 gi|531033897|emb|HF586473.1|
NODE_4 gi|190702157|gb|EF710641.1|
NODE_5 gi|54109776|emb|AJ632317.1|
重複が除けたので、ここからsubject_IDだけ保存する。
cut -f 2 subject_and_query_ID_unique > subject_ID_unique
user$ head subject_ID_unique
gi|545742605|emb|FR873486.1|
gi|332139268|gb|HQ009547.1|
gi|531033897|emb|HF586473.1|
gi|190702157|gb|EF710641.1|
gi|54109776|emb|AJ632317.1|
gi|332139193|gb|HQ009535.1|
gi|531033897|emb|HF586473.1|
gi|531033897|emb|HF586473.1|
gi|531034048|emb|HF586474.1|
gi|531033897|emb|HF586473.1|
Entrez IDだけ保存する。
cut -f 2 -d "|" subject_ID_unique > Entrez_ID
user$ head Entrez_ID
545742605
332139268
531033897
190702157
54109776
332139193
531033897
531033897
531034048
531033897
IDリストが入手できた。
ここから先はDAVIDの変換ツールを使っても良いが、今回はEntrez Directのコマンドを使う。ターミナルで以下のコマンドをペーストしてツールをダウンロード。
cd ~
perl -MNet::FTP -e
'$ftp = new Net::FTP("ftp.ncbi.nlm.nih.gov", Passive => 1);
$ftp->login; $ftp->binary;
$ftp->get("/entrez/entrezdirect/edirect.tar.gz");'
gunzip -c edirect.tar.gz | tar xf -
rm edirect.tar.gz
export PATH=$PATH:$HOME/edirect
./edirect/setup.sh
ホームにedirect/ができる。パスを通しておく。
echo 'export PATH=$PATH:/Users/user/edirect/' >> ~/.bash_profile
source ~/.bash_profile
efetchコマンドでxmlファイルを出力し、xmlからxtractコマンドで<tile>を抽出する。IDを1つだけ探すなら以下のようなコマンドになる。
efetch -id ID -db nucleotide -format docsum | xtract -pattern DocumentSummary -element 'Title' #IDのところはEntrez_IDの番号を入力。
実行例
user$ efetch -id 545742605 -db nucleotide -format docsum | xtract -pattern DocumentSummary -element 'Title'
Cotesia congregata bracovirus segment 28, complete sequence
前半の出力は下のようなxmlファイルである。

Titleの項目が生物名に使えそうである。
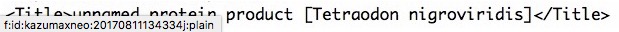
この行のタグの間をEntrez Directのxtractコマンドで抽出している。
今回はEntrez_IDのリストに対して実行するので、ループで順番に処理させる。
while read line; do title=$(efetch -id $line -db nucleotide -format docsum | xtract -pattern DocumentSummary -element 'Title'); echo "$line $title"; done<Entrez_ID > title_list.txt
user$ head -5 output
545742605 Cotesia congregata bracovirus segment 28, complete sequence
332139268 Cotesia vestalis bracovirus segment c24, complete sequence
531033897 Cotesia congregata sequence containing Cotesia congregata bracovirus proviral locus 2 (PL2)
190702157 Cotesia sesamiae Mombasa bracovirus clone BAC 6L7, complete sequence
54109776 Cotesia congregata virus complete genome, segment Circle14
IDから<Title>部分の情報に変換できた。
このような流れで、全contigをblastにかけてベストヒットのアノテーションリストを入手できる。
集計する。1列目は数値順、2列めは辞書順でソート。
sort -k 1,1n -k 2 title_list.txt |uniq -c |sort -r > ranking.txt
591 1228866639 Papilio machaon Ycf2-like protein
7 1228866639 PREDICTED: Helianthus annuus
7 1228808943Medicago truncatula cytochrome b559
2 1044533238 PREDICTED: Vigna angularis LINE-1
.
.
.
省略
上のような集計結果を出力できた。大半のcontigはPapilio machaonにbest hitしていた。
今回はリストを入手しbest hitだけ残してblast解析を行ったが、NCBIのblast結果からxmlファイルもダウンロードできる。ケースによってはそちらを使った方が早い。また行程がやや煩雑なので、頻繁に行うならもう少し工夫してスクリプト化した方が良いと思います。blastp、blastx等も流れは同じです。
追記
https://ncbiinsights.ncbi.nlm.nih.gov/2018/06/14/5-new-youtube-videos-blast-medgen-pubchem-api-keys/
NCBIの中の人のプレゼンテーションのです。投稿日時は2018/05/17。ほぼような同じことをやっていますが、ずっと包括的な説明になっていています。23分あります。詳しく知りたい方はどうぞ。
blastコマンドもバージョンアップしています。また紹介します。
ゲノム(fasta)、シーケンスデータ(sam, bam, fastq)のtophit 生物種を解析したいなら、minHashを使うBBsketchが圧倒的に高速です。fastq 10万リードでも数秒でtop10 hitをrefseqから検索してくれます。blastの結果と比較してみてください。
追記
localにダウンロードして検索したいときはこちらを参考にして下さい。ダウンロードツールと、それをデータベースにしてblast検索する流れをまとめています。
引用
Entrez Direct: E-utilities on the UNIX Command Line
https://www.ncbi.nlm.nih.gov/books/NBK179288/
Biostar
https://www.biostars.org/p/110000/
関連