EMBOSSのpepstatsは、1つ以上のタンパク質配列からタンパク質の特性に関する様々な統計情報を出力する。
emboss pepstats
https://emboss.sourceforge.net/apps/cvs/emboss/apps/pepstats.html
インストール
conda|mambaやbrewで導入できる。
#bioconda
mamba install -c bioconda -y emboss
#homebrew
brew install emboss
> pepstats -h
実行方法
入力のEST配列とgenome.fasta、出力名を指定する。est2genomeとだけ叩くと、対話モードで実行できる。
pepstats input.faa out.stats
#対話モード
pepstats
> pepstats -h
Calculate statistics of protein properties
Version: EMBOSS:6.6.0.0
Standard (Mandatory) qualifiers:
[-sequence] seqall Protein sequence(s) filename and optional
format, or reference (input USA)
[-outfile] outfile [*.pepstats] Pepstats program output file
Additional (Optional) qualifiers: (none)
Advanced (Unprompted) qualifiers:
-aadata datafile [Eamino.dat] Amino acid properties
-mwdata datafile [Emolwt.dat] Molecular weight data for amino
acids
-pkdata datafile [Epk.dat] Values of pKa for amino acids
-[no]termini boolean [Y] Include charge at N and C terminus
-mono boolean [N] Use monoisotopic weights
General qualifiers:
-help boolean Report command line options and exit. More
information on associated and general
qualifiers can be found with -help -verbose
入力配列

出力
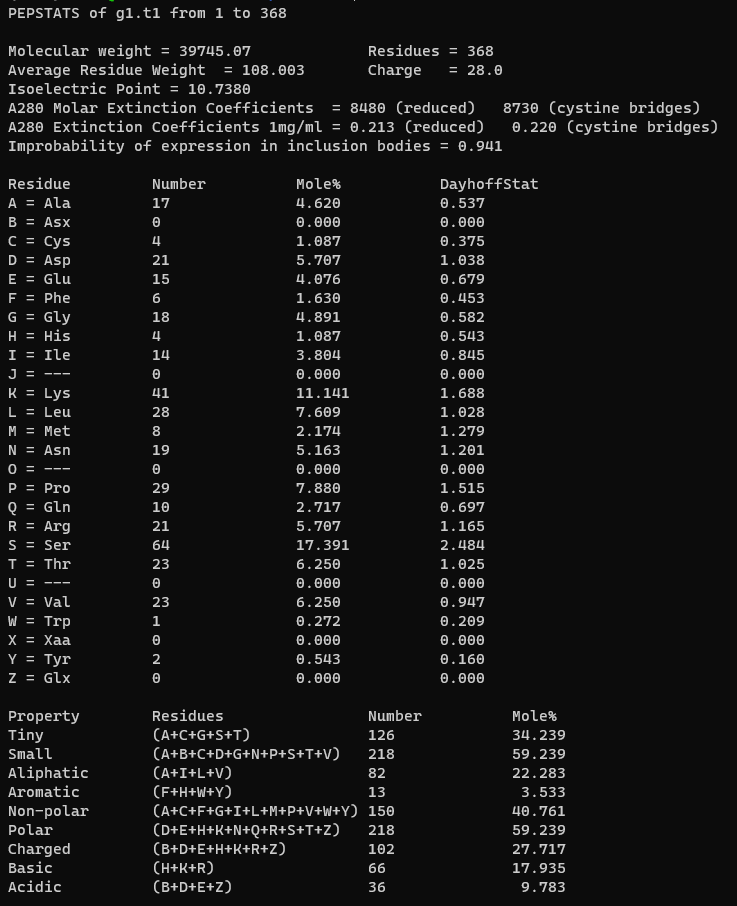
上の出力には以下が含まれる。
- 分子量(Molecular weight)
- 残基数(Residues)
- 平均残基重量(Average Residue Weight)
- 荷電(Charge)
- 等電点(Isoelectric Point)
- A280モル吸光係数 (A280 Molar Extinction Coefficients)
- A280モル吸光係数 1mg/ml
- インクルージョンボディとして発現する確率(Improbability of expression in inclusion bodies)
- アミノ酸の種類ごとに:数、mol %、DayhoffStat
- 各アミノ酸の物理化学的クラス:数、モルパーセント
その他
- multi-fastaを指定した場合、配列ごとに統計が計算されて出力される。
- 使用されているHarrisonモデルによってあるタンパク質がおそらく封入体で発現すると予測されても、細胞質に溶けないという意味ではない。タンパク質が封入体で発現するかどうかは配列だけでは確実には予測できず、ほかの多くの要因にも依存している(レポジトリ参照)。
- embossのサイトでは、類似サブコマンドとして以下も紹介されている。
- backtranseq タンパク質配列を逆翻訳する
- charge タンパク質の電荷プロット
- checktrans タンパク質のSTOPコドンとORFの統計情報を報告
- compseq 配列中の二量体/三量体/その他のワード数を数える
- emowse 質量分析によるタンパク質の同定
- freak 残基/塩基頻度表またはプロット
- iep タンパク質の等電点を計算
- mwcontam 一連のファイル間で一致するmolwtsを表示
- mwfilter 質量分析出力からノイズの多いmolwtsをフィルタリング
- octanol タンパク質のハイドロパシーを表示
- pepinfo 単純なアミノ酸特性を並列にプロット
- pepwindow タンパク質のハイドロパシーを表示
- pepwindowall 一連の配列のタンパク質ハイドロパシーを表示
引用
EMBOSS: the European Molecular Biology Open Software Suite.
Rice P, Longden I, Bleasby A
Trends Genet. 2000 Jun;16(6):276-7
EMBL-EBIでも使用可能
https://www.ebi.ac.uk/Tools/seqstats/emboss_pepstats/
関連