2020 1/25 タイトル修正、統合TVリンク追加
シーケンシングコストの劇的な低下により、個人や研究者グループが以前に研究されていない生物からゲノムまたはトランスクリプトーム配列を生成する多くの機会が生まれている。多くの研究の疑問には、小規模または大規模な配列比較が必要であり、BLAST(Basic Local Alignment Search Tool)は、このような多くの分析で最も確立されたツールである(Altschul et al、1990; Camacho et al、2009)。残念ながら、新しいデータのBLAST分析は困難な場合がある。 NCBI(National Center for Biotechnology Information)などのセントラルBLASTリポジトリに新しいデータが送信されて公開されるまでに遅延があり、そのようなリポジトリで実行できるクエリはごくわずかである。 BLASTはローカルにダウンロードしてインストールできるが、コマンドラインインターフェイスの経験のない研究者にとっては使用が難しい場合がある。最後に、このようなハードルを克服するための商用ソフトウェアは、多くの研究所にとって費用がかかりすぎる。
ここでは、カスタムデータセットでBLAST検索を実行および解釈する生物学者の研究者、および共有ラボまたはコミュニティデータベースを設定する生物情報学者の生産性を高めるために設計されたBLASTの無料のグラフィカルインターフェイスであるSequenceserverを紹介する。(一部略)
通常、macOSまたはLinuxを実行しているコンピューターへのSequenceserverのインストールは迅速で、必要なコマンドは1つまたは少数である(オンラインドキュメント参照)。
HP
http://sequenceserver.com/#installation
help
http://sequenceserver.com/#support
統合TV
インストール
依存
sudo gem install sequenceserver
> sequenceserver -h
$ sequenceserver -h
SUMMARY
custom, local, BLAST server
USAGE
sequenceserver [options]
EXAMPLE
# Launch SequenceServer. This will read configuration from
# ~/.sequenceserver.conf, if present.
$ sequenceserver
# Use a different config file.
$ sequenceserver -c ~/.sequenceserver.ants.conf
# Set number of threads to use. This will save the number
# of threads to use in config file.
$ sequenceserver -s -n 16
# See if you have FASTA files in database dir that haven't
# been converted into BLAST database.
$ sequenceserver -u
# Search for FASTA files in database dir that haven't been
# converted into BLAST database yet, and convert them.
$ sequenceserver -m
DESCRIPTION
SequenceServer lets you rapidly set up a BLAST+ server with an intuitive user
interface for use locally or over the web. If BLAST+ is not installed on your
system, SequenceServer will offer to install BLAST+ for you. You should only
ever have to point it to a directory of FASTA files / BLAST+ databases.
In a given directory, SequenceServer is able to tell FASTA files that are yet
to be formatted for use with BLAST+ and format them, and FASTA files that are
already formatted for use with BLAST+, heuristically skipping all other files
in the directory. Directories are scanned recursively. Type of sequences in a
FASTA file is detected automagically. `parse_seqids` and `hash_index` options
of `makeblastdb` are used to create BLAST+ databases.
-c, --config_file Use the given configuration file
--config Same as --config_file (deprecated)
-b, --bin Load BLAST+ binaries from this directory
-d, --database_dir Read FASTA and BLAST database from this directory
-n, --num_threads Number of threads to use to run a BLAST search
-r, --require Load extension from this file
-H, --host Host to run SequenceServer on
-p, --port Port to run SequenceServer on
-s, --set Set configuration value in default or given config file
-m, --make-blast-databases Create BLAST databases
--download-taxdb Download the taxdb files
-l, --list_databases List BLAST databases
-u, --list-unformatted-fastas List unformatted FASTA files
-i, --interactive Run SequenceServer in interactive mode
-D, --devel Start SequenceServer in development mode
-v, --version Print version number of SequenceServer that will be loaded
-h, --help Display this help message.
使い方
データベースのfasta配列があるディレクトリに移動して、sequenceserverを立ち上げる。データベースとする配列をグループごとにサブディレクトリにまとめておくと、ラン時にそのデータベースを指定できる。
cd path/to/database_fasta/
sequenceserver
データベースのパスを聞かれる。カレントにあるなら空白のままエンターキーを押して確定する。

http://localhost:4567にアクセスする。
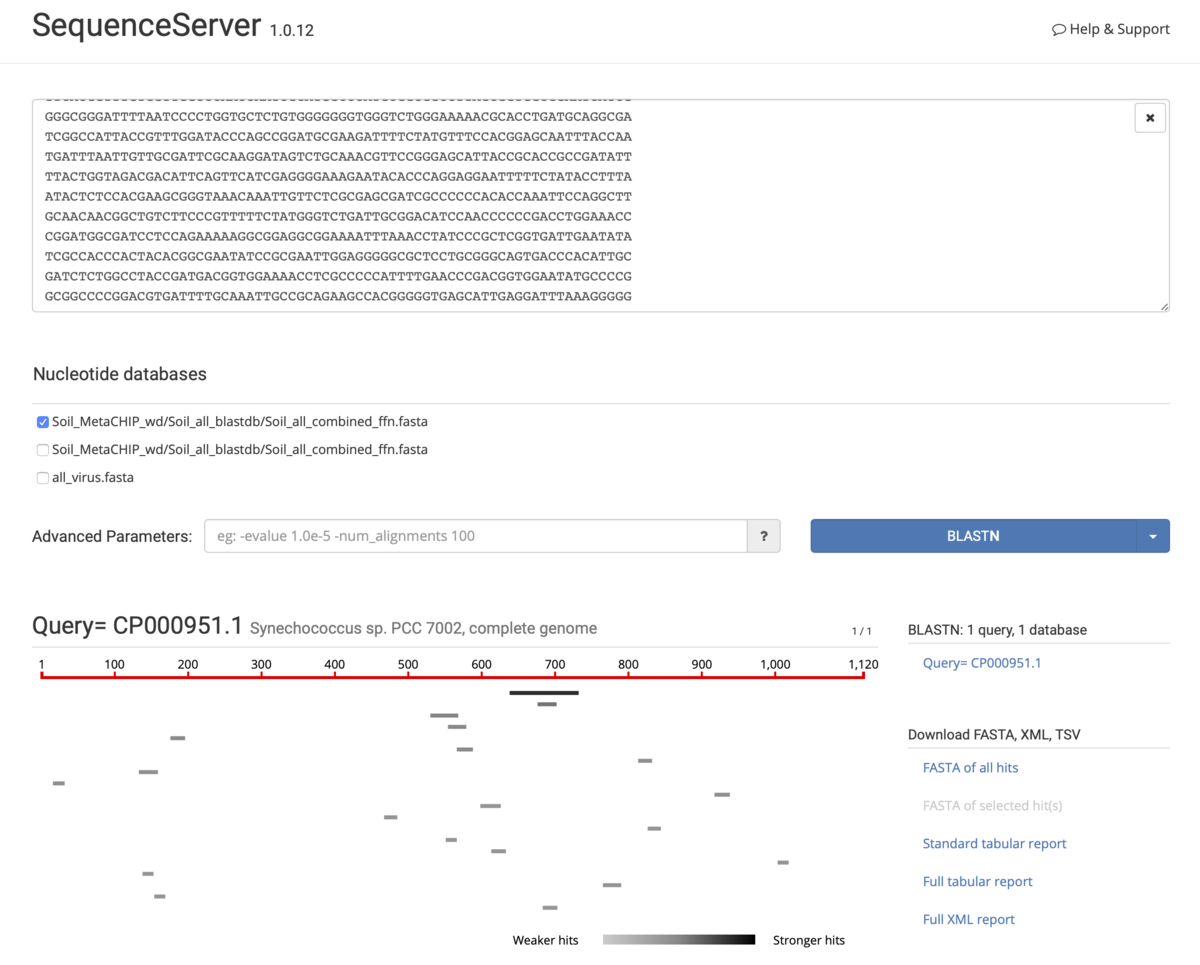
カレントのfastaとサブフォルダにあるfasta配列が自動で認識され、データベースとなる。配列をウィンドウに貼り付け、データベース(カレントにあるfastaごとにデータベースになっている)を指定して実行する。


e-valueとblastn/tblastxかを選択して実行する。

実行結果

引用
Sequenceserver: A Modern Graphical User Interface for Custom BLAST Databases
Anurag Priyam, Ben J Woodcroft, Vivek Rai, Ismail Moghul, Alekhya Munagala, Filip Ter, Hiten Chowdhary, Iwo Pieniak, Lawrence J Maynard, Mark Anthony Gibbins, HongKee Moon, Austin Davis-Richardson, Mahmut Uludag, Nathan S Watson-Haigh, Richard Challis, Hiroyuki Nakamura, Emeline Favreau, Esteban A Gómez, Tomás Pluskal, Guy Leonard, Wolfgang Rumpf, Yannick Wurm
Molecular Biology and Evolution, Published: 14 August 2019
関連