2019 7/6 インストール、ラン追記
MyCCは全プロセスを自動化したメタゲノム解析ツール。contigのfastaファイルを入力すると、配列の特性に従って自動で分類し、binning向けに色がついた図を描画し、さらにクラスタリングされたfastaまで出力することができる。既存のカバレッジやペアリードのつながりなどで分類する手法と比較して、クラスタリング精度が高いと言われている。また、全プロセスは完全に自動化されている。dockerのイメージや仮想イメージからMyCCを起動すれば、fastaを入力するだけで全て自動でランして結果を返してくれる。
解析の流れは以下のようになっている。
1、prodicalでコード領域を予測。
2、fetchMGを使い、シングルコピーでユニバーサルなオーソログ遺伝子40を抽出。
3、speciesレベルのマーカー遺伝子をUCLUSTで抽出。
4、4-mer頻度からgenomic signitureを分析し、Barnes-HUt-SNEで2次元に分類。
5、非階層的クラスタリング手法のaffinity propagationを使いplotをクラスタリング。
インストール
ランにはfetchMG、Prodigal、UCLUSTなどが必要でであるが、オーサーらによりovaファイルが配布されている。virttual PC内で仮想イメージを起動することで、すぐにランできるようになっている。
ovfファイルのダウンロード
https://sourceforge.net/projects/sb2nhri/files/MyCC/
- Virtualboxダウンロード=> リンク(OSXを選択)
- VMware=>リンク
VMwareへのインポートはこちらでは検証していません。
他にもdockerイメージやソースファイルが提供されている。
sb2nhri - Browse /MyCC at SourceForge.net
dockerイメージ
docker pull 990210oliver/mycc.docker:v1
#run
docker run 990210oliver/mycc.docker:v1 MyCC.py
> docker run 990210oliver/mycc.docker:v1 MyCC.py -h
Usage:
MyCC.py [inputfile] [4mer/5mer/56mer default:4mer] [Options]
Options:
-a A file having coverage information.
-t Minimum contig length. [defaults: 1000 bp]
-lt A fraction of contigs for the first-stage clustering. [default: 0.7]
-ct Minimum contig lengh for first stage clustering. [bp]
-meta Change to meta mode of Prodigal. [default: single]
-p Perplexity for BH-SNE. [default: 20, range between 5 and 50]
-pm To set preferences all equal to the median of the other similarities for affinity propagation.
-st Maximum distance for sparse format of affinity propagation. [default: 500]
-mask To mask repetitive sequences.
-keep To keep temporary files.
ラン
virtual PC内にfastaを読み込ませる。

ターミナルからMyCCをラン。
MyCC.py metagenomic_contigs.fasta
dockerイメージを使ってランするなら
#カレントにmetagenome_contigs.fastaがある状態で以下のように打つ
docker run --rm -itv $PWD:/data/ -w /data \
990210oliver/mycc.docker:v1 MyCC.py metagenome_contigs.fasta
テストでIndex of /dna/RD/Metagenome_RD/MetaBAT/Software/MockupのMockメタゲノム(25 bacteriaを混ぜた擬似メタゲノムのシーケンスデータ)のアセンブルデータを解析してみた。100MBのfastaファイルだったが、解析は30分程度で終了した。
出力された図。
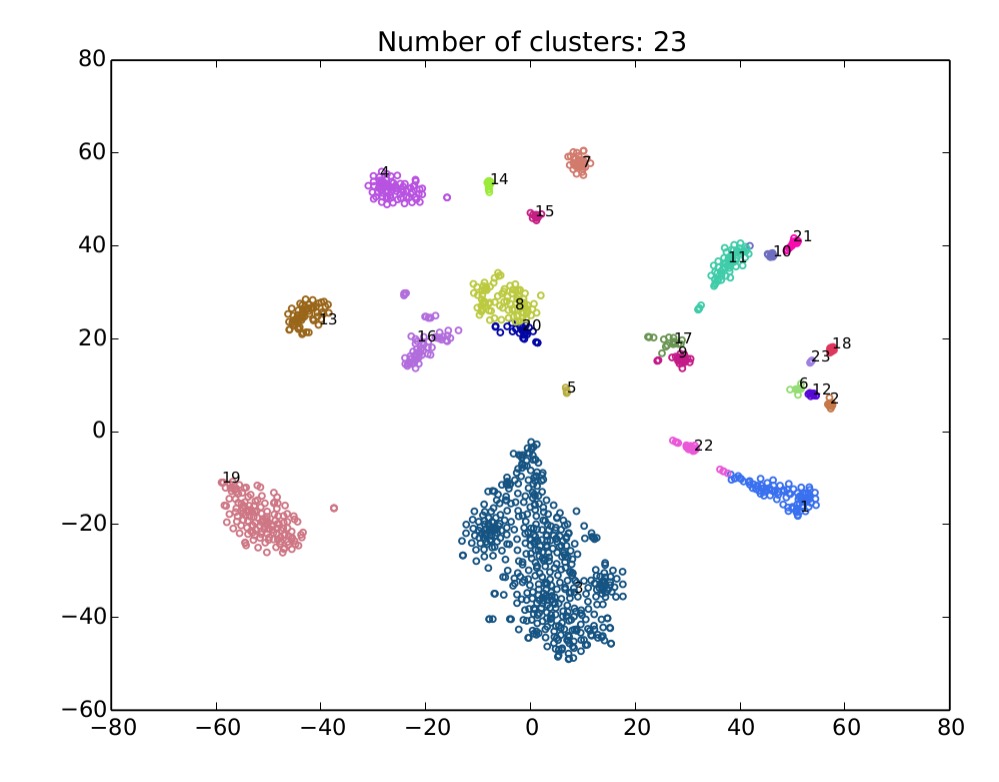
23クラスター認識されている。
Cluster.summaryを開く。
user$ cat /Users/user/Downloads/Cluster.summary
Cluster WholeGenome N50 NoOfCtg LongestCtgLen AvgLenOfCtg Cogs
Cluster.1.fasta 4070502 70156 97 268872 21423 35
Cluster.2.fasta 4256229 524907 13 896543 192492 35
Cluster.3.fasta 11381431 24260 981 325606 5820 21
Cluster.4.fasta 3613209 84946 92 176933 19893 35
Cluster.5.fasta 3944578 1281749 7 1811839 441941 35
Cluster.6.fasta 4833441 1145236 12 1295155 203365 42
Cluster.7.fasta 2161903 112927 36 253843 32019 36
Cluster.8.fasta 8313943 163761 96 478860 44284 35
Cluster.9.fasta 3259866 254968 28 556331 63405 36
Cluster.10.fasta 3624126 555508 11 777357 178603 37
Cluster.11.fasta 4329879 100800 68 289305 32544 39
Cluster.12.fasta 4735197 604725 11 1268240 226359 32
Cluster.13.fasta 5575100 131134 60 388470 47221 33
Cluster.14.fasta 3261204 901082 15 1093628 132980 35
Cluster.15.fasta 1862577 233546 14 499879 81212 36
Cluster.16.fasta 5338418 159508 67 481874 41626 34
Cluster.17.fasta 4012035 625944 20 1020404 125987 35
Cluster.18.fasta 3176251 454949 13 747195 135258 37
Cluster.19.fasta 3611880 32457 178 121052 10257 36
Cluster.20.fasta 1749350 134168 26 274886 38596 33
Cluster.21.fasta 4087425 443282 16 712360 147145 36
Cluster.22.fasta 5209287 387726 27 926513 99767 35
Cluster.23.fasta 2125683 1546362 5 1546362 309272 36
そのほか、affinity propagationによってクラスタリングされたfastaも保存されている。
リアルデータ(Lake_Huron_Sinkhole_Photosynthetic_Microbial_Mats_Metagenome)でテストしてみた(リンク)。15 GBx2のペアードエンドfastqのアセンブルにはmetaspadesを用いた(-k auto)。78000 contigできた。
下図:MyCC解析結果。

20クラスター検出された。
引用
Accurate binning of metagenomic contigs via automated clustering sequences using information of genomic signatures and marker genes
Hsin-Hung Lin & Yu-Chieh Liao
Scientific Reports 6, Article number: 24175
https://www.nature.com/articles/srep24175
Alignment-free Visualization of Metagenomic Data by Nonlinear Dimension Reduction
https://www.nature.com/articles/srep04516