2018 10/30 コード修正
2019 10/11 インストール追記、関連ツールリンク追記
2019 10/12 help追記、
2020 2/1 間違ったdockerリンク消去
de novo transcriptomeのアノテーションツールとしてblast2GOがよく知られているが、Trinotateというツールが発表された(論文はまだ)。Trinotateは非モデル生物のデータにも対応したde novo transcriptome向けアノテーションツールである。解析・付与できるアノテーション情報は、blastの解析結果、hmmerのドメイン検索結果、SignalPによるトランジットペプチドの予測結果、TMHMMによる膜タンパク質の予測結果、eggNOG/protein/Kegg databasesによるオーソログ、GO、 keggデータベースの情報などである。
各ツールを使った解析が終わると、Trinotateは各アノテーション結果を統合してSQLiteのデータベースにまとめてくれる。デーベース化することで柔軟かつ高速に取り扱うことが可能になる。データベースを作ったら、Trinotateのツールでローカルサーバーを立てて、htmlベースでグラフィカルにGO解析を行うことができる。
Blast2GOとの違いは、このペーパー
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4956038/#pone.0158565.ref026のFig .1がわかりやすいと思う。de novo transcriptome解析を行い、Blast2GOとTrinotateでアノテーションをつけている。
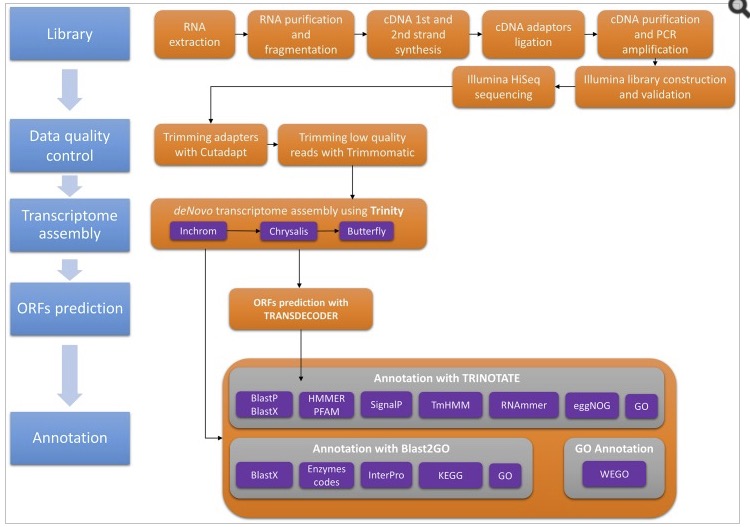
The De Novo Transcriptome and Its Functional Annotation in the Seed Beetle Callosobruchus maculatus より引用。
こちらも参考になる。
アノテーション情報を統合できるのは魅力的である。しかもblast2goの有償版と違いこちらは無料であり、高速に使えるなら心強い。論文発表前ではあるが、すでに7つの論文で使われている(リンクの一番下を参照)。導入して使い勝手をテストしてみる。問題がなさそうなら、普段の解析に取り入れることを目標に進める。
追記1; perlのライブラリを全て入れて、2017/12月の最新版を入れ直すと、最後までランできるようになりました。
追記2; condaを使って導入する。
まとめ直しました。
インストール
追記
一部依存を除き、condaでインストールできる(リンク)。
#bioconda 依存が多いツールは仮想環境を作って導入するのが無難
conda create -n trinotate
conda activate trinotate
conda install -c bioconda trinotate
> autoTrinotate.pl -h
$ autoTrinotate.pl -h
##############################################################################
#
# Required:
#
# --Trinotate_sqlite <string> Trinotate.sqlite boilerplate database
#
# --transcripts <string> transcripts.fasta
#
# --gene_to_trans_map <string> gene-to-transcript mapping file
#
# --conf <string> config file
#
# --CPU <int> number of threads to use.
#
#
##############################################################################
> Build_Trinotate_Boilerplate_SQLite_db.pl
$ Build_Trinotate_Boilerplate_SQLite_db.pl
usage: //anaconda3/envs/trinotate/bin/Build_Trinotate_Boilerplate_SQLite_db.pl Database_prefix [no_cleanup_flag]
以前苦労していた時==============================================
TMHMMがmacでは動かなかった(decodeanhmm.Linux_x86_64がlinux用にbuidされている)。cent OS6に導入する。
Trinotateのダウンロード(本体)
https://github.com/Trinotate/Trinotate/releases
perlのモジュールを適当な場所にコピーし、読み込めるようにしておく。
sudo cp -i ~/Trinotate-3.0.2/PerlLib/*pm /usr/share/perl5/
追記
その他、不足している、動かないperlのライブラリがあったら、
https://sourceforge.net/p/trinotate/mailman/message/31312982/の解答を参考に導入してください。私はリンク先の質問者と同じライブラリが入っていませんでした。
依存するもの
- blast+
- Trinity(utilityツールも使う)- Trinitiyのアセンブルデータを使うように設計されている
- TransDecoder - code領域探索
- sqlite - データベース
- NCBI BLAST+ - ホモロジー検索
- tmhmm v2 - 膜タンパク質探索
- RNAMMER - rNRA探索
- HMMER - PFAMに対してドメイン探索
- signalP v4 - シグナルペプチド切断領域の探索
いくつかはbrewでインストールできる。"brew list"で確認し、持ってないツールだけ導入。
brew install blast
brew install trinity
brew install sqlite
brew install TransDecoder
brew install HMMER
brew install RNAMMER
注;Trinityのutil/も必要だが、brewでは入らない。
GitHub - trinityrnaseq/trinityrnaseq: Trinity RNA-Seq de novo transcriptome assemblyからダウンロードしておく。必要になる。
Trinityと同じく、TransDecoderのutil/も必要になる。ダウンロードしておく。
Releases · TransDecoder/TransDecoder · GitHub
以下はTransDecoder.Predictの371行目についてのエラーメッセージが出る人だけ。
cd-hit-estにパスが通っていても変な場所から取ってこようとしてバグが起こる。応急措置で
my ($cd_hit_est_exec) = &check_program('cd-hit-est');
↓
my ($cd_hit_est_exec) = 'cd-hit-est';
に修正。
TMHMM、SignalPのインストールについては別にまとめています。こちらを参考にしてください。
TMHMM
SignalP
RNAMMER、HMMER、TransDecorderはbrewで入ります。
==============================================
データベースの準備
データのダウンロード
cd Trinotate-3.0.2/ #移動
admin/Build_Trinotate_Boilerplate_SQLite_db.pl Trinotate #スクリプト実行
fam-A.hmm.gz、uniprot_sprot.pep、Trinotate.sqliteがダウンロードされる。Trinotate.sqliteは予めUniprotのデータ(SwissProt、Uniref90)からTrinotateでデータベース化されたファイルである。自分で準備する場合、Trinotateのツールで作ることになる。
blastデータベースの準備
makeblastdb -in uniprot_sprot.pep -dbtype prot
pfamデータベースの準備
gunzip Pfam-A.hmm.gz #解凍
hmmpress Pfam-A.hmm #index作成
ここで一旦テストランを行う。
テストラン
Trinotateは複数のツールを走らせる上に、あまり堅牢な作りでないので少しのことでバグが出る。テストランを行ってエラーが出ないか確実に見極めて使う必要がある。
auto/testing/にTrinityでアセンブルされたデータが準備されている。
cd auto/testing/
ls -l #ファイルを確認
$ ls -l
total 8344
-rwxr-xr-x@ 1 user staff 620 3 3 06:13 cleanme.pl
-rwxr-xr-x@ 1 user staff 3929 3 3 06:13 conf.txt
-rwxr-xr-x@ 1 user staff 170122 3 3 06:13 mini_sprot.pep.gz
-rwxr-xr-x@ 1 user staff 63469 3 3 06:13 myTrinity.fasta.gene_to_trans_map.gz
-rwxr-xr-x@ 1 user staff 4019376 3 3 06:13 myTrinity.fasta.gz
-rwxr-xr-x@ 1 user staff 674 3 3 06:13 runMe.sh
myTrinity.fasta.gene_to_trans_map.gzとmyTrinity.fasta.gzを解凍する。
.pepファイルのblastデータベースを作る。
makeblastdb -in mini_sprot.pep -dbtype prot
confファイルを編集する。auto/conf.txtの13行目から25行目が編集する箇所である。
13-25行目を以下のように編集した。
----------------------------------------------------------------------------------------------------
# ** edit the progs and dbs section to point to your local resources.
# progs
TRANSDECODER_DIR=/home/uesaka/TransDecoder-3.0-2.1/
BLASTX_PROG=blastx
BLASTP_PROG=blastp
SIGNALP_PROG=~/signalp-4.1/signalp
TMHMM_PROG=/usr/local/bin/tmhmm
RNAMMER_TRANS_PROG=/home/uesaka/Trinotate-3.0.2/util/rnammer_support/RnammerTranscriptome.pl
RNAMMER=~/.linuxbrew/bin/rnammer
HMMSCAN_PROG=hmmscan
# dbs
SWISSPROT_PEP=mini_sprot.pep
PFAM_DB=/home/uesaka/Trinotate-3.0.2/Pfam-A.hmm
----------------------------------------------------------------------------------------------------
プログラムのパスによって変えてください。
テストラン
perl ../autoTrinotate.pl --Trinotate_sqlite ../../Trinotate.sqlite --transcripts myTrinity.fasta --gene_to_trans_map myTrinity.fasta.gene_to_trans_map --conf conf.txt --CPU 20
- --Trinotate_sqlite Trinotate.sqlite boilerplate database.
- --transcripts Transcripts.fasta.
- --gene_to_trans_map Gene-to-transcript mapping file.
- --conf Config file.
- --CPU Number of threads to use.
上記で指定しているファイルだが、--gene_to_trans_mapで指定しているGene-to-transcript mappingはtrinityのツールで作る。テストデータには最初から用意されている。--Trinotate_sqliteで指定しているTrinotate.sqliteは最初のデータベースの準備のところでダウンロードしたファイルである。中身はUniprotのデータ(SwissProt、Uniref90)をデータベース化したファイルになる。
Triniityでアセンブルされたcontigは15Mほどのサイズで、解析にはかなりの時間がかかる。終わるとExcelファイルで結果が出力される。

アノテーションできなかったセルや、ツールが正常に動作しなかったfeatureの列は"."で表示される 。その場合、公式の解説を真似してランできなかったツールだけ個別に走らせた方が早いかもしれない。
引用
The De Novo Transcriptome and Its Functional Annotation in the Seed Beetle Callosobruchus maculatus
Ahmed Sayadi, Elina Immonen, Helen Bayram, and Göran Arnqvist*
PLoS One. 2016; 11(7): e0158565. Published online 2016 Jul 21.
解析の流れ
http://informatics.fas.harvard.edu/trinotate-workflow-example-on-odyssey.html
https://omictools.com/transcriptome-annotation-category
関連