個々のタンパク質の構造的および機能的情報が蓄積されているが(ref.1–3)、それらの相互作用に関する知識は断片化されたままになっている。いくつかの相互作用は、たとえばlarge cellular machineries(ref.4〜6)の3次元再構築のコンテキストで非常によく文書化され理解されているが、他の相互作用は、これまでのところ遺伝的観察や統計的予測などの間接的な証拠によってのみ示唆されている。さらに、潜在的なタンパク質間相互作用の空間は、個々の分子の固有の分子機能の空間よりもはるかに大きく、またコンテキストに依存している。相互作用は、特定の細胞タイプまたは特定の生理学的条件に限定されるだけでなく、それらの特異性と強度も、義務的で高度に特異的で安定した結合から、よりつかの間の比較的非特異的な出会いまでさまざまである。純粋に機能的な観点から見ると、転写因子が別のタンパク質の発現と生産の調節に役立つ場合や、2つの酵素が拡散を介して特定の基質を交換する場合など、タンパク質はまったく触れずに特異的に相互作用することさえできる。
間違いなく、さまざまな形態のタンパク質間結合の共通点は情報の流れである。生物学的に意味のあるインターフェイスが進化し、細胞を介した情報の流れが可能になり、機能システムを実装するために不可欠である。したがって、すべてのタイプのタンパク質間相互作用を1つのフレームワークの下で収集して統合することが望ましい。これにより、疾患モジュールの識別(ref.7,8)からバイオマーカーの発見(ref.9–11)に至るまで、さまざまな分野のデータ分析パイプラインがサポートされ、手動での閲覧、アドホックな発見、アノテーションが可能になる。
タンパク質間相互作用は、多数のオンラインデータベース((ref.12,13)でレビュー)から収集できる。一次インタラクションデータベース(ref.3,15–18)は、ソース文献から直接実験的インタラクションの証拠に共同でアノテーションを付けており、IMExコンソーシアム(ref.19)を通じてその取り組みを調整している。メタデータのキュレーション、共通の名前空間の維持、オントロジーと標準の考案など、非常に価値のある追加サービスを提供する。タンパク質間相互作用情報の第2のソースは、計算予測の取り組みによって提供される。その一部は、専用のデータベースによってホストされている(ref.20,21)。最後に、データベースの3番目のクラスは、最も広い範囲でのタンパク質相互作用に特化しており、アノテーション付きパスウェイナリッジ、テキストマイニング結果、生物間移動またはその他の付属情報を含む、多くの場合、一次相互作用と予測相互作用の両方を統合する。 STRINGデータベース(「相互作用する遺伝子/タンパク質の検索ツール」)は、後者のクラスに属し、GeneMania(ref.22)、FunCoup(ref.23)、I2D(ref.24)、ConsensusPathDb(ref.25)、IMP(ref.26)およびHumanNet (ref.27)—そのほとんどは最近(ref.7)でレビューおよびベンチマークされた。
STRINGは初期の取り組みの1つであり(ref.28)、主に(i)高いカバレッジ、(ii)使いやすさ、(iii)一貫したスコアリングシステムを通じて差別化を図っている。現在最大数の生物(5090)とタンパク質(2460万)を特徴とし、非常に幅広く多様なベンチマークデータソースを持ち、オンラインで使用するための直感的で高速なビューアーを提供している。また、APIを介したプログラムによるアクセス、Cytoscapeアプリ(http://apps.cytoscape.org/apps/stringapp)を介したアクセス、個々の種のネットワークをカバーするダウンロードページなど、多くの追加のデータアクセスポイントも備えている。このWebサイトでは、ユーザーは検索と遺伝子セットにログオンして保存することができ、特定の相互作用の基礎となる証拠を検査するエビデンスビューアーが含まれている。また、ネットワーク強化統計や機能強化検出など、後者の2つの異なる概念フレームワークを使用して、入力/検索データに関する高レベルの情報をユーザーに提供する(論文の以下を参照)。 STRINGの多くの機能が利用可能になり、以前に説明されている(ref.28〜31)。現在、Webサイトには毎日約3500の異なるユーザーがアクセスしている。ホスティング施設は最近、複製され、商用の負荷分散装置の下に置かれ、安定性と容量が追加された。ユーザーは複数のタンパク質を同時に送信し、大規模なネットワークを視覚化できる。 Cytoscape stringAppは、数千のタンパク質のネットワークサイズも処理できる。 STRINGは、ゲノム、タンパク質、名前空間を、orthology(eggNOG(ref.32))、小分子(STITCH(ref.33))、タンパク質の存在量(PaxDB(ref.34))、組織発現(TISSUES)およびウイルス(Viruses.STRING(ref.36))、それぞれ専用のいくつかの姉妹プロジェクトと共有している。他のオンラインリソース(STRINGの最大のプライマリデータソースの1つであるIMExコンソーシアムを含む)とともに、STRINGデータベースは最近、欧州コアデータリソースのステータスを獲得した。
STRINGの基本的な相互作用単位は「機能的関連」、つまり特定の生物学的機能に共に寄与する2つのタンパク質間のリンクである(ref.38〜40)。このように2つのタンパク質を関連付けるには、物理的に相互作用する必要はない。代わりに、細胞内の機能的役割の少なくとも一部がオーバーラップすれば十分である。このオーバーラップ機能は、パスウェイマップまたは機能マップとして広く修飾するのに十分に特異的でなければならない(対照的に、オーバーラップ機能として単に「代謝」を共有することは不明確すぎる)。この定義により、同じパスウェイ内の阻害剤と活性化剤など、互いに拮抗するタンパク質でさえ機能的に関連付けることができる。 STRINGの機能的関連付けの望ましい特異性カットオフは、KEGGパスウェイマップのアノテーション粒度にほぼ対応する(ref.41)。これにより、タンパク質を相同性によって大部分グループ化するマップ(「ABCトランスポーター」など)が考慮から除外される。
STRINGデータベースの関連性の証拠はすべて、7つの独立した「チャネル」のいずれかに分類される。ゲノムコンテキスト情報に基づく3つの予測チャネル(以下を参照)、および(i)共発現、(ii)テキストマイニング、(iii)生化学/遺伝データ(「実験」)および(iv)以前にキュレートされたパスウェイおよびタンパク質複合体の知識(「データベース」)。ユーザーは、すべてのチャネルを個別にまたは組み合わせて無効にできる。各チャネルについて、基礎となる証拠を調べるためのviewersだけでなく、個別のインタラクションスコアも利用できる(論文図1)。一般に、STRINGのインタラクションスコアは、特定のインタラクションの強さまたは特異性を表すものではなく、利用可能なすべての証拠が与えられた場合に、0から1のスケールで関連が真であるというおおよその信頼を表すことを意味する。 STRINGのスコアは、両方のタンパク質パートナーに既に機能的にアノテーションが付けられている関連付けのサブセットを使用してベンチマークされる。このため、KEGGパスウェイマップ(ref.41)がゴールドスタンダードとして使用されているため、機能的関連付けの粒度も暗黙的に決定される。
(以下略)
Help center
https://string-db.org/cgi/help.pl?sessionId=RTrIIG790cxb
使い方
STRING: functional protein association networks にアクセスする。
Search
タンパク質名、名前、またはアミノ酸配列(任意の形式)を入力する。入力ワードは単独、複数どちらも対応している。
生物は一般名も使用できる。
結果
ネットワークで予測されるタンパク質グループの関連が示される。ノードはタンパク質、エッジは予測される機能的関連を表す。2つのノード間の物理的な距離には意味がない。
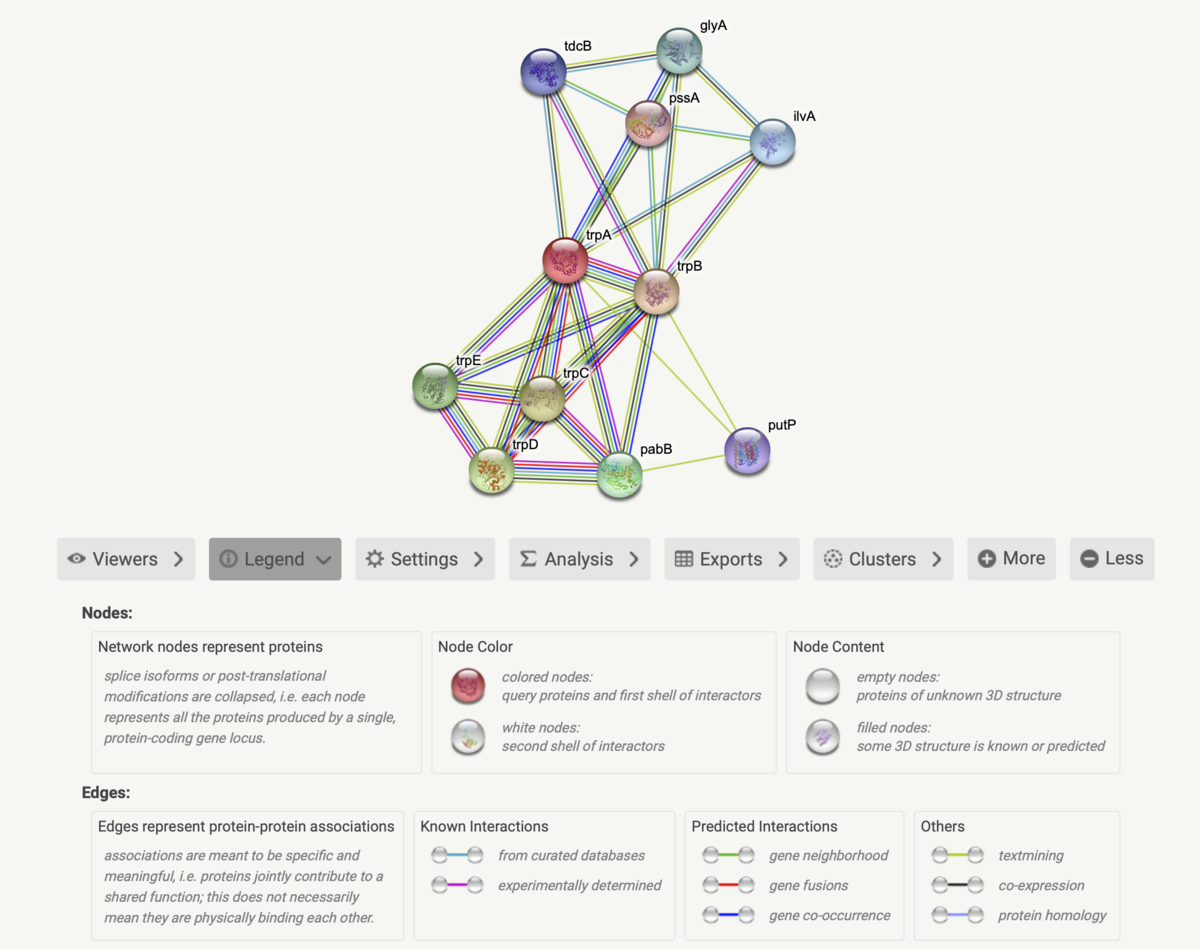
エッジは最大7色の線で描画される。以下のエビデンスを持っている。
- Red line - indicates the presence of fusion evidence
- Green line - neighborhood evidence
- Blue line - cooccurrence evidence
- Purple line - experimental evidence
- Yellow line - textmining evidence
- Light blue line - database evidence
- Black line - coexpression evidence.
ノードをクリックすると、タンパク質に関するいくつかの詳細が表示される。

エッジをクリックすると、詳細な証拠の内訳が表示される。

セッティングからconfidenceモードに変更。confidenceモードでは、線の太さは相互作用の信頼性予測の度合いを表す。

モードをNetworkから切り替える。エビデンスモードの7種類のエッジについて、それぞれ調べることができる。まずConserved Neighborhoodモード(上の図で緑色エッジがNeighborhoodエビデンス)に切り替える。Viewers => Neighborhoodを選択。
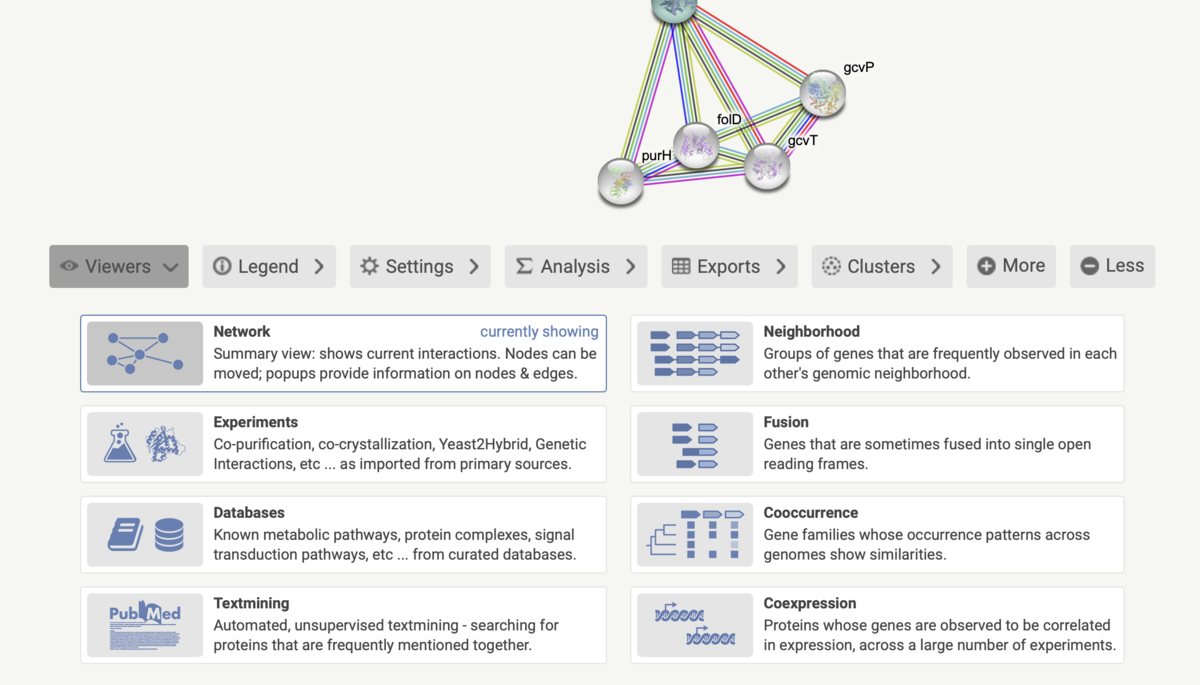
このNeighborhoodモードでは、(原核生物)ゲノムの近接したポジションに繰り返し発生する遺伝子が表示される。実行中に一緒に配置された遺伝子は、黒い線でリンクされる(最大許容遺伝子間距離は300塩基対)。 特定の種に対して複数の実行がある場合、これらは空白で区切られていることに注意する。 実行中に現在のスコアのしきい値を下回る他の遺伝子がある場合、それらは小さな白い三角形として描かれる。

それからLegendに切り替えることで、図のレジェンドを表示できるViewers右のLegendを選択。表示中の図に合わせてLegendが切り替わる。

Co-occurrenceモードでは種間でリンクされたタンパク質の有無がまとめられる。 タンパク質はページの上部に表示され、種名の系統樹は左側に配置される。それらの 種のタンパク質の存在は赤い四角でマークされ、ない場合は空白表示になる。 赤四角の色の強度は、相同なタンパク質の保存量を反映している。

Fusionモードでは、種ごとの個々の遺伝子融合イベントが表示される。 融合が起こる種は左にリストされている。 遺伝子は、Legendに従って色付けされている。

遺伝子をクリックすると、より詳細な情報が得られます。
Co-expressionモードでは、同じ種または他の種で共発現される遺伝子が表示される。 共発現は赤い四角で示される。

正方形の色が濃いほど、発現データの関連性スコアが高くなる。
他にDatabasesとText miningがある。Databasesモードには、キュレーションされたデータベースから収集された重要なタンパク質相互作用グループのリストが表示される。 Text miningモードには、文献から抽出された重要なタンパク質相互作用グループのリストが表示される。文献リンクとともに文献のタイトルと要約が表示される。
exportから図はPNG形式でダウンロードできる。
powerpointに貼ったところ。

引用
STRING v11: protein-protein association networks with increased coverage, supporting functional discovery in genome-wide experimental datasets.
Szklarczyk D1, Gable AL1, Lyon D1, Junge A2, Wyder S1, Huerta-Cepas J3, Simonovic M1, Doncheva NT2,4, Morris JH5, Bork P6,7,8,9, Jensen LJ2, Mering CV
Nucleic Acids Res. 2019 Jan 8;47(D1):D607-D613
参考
統合TV
STRINGの使い方 基本編
STRINGの使い方 応用編