2023/07/08 追記
構造予測手法が何百万もの一般に利用可能なタンパク質構造を生成しているため、これらのデータベースを検索することがボトルネックになりつつある。Foldseekは、タンパク質内の3次アミノ酸相互作用を構造アルファベット上の配列として記述することにより、クエリタンパク質の構造をデータベースと整合させる。Foldseekは、Dali、TM-align、CEの感度のそれぞれ86%、88%、133%で、計算時間を4~5桁短縮する。
https://github.com/steineggerlab/foldseek/wiki
Tutorial Video
インストール
依存
FoldseekはLinuxとmacOSでテストされている。Windowsのプレビュー版も提供されている。OpenMPを通じてマルチコアシステムを活用する。最適なパフォーマンスを得るにはAVX2命令セットをサポートするシステムが必要だが、SSE4.1やSSE2を搭載した古いシステムもサポートしている。PPC64LEとARM64プロセッサ・アーキテクチャもサポートしている(wiki参照)。
# Linux AVX2 build (check using: cat /proc/cpuinfo | grep avx2)
wget https://mmseqs.com/foldseek/foldseek-linux-avx2.tar.gz; tar xvzf foldseek-linux-avx2.tar.gz; export PATH=$(pwd)/foldseek/bin/:$PATH
# Linux SSE4.1 build (check using: cat /proc/cpuinfo | grep sse4_1)
wget https://mmseqs.com/foldseek/foldseek-linux-sse41.tar.gz; tar xvzf foldseek-linux-sse41.tar.gz; export PATH=$(pwd)/foldseek/bin/:$PATH
# MacOS
wget https://mmseqs.com/foldseek/foldseek-osx-universal.tar.gz; tar xvzf foldseek-osx-universal.tar.gz; export PATH=$(pwd)/foldseek/bin/:$PATH
#Other precompiled binaries
# Conda installer (Linux and macOS)
mamba install -c conda-forge -c bioconda foldseek -y
$ foldseek
Foldseek enables fast and sensitive comparisons of large structure sets. It reaches sensitivities similar to state-of-the-art structural aligners while being at least 20,000 times faster.
Please cite:
van Kempen, M., Kim, S.S., Tumescheit, C., Mirdita, M., Lee, J., Gilchrist, C.L.M., Söding, J., and Steinegger, M. Fast and accurate protein structure search with Foldseek. Nature Biotechnology, doi:10.1038/s41587-023-01773-0 (2023)
foldseek Version: bc669d4b9d90901c724f5d9bfac3e7cefde2ef15
© Michel van Kempen, Stephanie Kim, Charlotte Tumescheit, Milot Mirdita, Jeongjae Lee, Cameron L. M. Gilchrist, Johannes Söding, Martin Steinegger
usage: foldseek <command> [<args>]
Easy workflows for plain text input/output
easy-search Structual search
easy-cluster Slower, sensitive clustering
easy-rbh Find reciprocal best hit
Main workflows for database input/output
createdb Convert PDB/mmCIF/tar[.gz]/DB files to a db
search Sensitive homology search
rbh Reciprocal best hit search
cluster Slower, sensitive clustering
Input database creation
databases List and download databases
createindex Store precomputed index on disk to reduce search overhead
createclusearchdb Build a searchable cluster database allowing for faster searches
Format conversion for downstream processing
convertalis Convert alignment DB to BLAST-tab, SAM or custom format
convert2pdb Convert a foldseek structure db to a multi model PDB file
An extended list of all modules can be obtained by calling 'foldseek -h'.
$ foldseek databases
usage: foldseek databases <name> <o:sequenceDB> <tmpDir> [options]
Name Type Taxonomy Url
- Alphafold/UniProt Aminoacid yes https://alphafold.ebi.ac.uk/
- Alphafold/UniProt50 Aminoacid yes https://alphafold.ebi.ac.uk/
- Alphafold/Proteome Aminoacid yes https://alphafold.ebi.ac.uk/
- Alphafold/Swiss-Prot Aminoacid yes https://alphafold.ebi.ac.uk/
- ESMAtlas30 Aminoacid - https://esmatlas.com
- PDB Aminoacid yes https://www.rcsb.org
options:
--tsv BOOL Return output in TSV format [0]
--compressed INT Write compressed output [0]
--threads INT Number of CPU-cores used (all by default) [56]
-v INT Verbosity level: 0: quiet, 1: +errors, 2: +warnings, 3: +info [3]
references:
- Mirdita M, Steinegger M, Breitwieser F, Soding J, Levy Karin E: Fast and sensitive taxonomic assignment to metagenomic contigs. Bioinformatics, btab184 (2021)
- van Kempen, M., Kim, S.S., Tumescheit, C., Mirdita, M., Lee, J., Gilchrist, C.L.M., Söding, J., and Steinegger, M. Fast and accurate protein structure search with Foldseek. Nature Biotechnology, doi:10.1038/s41587-023-01773-0 (2023)
Show an extended list of options by calling 'foldseek databases -h'.
> foldseek easy-search -h
usage: foldseek easy-search <i:PDB|mmCIF[.gz]> ... <i:PDB|mmCIF[.gz]>|<i:stdin> <i:targetFastaFile[.gz]>|<i:targetDB> <o:alignmentFile> <tmpDir> [options]
By Martin Steinegger <martin.steinegger@snu.ac.kr>
options: prefilter:
--comp-bias-corr INT Correct for locally biased amino acid composition (range 0-1) [1]
--comp-bias-corr-scale FLOAT Correct for locally biased amino acid composition (range 0-1) [1.000]
--seed-sub-mat TWIN Substitution matrix file for k-mer generation [aa:3di.out,nucl:3di.out]
-s FLOAT Sensitivity: 1.0 faster; 4.0 fast; 7.5 sensitive [9.500]
-k INT k-mer length (0: automatically set to optimum) [6]
--k-score TWIN k-mer threshold for generating similar k-mer lists [seq:2147483647,prof:2147483647]
--max-seqs INT Maximum results per query sequence allowed to pass the prefilter (affects sensitivity) [1000]
--split INT Split input into N equally distributed chunks. 0: set the best split automatically [0]
--split-mode INT 0: split target db; 1: split query db; 2: auto, depending on main memory [2]
--split-memory-limit BYTE Set max memory per split. E.g. 800B, 5K, 10M, 1G. Default (0) to all available system memory [0]
--diag-score BOOL Use ungapped diagonal scoring during prefilter [1]
--exact-kmer-matching INT Extract only exact k-mers for matching (range 0-1) [0]
--mask INT Mask sequences in k-mer stage: 0: w/o low complexity masking, 1: with low complexity masking [0]
--mask-prob FLOAT Mask sequences is probablity is above threshold [1.000]
--mask-lower-case INT Lowercase letters will be excluded from k-mer search 0: include region, 1: exclude region [1]
--min-ungapped-score INT Accept only matches with ungapped alignment score above threshold [30]
--spaced-kmer-mode INT 0: use consecutive positions in k-mers; 1: use spaced k-mers [1]
--spaced-kmer-pattern STR User-specified spaced k-mer pattern
--local-tmp STR Path where some of the temporary files will be created
--exhaustive-search BOOL Turns on an exhaustive all vs all search by by passing the prefilter step [0]
align:
--min-seq-id FLOAT List matches above this sequence identity (for clustering) (range 0.0-1.0) [0.000]
-c FLOAT List matches above this fraction of aligned (covered) residues (see --cov-mode) [0.000]
--cov-mode INT 0: coverage of query and target
1: coverage of target
2: coverage of query
3: target seq. length has to be at least x% of query length
4: query seq. length has to be at least x% of target length
5: short seq. needs to be at least x% of the other seq. length [0]
--max-rejected INT Maximum rejected alignments before alignment calculation for a query is stopped [2147483647]
--max-accept INT Maximum accepted alignments before alignment calculation for a query is stopped [2147483647]
-a BOOL Add backtrace string (convert to alignments with mmseqs convertalis module) [0]
--sort-by-structure-bits INT sort by bits*sqrt(alnlddt*alntmscore) [1]
--alignment-mode INT How to compute the alignment:
0: automatic
1: only score and end_pos
2: also start_pos and cov
3: also seq.id [3]
--alignment-output-mode INT How to compute the alignment:
0: automatic
1: only score and end_pos
2: also start_pos and cov
3: also seq.id
4: only ungapped alignment
5: score only (output) cluster format [0]
-e DOUBLE List matches below this E-value (range 0.0-inf) [1.000E+01]
--min-aln-len INT Minimum alignment length (range 0-INT_MAX) [0]
--seq-id-mode INT 0: alignment length 1: shorter, 2: longer sequence [0]
--alt-ali INT Show up to this many alternative alignments [0]
--gap-open TWIN Gap open cost [aa:10,nucl:10]
--gap-extend TWIN Gap extension cost [aa:1,nucl:1]
profile:
--num-iterations INT Number of iterative profile search iterations [1]
misc:
--tmscore-threshold FLOAT accept alignments with a tmsore > thr [0.0,1.0] [0.000]
--tmalign-hit-order INT order hits by 0: (qTM+tTM)/2, 1: qTM, 2: tTM, 3: min(qTM,tTM) 4: max(qTM,tTM) [0]
--tmalign-fast INT turn on fast search in TM-align [1]
--lddt-threshold FLOAT accept alignments with a lddt > thr [0.0,1.0] [0.000]
--prefilter-mode INT prefilter mode: 0: kmer/ungapped 1: ungapped, 2: nofilter [0]
--alignment-type INT How to compute the alignment:
0: 3di alignment
1: TM alignment
2: 3Di+AA [2]
--cluster-search INT first find representative then align all cluster members [0]
--mask-bfactor-threshold FLOAT mask residues for seeding if b-factor < thr [0,100] [0.000]
--file-include STR Include file names based on this regex [.*]
--file-exclude STR Exclude file names based on this regex [^$]
--format-mode INT Output format:
0: BLAST-TAB
1: SAM
2: BLAST-TAB + query/db length
3: Pretty HTML
4: BLAST-TAB + column headers
5: Calpha only PDB super-posed to query
BLAST-TAB (0) and BLAST-TAB + column headers (4)support custom output formats (--format-output)
(5) Superposed PDB files (Calpha only) [0]
--format-output STR Choose comma separated list of output columns from: query,target,evalue,gapopen,pident,fident,nident,qstart,qend,qlen
tstart,tend,tlen,alnlen,raw,bits,cigar,qseq,tseq,qheader,theader,qaln,taln,mismatch,qcov,tcov
qset,qsetid,tset,tsetid,taxid,taxname,taxlineage,
lddt,lddtfull,qca,tca,t,u,qtmscore,ttmscore,alntmscore,rmsd,prob
[query,target,fident,alnlen,mismatch,gapopen,qstart,qend,tstart,tend,evalue,bits]
--greedy-best-hits BOOL Choose the best hits greedily to cover the query [0]
common:
--db-load-mode INT Database preload mode 0: auto, 1: fread, 2: mmap, 3: mmap+touch [0]
--threads INT Number of CPU-cores used (all by default) [10]
-v INT Verbosity level: 0: quiet, 1: +errors, 2: +warnings, 3: +info [3]
--sub-mat TWIN Substitution matrix file [aa:3di.out,nucl:3di.out]
--max-seq-len INT Maximum sequence length [65535]
--compressed INT Write compressed output [0]
--remove-tmp-files BOOL Delete temporary files [1]
--mpi-runner STR Use MPI on compute cluster with this MPI command (e.g. "mpirun -np 42")
--force-reuse BOOL Reuse tmp filse in tmp/latest folder ignoring parameters and version changes [0]
expert:
--zdrop INT Maximal allowed difference between score values before alignment is truncated (nucleotide alignment only) [40]
--taxon-list STR Taxonomy ID, possibly multiple values separated by ','
--chain-name-mode INT Add chain to name:
0: auto
1: always add
[0]
--write-mapping INT write _mapping file containing mapping from internal id to taxonomic identifier [0]
--coord-store-mode INT Coordinate storage mode:
1: C-alpha as float
2: C-alpha as difference (uint16_t) [2]
--write-lookup INT write .lookup file containing mapping from internal id, fasta id and file number [1]
--db-output BOOL Return a result DB instead of a text file [0]
examples:
# Search a single/multiple PDB file against a set of PDB files
foldseek easy-search examples/d1asha_ examples/ result.m8 tmp
# Format output differently
foldseek easy-search examples/d1asha_ examples/ result.m8 tmp --format-output query,target,qstart,tstart,cigar
# Align with TMalign (global)
foldseek easy-search examples/d1asha_ examples/ result.m8 tmp --alignment-type 1
# Skip prefilter and perform an exhaustive alignment (slower but more sensitive)
foldseek easy-search examples/d1asha_ examples/ result.m8 tmp --exhaustive-search 1
references:
- van Kempen, M., Kim, S.S., Tumescheit, C., Mirdita, M., Lee, J., Gilchrist, C.L.M., Söding, J., and Steinegger, M. Fast and accurate protein structure search with Foldseek. Nature Biotechnology, doi:10.1038/s41587-023-01773-0 (2023)
> foldseek easy-cluster -h
$ foldseek easy-cluster -h
usage: foldseek easy-cluster <i:PDB|mmCIF[.gz]> ... <i:PDB|mmCIF[.gz]> <o:clusterPrefix> <tmpDir> [options]
By Martin Steinegger <martin.steinegger@snu.ac.kr>
options: prefilter:
--seed-sub-mat TWIN Substitution matrix file for k-mer generation [aa:3di.out,nucl:3di.out]
-s FLOAT Sensitivity: 1.0 faster; 4.0 fast; 7.5 sensitive [4.000]
-k INT k-mer length (0: automatically set to optimum) [0]
--k-score TWIN k-mer threshold for generating similar k-mer lists [seq:2147483647,prof:2147483647]
--max-seqs INT Maximum results per query sequence allowed to pass the prefilter (affects sensitivity) [300]
--split INT Split input into N equally distributed chunks. 0: set the best split automatically [0]
--split-mode INT 0: split target db; 1: split query db; 2: auto, depending on main memory [2]
--split-memory-limit BYTE Set max memory per split. E.g. 800B, 5K, 10M, 1G. Default (0) to all available system memory [0]
--comp-bias-corr INT Correct for locally biased amino acid composition (range 0-1) [1]
--comp-bias-corr-scale FLOAT Correct for locally biased amino acid composition (range 0-1) [1.000]
--diag-score BOOL Use ungapped diagonal scoring during prefilter [1]
--exact-kmer-matching INT Extract only exact k-mers for matching (range 0-1) [0]
--mask INT Mask sequences in k-mer stage: 0: w/o low complexity masking, 1: with low complexity masking [1]
--mask-prob FLOAT Mask sequences is probablity is above threshold [0.900]
--mask-lower-case INT Lowercase letters will be excluded from k-mer search 0: include region, 1: exclude region [1]
--min-ungapped-score INT Accept only matches with ungapped alignment score above threshold [30]
--spaced-kmer-mode INT 0: use consecutive positions in k-mers; 1: use spaced k-mers [1]
--spaced-kmer-pattern STR User-specified spaced k-mer pattern
--local-tmp STR Path where some of the temporary files will be created
align:
-c FLOAT List matches above this fraction of aligned (covered) residues (see --cov-mode) [0.000]
--cov-mode INT 0: coverage of query and target
1: coverage of target
2: coverage of query
3: target seq. length has to be at least x% of query length
4: query seq. length has to be at least x% of target length
5: short seq. needs to be at least x% of the other seq. length [0]
--sort-by-structure-bits INT sort by bits*sqrt(alnlddt*alntmscore) [1]
-a BOOL Add backtrace string (convert to alignments with mmseqs convertalis module) [0]
--alignment-mode INT How to compute the alignment:
0: automatic
1: only score and end_pos
2: also start_pos and cov
3: also seq.id [0]
--alignment-output-mode INT How to compute the alignment:
0: automatic
1: only score and end_pos
2: also start_pos and cov
3: also seq.id
4: only ungapped alignment
5: score only (output) cluster format [0]
-e DOUBLE List matches below this E-value (range 0.0-inf) [1.000E+01]
--min-seq-id FLOAT List matches above this sequence identity (for clustering) (range 0.0-1.0) [0.000]
--min-aln-len INT Minimum alignment length (range 0-INT_MAX) [0]
--seq-id-mode INT 0: alignment length 1: shorter, 2: longer sequence [0]
--alt-ali INT Show up to this many alternative alignments [0]
--max-rejected INT Maximum rejected alignments before alignment calculation for a query is stopped [2147483647]
--max-accept INT Maximum accepted alignments before alignment calculation for a query is stopped [2147483647]
--gap-open TWIN Gap open cost [aa:10,nucl:10]
--gap-extend TWIN Gap extension cost [aa:1,nucl:1]
clust:
--cluster-mode INT 0: Set-Cover (greedy)
1: Connected component (BLASTclust)
2,3: Greedy clustering by sequence length (CDHIT) [0]
--max-iterations INT Maximum depth of breadth first search in connected component clustering [1000]
--similarity-type INT Type of score used for clustering. 1: alignment score 2: sequence identity [2]
--single-step-clustering BOOL Switch from cascaded to simple clustering workflow [0]
--cluster-steps INT Cascaded clustering steps from 1 to -s [3]
--cluster-reassign BOOL Cascaded clustering can cluster sequence that do not fulfill the clustering criteria.
Cluster reassignment corrects these errors [0]
kmermatcher:
--weights STR Weights used for cluster priorization
--cluster-weight-threshold FLOAT Weight threshold used for cluster priorization [0.900]
--kmer-per-seq INT k-mers per sequence [21]
--kmer-per-seq-scale TWIN Scale k-mer per sequence based on sequence length as kmer-per-seq val + scale x seqlen [aa:0.000,nucl:0.200]
--adjust-kmer-len BOOL Adjust k-mer length based on specificity (only for nucleotides) [0]
--hash-shift INT Shift k-mer hash initialization [67]
--include-only-extendable BOOL Include only extendable [0]
--ignore-multi-kmer BOOL Skip k-mers occurring multiple times (>=2) [0]
misc:
--tmscore-threshold FLOAT accept alignments with a tmsore > thr [0.0,1.0] [0.000]
--lddt-threshold FLOAT accept alignments with a lddt > thr [0.0,1.0] [0.000]
--rescore-mode INT Rescore diagonals with:
0: Hamming distance
1: local alignment (score only)
2: local alignment
3: global alignment
4: longest alignment fulfilling window quality criterion [0]
--tmalign-hit-order INT order hits by 0: (qTM+tTM)/2, 1: qTM, 2: tTM, 3: min(qTM,tTM) 4: max(qTM,tTM) [0]
--tmalign-fast INT turn on fast search in TM-align [1]
--mask-bfactor-threshold FLOAT mask residues for seeding if b-factor < thr [0,100] [0.000]
--file-include STR Include file names based on this regex [.*]
--file-exclude STR Exclude file names based on this regex [^$]
common:
--sub-mat TWIN Substitution matrix file [aa:3di.out,nucl:3di.out]
--max-seq-len INT Maximum sequence length [65535]
--db-load-mode INT Database preload mode 0: auto, 1: fread, 2: mmap, 3: mmap+touch [0]
--threads INT Number of CPU-cores used (all by default) [10]
--compressed INT Write compressed output [0]
-v INT Verbosity level: 0: quiet, 1: +errors, 2: +warnings, 3: +info [3]
--remove-tmp-files BOOL Delete temporary files [1]
--force-reuse BOOL Reuse tmp filse in tmp/latest folder ignoring parameters and version changes [0]
--mpi-runner STR Use MPI on compute cluster with this MPI command (e.g. "mpirun -np 42")
expert:
--taxon-list STR Taxonomy ID, possibly multiple values separated by ',' []
--zdrop INT Maximal allowed difference between score values before alignment is truncated (nucleotide alignment only) [40]
--filter-hits BOOL Filter hits by seq.id. and coverage [0]
--sort-results INT Sort results: 0: no sorting, 1: sort by E-value (Alignment) or seq.id. (Hamming) [0]
--chain-name-mode INT Add chain to name:
0: auto
1: always add
[0]
--write-mapping INT write _mapping file containing mapping from internal id to taxonomic identifier [0]
--coord-store-mode INT Coordinate storage mode:
1: C-alpha as float
2: C-alpha as difference (uint16_t) [2]
--write-lookup INT write .lookup file containing mapping from internal id, fasta id and file number [1]
examples:
foldseek easy-cluster examples/ result tmp
# Cluster output
# - result_rep_seq.fasta: Representatives
# - result_all_seq.fasta: FASTA-like per cluster
# - result_cluster.tsv: Adjacency list
# Important parameter: --min-seq-id, --cov-mode and -c
# --cov-mode
# 0 1 2
# Q: MAVGTACRPA 60% IGN 60%
# T: -AVGTAC--- 60% 100% IGN
# -c 0.7 - + -
# -c 0.6 + + +
references:
- van Kempen, M., Kim, S.S., Tumescheit, C., Mirdita, M., Lee, J., Gilchrist, C.L.M., Söding, J., and Steinegger, M. Fast and accurate protein structure search with Foldseek. Nature Biotechnology, doi:10.1038/s41587-023-01773-0 (2023)
データベース
いくつか利用できる。画像はレポジトリより。
#PDBをpdbとしてカレントディレクトリにダウンロードする。
foldseek databases PDB pdb ./
テストラン
easy-search
easy-searchモジュールは、PDB/mmCIFフォーマット(フラットまたはgzip)でフォーマットされた単一または複数のクエリ構造を、ターゲットデータベース、フォルダ、または単一のタンパク質構造に対して検索する。
git clone https://github.com/steineggerlab/foldseek.git
foldseek easy-search foldseek/example/d1asha_ swissprotDB ./output.txt /tmp/
デフォルトでは、アラインメント情報をタブ区切りのファイルとして出力する。
output.txt

デフォルトのフィールドは、query,target,fident,alnlen,mismatch,gapopen,qstart,qend,tstart,tend,evalue,bits。クエリとターゲット識別子、α座標、アラインメントのTMスコア、クエリ長もしくは ターゲット長で正規化したTMスコア、平均LDDT、そのほか。詳細はレポジトリで説明されている。
"--format-output" オプションでフィールドをカスタマイズできる。
foldseek easy-search foldseek/example/d1asha_ swissprotDB ./output.txt /tmp/ --format-mode 3
結果をHTML形式で出力するには"--format-mode 3"を指定する。
foldseek easy-search foldseek/example/d1asha_ swissprotDB ./output.txt /tmp/ --format-output "query,target,qaln,taln"
easy-cluster (ref.2)
easy-clusterアルゴリズムは、構造アライメントを用いて代表的なタンパク質に構造を割り当てることにより、構造クラスタリングを行う。フラットファイルとgzipファイルの両方をサポートし、PDBまたはmmCIF形式の入力を受け付ける(レポジトリより)。
foldseek easy-cluster example/ outprefix tmp -c 0.9
- -c List matches above this fraction of aligned (covered) residues (see --cov-mode) [0.000]
出力

res_cluster.tsv

以下の接頭辞を持つ3つの出力ファイルを生成する:(1) _clu.tsv, (2) _repseq.fasta, (3) _allseq.fasta。最初のファイル(1)は代表配列からメンバー配列へのマッピングを記述したタブ区切りのファイル(上の画像2枚目)で、2番目のファイル(2)は代表配列のみを含み、3番目のファイル(3)は全てのクラスターメンバー配列を含む(レポジトリより)。exampleでは3つのクラスタに分類された。
Foldseekはweb上でも使用できる。
(一部古い情報があります)
さらにFoldseek clusterもweb上で利用できる。
https://cluster.foldseek.com/にアクセスする。
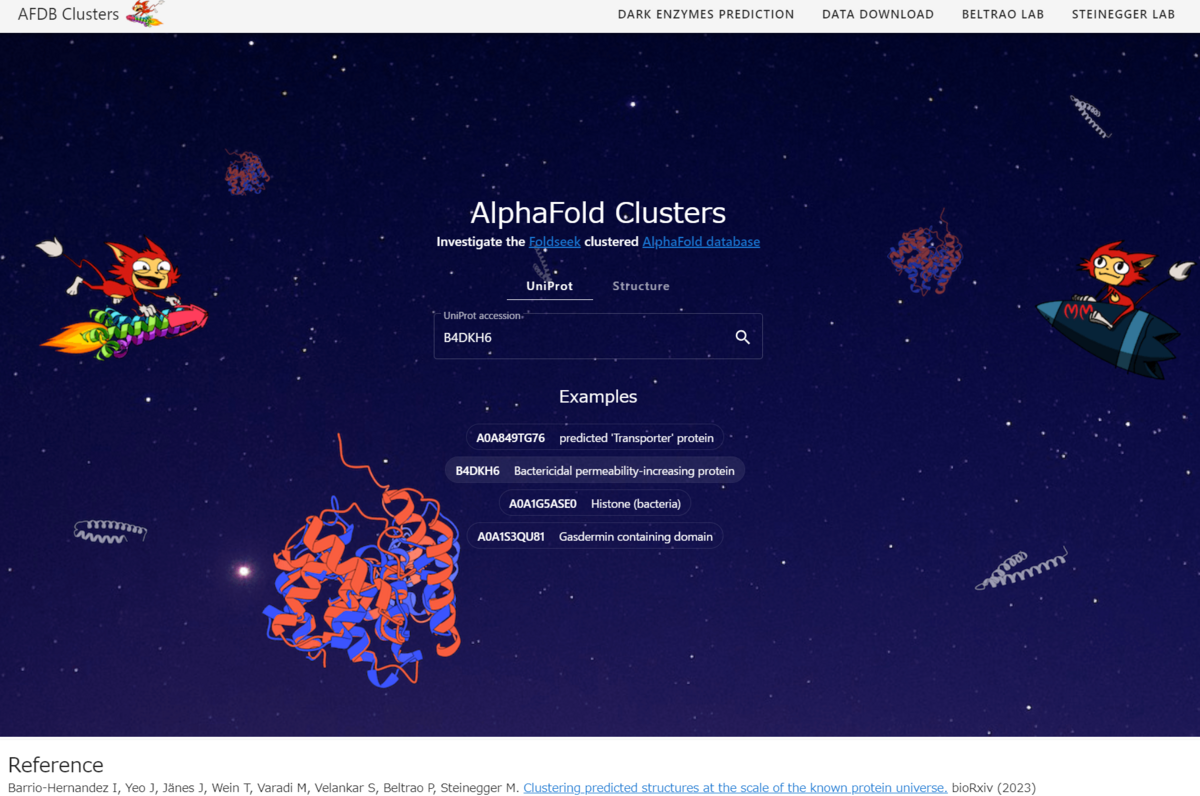
あまり説明がないが、利用できるのは、Uniprot Alphafoldをfoldseekでクラスター化した結果だと思われる。
Uniprot accession IDを指定する。不明ならBLASTサーチして全長が100%マッチする配列のIDを取り出す。もしくは

PDBファイルも使用できる。

事前計算された結果を読み込むので、結果は直ぐに表示される。
出力例

そのクラスタに含まれるタンパク質構造数、平均長、平均pLDDT(構造の一致度を示す0~100の値)などが示される。

そのクラスタのメンバーのタンパク質をどのような分類の生物が持っているかも示される。

図の分類をクリックすると、その分類でのリストのみ表に提示される。

その下には、同一のクラスタではないが類似したクラスタの代表に関するリスト”Similar clusters”も表示される。タンパク質によってはこちらのほうに目的のクラスタがある可能性もあるので、代表のアノテーションをよく見る必要がある。
結果のリストをダウンロードしたいが、その機能は提供されていないようだった。DATA Downloadからクラスタ情報をダウンロードしてパースする必要があると思われる。
- 例では比較的均一なアノテーションが得られているが、構造によっては、必ずしも統一的なアノテーションではないクラスタもあった。アノテーションが間違っているためにこのような結果が生じている可能性もあるが、アノテーションが正しいとするなら、タンパク質の折り畳みはよく似ているが、反応を触媒する構造の微小な違いは区別が難しいのかもしれない。そのクラスタの構造を入手し、より厳しい閾値で再度クラスタリングすれば分離できるのかもしれない(未テスト)。
引用
1
Fast and accurate protein structure search with Foldseek
Michel van Kempen, Stephanie S. Kim, Charlotte Tumescheit, Milot Mirdita, Jeongjae Lee, Cameron L. M. Gilchrist, Johannes Söding & Martin Steinegger
Nature Biotechnology (2023)
2
Clustering predicted structures at the scale of the known protein universe
Inigo Barrio Hernandez, Jingi Yeo, Jürgen Jänes, Tanita Wein, Mihaly Varadi, Sameer Velankar, Pedro Beltrao, Martin Steinegger
bioRxiv, Posted March 10, 2023.
"構造アライメントに基づくクラスタリングアルゴリズムFoldseek clusterを開発した。この手法を用いて、AFDBに含まれる全ての構造をクラスタリングし、227万個の非シングルトン構造クラスタを同定した。そのうち31%は注釈がなく、新規構造である可能性が高い。アノテーションのないクラスターは、AFDBに含まれる全タンパク質のわずか4%しか代表者がいない傾向がある。進化解析によると、ほとんどのクラスターは古い起源を持つが、4%は種特異的で、質の低い予測やデノボ遺伝子誕生の例であると考えられる。"
Foldseekとは直接関係ないコメント
Martin氏がTwitterで言及されていますが、xTrimoPGLMという新しいタンパク質言語モデルが発表されています(リンク)。十分にトレーニング後にいくつかベンチマークを行っていますが、その中には構造推定の精度に関するものもあって、AF2など有名な構造推論ツールを上回っています。驚くべきは学習量の多さで、1,000億パラメータ、1兆訓練トークンとなっています。論文中には"2023年1月18日から6月30日の間に、96台のNVIDIA DGX-A100 (8×40G) GPUノードのクラスタ上で1兆トークン(処理単位)を超える処理が行われた"
このように書かれており、膨大な計算リソースが使われ、現在もさらに継続中のようです。NVIDIA DGX-A100は、GPUユニット部分にNVIDIA A100 TensorコアGPU8台を含むAI向けサーバーです。購入すると保守無しでも1千万以上するようですが、これが96台も(つまり単純計算では768台のA100)使われているのは凄まじいの一言です。これまでの最大はESM2のESM2-15Bが最大(=150億パラメータ)だったと思いますが、1,000億(100B)というのは遥かに上回っています。
言語モデルの性能は学習時間、データ量、モデルサイズに関するべき乗則に従い改善されるスケーリング則があることが知られているそうです(参考文献)。実際にxTrimoPGLMの論文図1では、pre-trainingを指数関数的に増加させると性能がリニアに増す事が示されています。比較されているのはESM2-15Bなどです。
個人的に気になるのは、現在のモデルはオープンソースとして公開されていない点です。これはプレプリントだからなのか、GPT-4のような安全上の理由(=悪用されないか)、もしくは他の理由があるのでしょうか。学習元は世界中の研究者の成果そのものでしょうから、それが意図的に公開されないのは違和感を覚えます(主観的な意見です)。
