遺伝子間の進化的関係を明らかにすることは、比較生物学研究の基本である。ここでは、SHOOTを紹介する。SHOOTは、ユーザからのクエリー配列を系統樹のデータベースと照合し、クエリー配列が正しく配置された系統樹を返す。SHOOTはBLAST検索に匹敵する速度でこの解析を行うことを示す。また、SHOOTによる系統樹の配置は従来の樹木推論と同程度の精度を持ち、高い精度でオルソログを同定できることを実証した。SHOOTは、新規クエリ配列の系統解析のための高速かつ高精度なツールである。オンラインで www.shoot.bio から入手できる。
従来の配列オーソロジー解析は、まず類似性を持つ遺伝子配列群を集め、この群に対して系統樹推論を行い、それらの遺伝子間の関係を推論するという手順がとられていた。SHOOTアルゴリズムは、このような系統樹解析を2段階のアプローチによるリアルタイム検索として実現するために設計された。第1段階はSHOOT系統データベースの先行構築、第2段階はクエリー配列に対するSHOOT検索の実行である(論文図1)。データベース作成段階では、相同性グループの推論、多重配列アラインメント、系統樹推論、相同性グループプロファイリングなどの複数の自動化ステップを含む(材料と方法)。このため、データベース検索前に、データベース内の全遺伝子間の系統的関係が既に確立されている。SHOOT検索では、アラインメントと系統樹がすでに計算されていることを利用して、クエリー遺伝子を事前に計算された遺伝子樹の中に配置するための正確な系統学的手法を、余分な計算をほとんど必要とせずに行うことができる。
FAQ
SHOOT, the phylogenetic sequence search engine, has been published!
— David Emms (@David__Emms) March 29, 2022
SHOOT is like BLAST, but instead of a list of similar sequences you get a maximum likelihood phylogenetic tree for your query gene sequence.
Try it👉 https://t.co/65j4Kf47oX
Article👉 https://t.co/V3hKU2gSS0
HPより
SHOOT.bioは系統樹検索エンジンです。
遺伝子ファミリーのデータベースに対してクエリ配列を検索し、クエリ配列を接ぎ木(grafted)した系統樹を提供します。SHOOTを使うと、以下のようなことができます。
- 他の生物における類似配列の検索(BLASTと同様)。
- クエリ配列とその近縁種の系統樹を作成する。
- 他の生物種におけるその遺伝子のオルソログを検索する。
- その遺伝子の進化史のマップを作成する。
- クエリ遺伝子の遺伝子ファミリーを検索する。
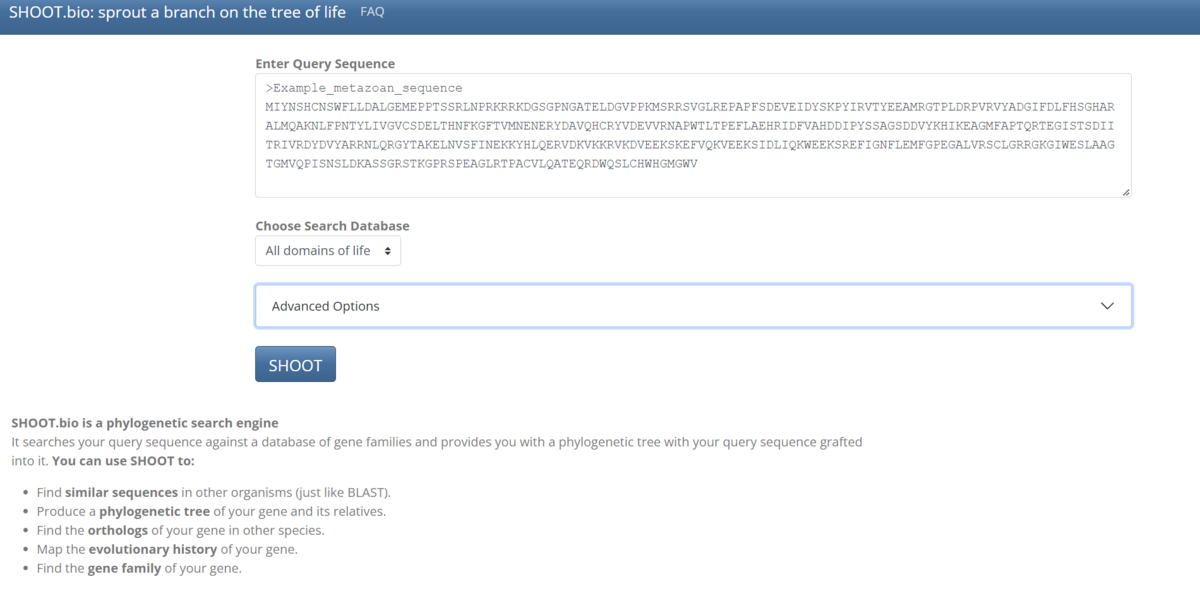
https://shoot.bio/にアクセスする。
デフォルトではExample_metazoan_sequenceというアミノ酸配列がセットされている。
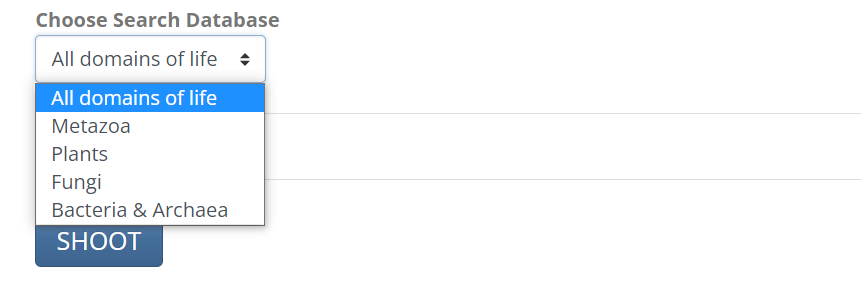
Domainを選択する。デフォルトではAll domains of lifeにになっている。
Advanced options
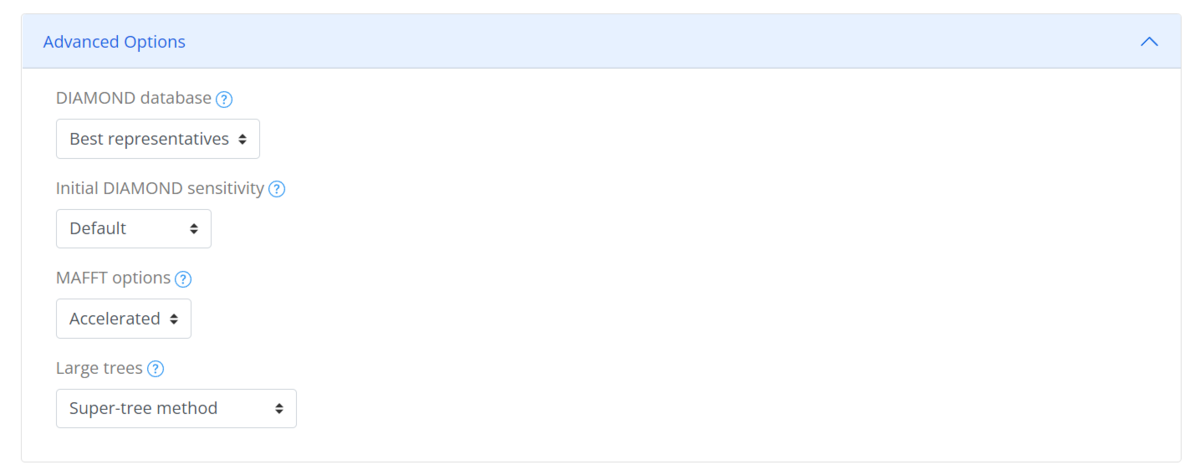
デフォルトのデータベース(相同グループの最良の代表者)には、各グループの配列多様性を最もよく表すようにk-meansクラスタリングで選択された配列が含まれている。ほとんどの場合、これはうまく機能し、クエリ遺伝子に対する正しい相同グループを容易に特定することができる。また、全相同性グループの全配列を含むデータベースを使用することも可能で、こちらの方がうまくいく場合もある(helpより)。

SHOOTのデフォルトでは、DIAMONDの通常感度での検索が実行される(DIAMOND紹介)。ヒットしない時に感度を上げるには、より低速だが Ultra-sensitive サーチに変更できる。

MAFFTは高いパフォーマンスを発揮する”retree 1 --maxiterate 0 --nofft"オプションで実行される。MAFFT をデフォルトのオプションで実行するように選択することもできる。

最大の遺伝子ツリー(>2500 gene)は、系統樹の配置に非常に時間がかかるため、サブツリーに分割されている。この場合、データベース内の最大40本のツリーが対象となる。このような大きなツリーに対して、DIAMONDはクエリー配列をサブツリーに割り当て、系統発生的手法(MSAとツリー推論)を使ってサブツリー内の正しい位置に遺伝子を配置する。このサブツリーを元のスーパーツリーに接ぎ木し、ユーザーに返すことで、より低い計算コストで完全な遺伝子ファミリーの中の遺伝子を表示することができる。(helpより)。

出力例
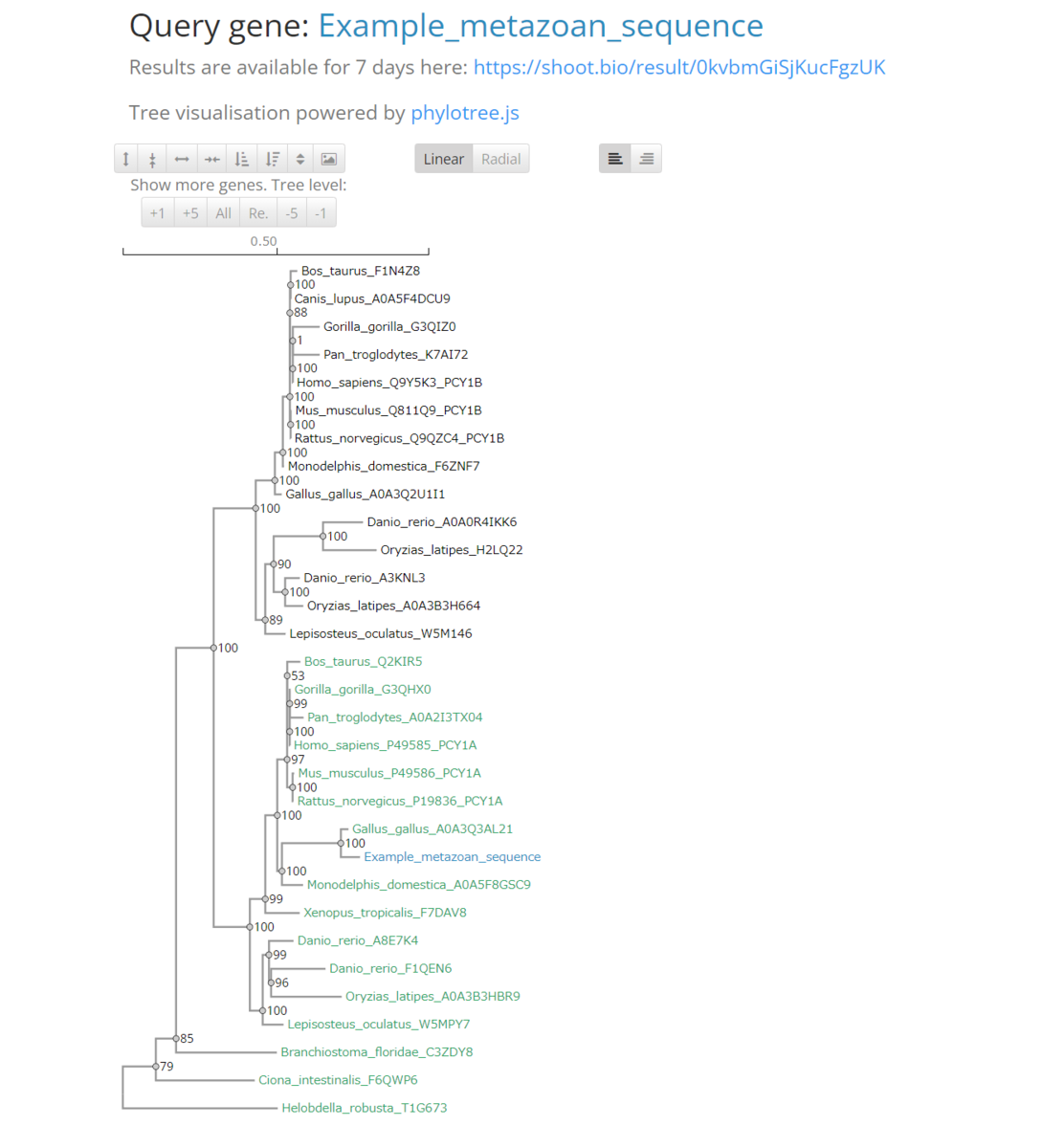
それぞれの配列はUniProtKBにリンクしている。
上のボタンから、系統樹の高さと幅を変更したり、ルート位置を上下反転させたりできる。右端は図のダウンロードボタンになっている。

Radicalに変更。

表示する遺伝子数を+、-ボタンで変更して系統樹のサイズを変えることができる。Allだと全遺伝子の系統樹が表示される。


引用
SHOOT: phylogenetic gene search and ortholog inference
David Mark Emms & Steven Kelly
Genome Biology volume 23, Article number: 85 (2022)
関連
このwebツールは内藤先生のリイートで知りました。
ありがとうございました。