2020 7/19 benchmark追記、一部修正
2020 10/15 condaインストール追記
2024/05/09 追記
Bwa-mem2はbwaのbwa-memアルゴリズムのネクストバージョンである。bwaと同じアラインメントを生成し、データセット、実行中のマシンに依存して~1.3~3.1倍高速になる。オリジナルのbwaはHeng Liによって開発された。bwa-mem2の性能向上は、主にIntelのParallel Computing LabのVasimuddin MdとSanchit Misraによって行われた。Bwa-mem2はMITライセンスで配布されている。
一般ユーザーは、リリースページにあるコンパイル済みのバイナリを使用することが推奨されている。これらのバイナリは Intel コンパイラでコンパイルされており、gcc でコンパイルされたバイナリよりも高速に動作する。プリコンパイルされたバイナリは、間接的に CPU のディスパッチ(解説)もサポートしている。bwa-mem2 バイナリは、実行中のマシンで利用可能な SIMD 命令セットに基づいて、最も効率的な実装を自動的に選択できる。
BWA-MEM2 production release: Produces alignments identical to BWA-MEM 0.7.17 and is "~1.3-3.0x faster depending on the use-case, dataset and the running machine". https://t.co/asCQah6la1
— Christopher Watson (@ChrisM_Watson) 2020年7月13日
9/11
BWAMEM2 index file size and its memory footprint are now ~16GB down from 42GB.
— Mohd Vasimuddin (@wasim_galaxy) 2020年9月10日
We are actively working on further reducing it down to ~10GB. @sanchit_misra@lh3lh3
#BWA #BWAMEM #BWAMEM2 #Genomics #Bioinformaticshttps://t.co/0s3E71TpqX
2021 3/9
New release of bwa-mem2 is out. This one has passed the validation test - to make sure it produces the same output at bwa v0.7.17 - on ~88 billion reads https://t.co/96a7C0NKHi #bwamem #bwamem2 @lh3lh3 @wasim_galaxy #Genomics pic.twitter.com/oN1CsSgMYa
— Sanchit Misra (@sanchit_misra) 2021年3月8日
インストール
ubuntu18.04でテストした(*1)。
CentOS6マシン上でコンパイルされたバイナリが用意されている。これをダウンロードして使用する。
curl -L https://github.com/bwa-mem2/bwa-mem2/releases/download/v2.0pre2/bwa-mem2-2.0pre2_x64-linux.tar.bz2 \
| tar jxf -
cd bwa-mem2-2.0pre2_x64-linux/
#bioconda (link)
conda install -c bioconda bwa-mem2 -y
> ./bwa-mem2
$ ./bwa-mem2
Usage: bwa-mem2 <command> <arguments>
Commands:
index create index
mem alignment
version print version number
> ./bwa-mem2 mem # 最適なSIMD対応バイナリが自動で選択される(赤字)
# ./bwa-mem2 mem
-----------------------------
Executing in AVX2 mode!!
-----------------------------
Usage: bwa2 mem [options] <idxbase> <in1.fq> [in2.fq]
Options:
Algorithm options:
-o STR Output SAM file name
-t INT number of threads [1]
-k INT minimum seed length [19]
-w INT band width for banded alignment [100]
-d INT off-diagonal X-dropoff [100]
-r FLOAT look for internal seeds inside a seed longer than {-k} * FLOAT [1.5]
-y INT seed occurrence for the 3rd round seeding [20]
-c INT skip seeds with more than INT occurrences [500]
-D FLOAT drop chains shorter than FLOAT fraction of the longest overlapping chain [0.50]
-W INT discard a chain if seeded bases shorter than INT [0]
-m INT perform at most INT rounds of mate rescues for each read [50]
-S skip mate rescue
-o output file name missing
-P skip pairing; mate rescue performed unless -S also in use
Scoring options:
-A INT score for a sequence match, which scales options -TdBOELU unless overridden [1]
-B INT penalty for a mismatch [4]
-O INT[,INT] gap open penalties for deletions and insertions [6,6]
-E INT[,INT] gap extension penalty; a gap of size k cost '{-O} + {-E}*k' [1,1]
-L INT[,INT] penalty for 5'- and 3'-end clipping [5,5]
-U INT penalty for an unpaired read pair [17]
Input/output options:
-p smart pairing (ignoring in2.fq)
-R STR read group header line such as '@RG\tID:foo\tSM:bar' [null]
-H STR/FILE insert STR to header if it starts with @; or insert lines in FILE [null]
-j treat ALT contigs as part of the primary assembly (i.e. ignore <idxbase>.alt file)
-v INT verbose level: 1=error, 2=warning, 3=message, 4+=debugging [3]
-T INT minimum score to output [30]
-h INT[,INT] if there are <INT hits with score >80% of the max score, output all in XA [5,200]
-a output all alignments for SE or unpaired PE
-C append FASTA/FASTQ comment to SAM output
-V output the reference FASTA header in the XR tag
-Y use soft clipping for supplementary alignments
-M mark shorter split hits as secondary
-I FLOAT[,FLOAT[,INT[,INT]]]
specify the mean, standard deviation (10% of the mean if absent), max
(4 sigma from the mean if absent) and min of the insert size distribution.
FR orientation only. [inferred]
Note: Please read the man page for detailed description of the command line and options.
他にbwa-mem2.avx2、 bwa-mem2.avx512bw、 bwa-mem2.sse41がある。必要に応じてパスを通す。
実行方法
1、indexing
bwa-mem2 index re.fasta

2、mapping
bwa-mem2 mem re.fasta pair1.fq.gz pair2.fq.gz -t 20 > out.sam
高速化と引き換えにメモリ使用量が増加し、indexサイズも相当大きくなっています。導入する際は注意して下さい。
FM-indexを1種類(2bit.64と8bit.32から2bit.64)にしたことと、接尾辞配列を8倍に圧縮したことにより、ディスク上のインデックスサイズが8倍、メモリ上のインデックスサイズが4倍小さくなった。例えば、ヒトゲノムの場合、ディスク上のインデックスサイズは〜80GBから〜10GBに減少し、メモリフットプリントは〜40GBから〜10GBに減少した。インデックス構造の変更(2020年10月10日のコミット#4b59796)により、インデックスを再構築する必要がある。(マニュアルより)
追記
シロイヌナズナゲノム(120-Mb)を使って簡単なベンチマークを行いました。3つの計算機を使い、スレッド数を変えて10GB fastqのbwa-mem2マッピングランタイム(real)とその時のピークメモリを比較した結果が次の表です。
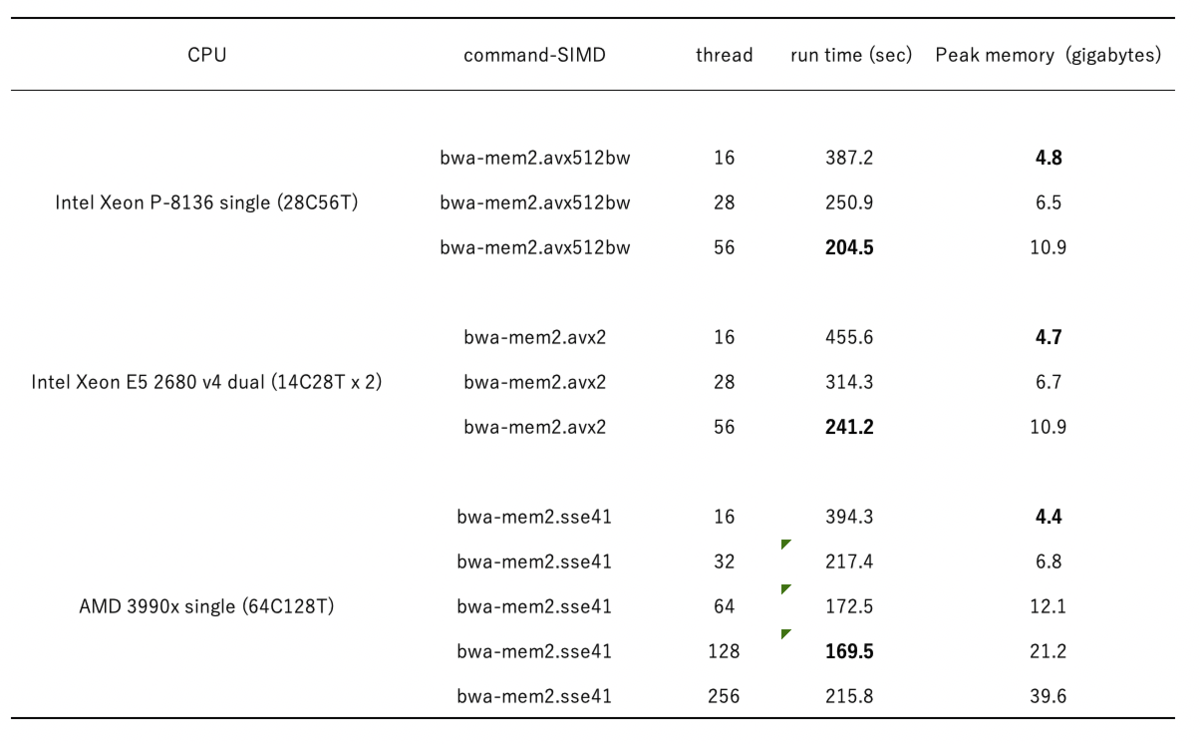
SR3990Xは結果が揺らいだので5回行なった平均値を載せています。他2つのCPUは1回ずつの結果です。SR3990Xは64スレッド指定してもCPU率はまだ半分近く余っていたので、念のためCPUのスレッド数の倍である256を指定し、飽和値指定した時のタイムも測定しました。
引用
https://github.com/bwa-mem2/bwa-mem2
Vasimuddin Md, Sanchit Misra, Heng Li, Srinivas Aluru. Efficient Architecture-Aware Acceleration of BWA-MEM for Multicore Systems. IEEE Parallel and Distributed Processing Symposium (IPDPS), 2019.
参考
*1
CPUがintelのxeon platinum だと、avx512bwも含めて配布されているバイナリ全てが動作した。xeon E5 2680 v4ではavx512bw以外は動作した。AMDのSR3990xだとSSE41 以外は動作しなかったため、ソースからビルドした。
関連
2021 12/11
With the recent (last year) advances in short-read aligners, it would be nice to see an independent benchmarking. I ran some of the tools in my small benchmarking, I include results and brief conclusions below. Could serve as inspiration for a more thorough benchmark. https://t.co/PHxBFLeA3a pic.twitter.com/iYBAI7vCiX
— Kristoffer Sahlin (@krsahlin) December 10, 2021