2020 6/21 タイトル変更
2021 11/23 インストール手順修正
2024/12/27 追記
MCScanは、複数のゲノムまたはサブゲノムをスキャンして、相同性のあると思われる染色体領域を特定し、遺伝子をアンカーにしてこれらの領域を整列させることができるアルゴリズムである。MCScanXツールキットは、syntenyとcollinearityの検出のために調整されたMCScanアルゴリズムを実装しており、結果の可視化と下流の追加解析のための14のユーティリティプログラムを組み込むことで、オリジナルのソフトウェアを拡張している。いくつかの植物ゲノムと遺伝子ファミリーへのMCScanXの適用例を示す。染色体の構造変化を効果的に解析し、系統や分類群の適応に寄与すると考えられる遺伝子ファミリーの拡大の歴史を明らかにするためにMCScanXを使用することができる。gene duplicationの様々なモードの統合的なビューは、特定のファミリーにおける従来の遺伝子ツリー解析を補完できる。MCScanXのソースコードとドキュメントは http://chibba.pgml.uga.edu/mcscan2/ から自由に入手できる。(2022/11/16現在リンク切れ)
MCScanXアルゴリズムはMCScan(19)の修正版である。全ゲノムBLASTPの結果は、染色体とscaffoldsのすべての可能なペアのコリニアブロックを計算するために使用される。まず、BLASTPのマッチは遺伝子の位置に応じてソートされる。タンデム配列に起因する局所的なコリニアな遺伝子ペアの多さを避けるために、連続したBLASTPマッチが共通の遺伝子を持ち、そのペアの遺伝子が5つ以下の遺伝子で区切られている場合、これらのマッチは、BLASTPのE値が最も小さい代表ペアを使用して折りたたまれる。次に、動的プログラミングは、2つの遺伝子ペア、uとvが、uがvに先行するパス上にあると仮定して、以下のスコアリングスキーマを使用して、最も高いスコアリングパス(すなわち、コリネア遺伝子ペアの連鎖)を見つけるために採用される。(以下略)
MCScanX HP
http://chibba.pgml.uga.edu/mcscan2/
https://twitter.com/search?q=MCScanX&src=typed_query
インストール
上のHPからダウンロードした。
git clone https://github.com/wyp1125/MCScanX.git
cd MCScanX/
make -j 8
export PATH=$PATH:$PWD
> ./MCScanX
$ ./MCScanX
[Usage] ./MCScanX prefix_fn [options]
-k MATCH_SCORE, final score=MATCH_SCORE+NUM_GAPS*GAP_PENALTY
(default: 50)
-g GAP_PENALTY, gap penalty (default: -1)
-s MATCH_SIZE, number of genes required to call a collinear block
(default: 5)
-e E_VALUE, alignment significance (default: 1e-05)
-m MAX_GAPS, maximum gaps allowed (default: 25)
-w OVERLAP_WINDOW, maximum distance (# of genes) to collapse BLAST matches (default: 5)
-a only builds the pairwise blocks (.collinearity file)
-b patterns of collinear blocks. 0:intra- and inter-species (default); 1:intra-species; 2:inter-species
-h print this help page
> ./MCScanX_h
$ ./MCScanX_h
[Usage] ./MCScanX_h prefix_fn [options]
-k MATCH_SCORE, final score=MATCH_SCORE+NUM_GAPS*GAP_PENALTY
(default: 50)
-g GAP_PENALTY, gap penalty (default: -1)
-s MATCH_SIZE, number of genes required to call collinear blocks
(default: 5)
-e E_VALUE, alignment significance (default: 1e-05)
-m MAX_GAPS, maximum gaps allowed (default: 25)
-w OVERLAP_WINDOW, maximum distance (# of genes) to collapse BLAST matches (default: 5)
-a only builds the pairwise blocks (.collinearity file)
-b patterns of collinear blocks. 0:intra- and inter-species (default); 1:intra-species; 2:inter-species
-c whether to consider homology scores. 0:not consider (default); 1: lower preferred; 2: higher preferred
-h print this help page
>./Duplicate_gene_classifier
$ ./Duplicate_gene_classifier
[Usage] ./Duplicate_gene_classifier prefix_fn [options]
-k MATCH_SCORE, final score=MATCH_SCORE+NUM_GAPS*GAP_PENALTY
(default: 50)
-g GAP_PENALTY, gap penalty (default: -1)
-s MATCH_SIZE, number of genes required to call a collinear block
(default: 5)
-e E_VALUE, alignment significance (default: 1e-05)
-w OVERLAP_WINDOW, maximum distance (# of genes) to collapse BLAST matches (default: 5)
-m MAX_GAPS, maximum gaps allowed (default: 25)
-n N_PROXIMAL, maximum distance (# of genes) to call proximal (default: 10)
-h print this help page
実行方法
調べたいゲノムのprotein.fasta、リファレンス側のゲノムのprotein.fastaとBED/GFF ファイルを用意する。BED/GFFファイルには染色体名、遺伝子名、スタート、エンドのポジションが書かれていなければならない。
$ head data/os_sb.gff
os5 Os05t0507200 25150045 25150392
os2 Os02t0774100 33540257 33541838
os5 Os05t0480600 23720056 23723583
os6 Os06t0299300 11200887 11202509
os7 Os07t0587100 24548577 24551667
os5 Os05t0526700 26286838 26287599
os1 Os01t0974500 44835553 44838812
os6 Os06t0608600 25100853 25103401
os6 Os06t0242600 7400125 7400986
os11 Os11t0120600 904677 906689
ファイル名の拡張子以外の名前は統一しておく必要がある。 BED ファイル名がxyz.gffなら、blast出力はxyz.blastという風になっていないといけない。
blast出力結果のテキスト
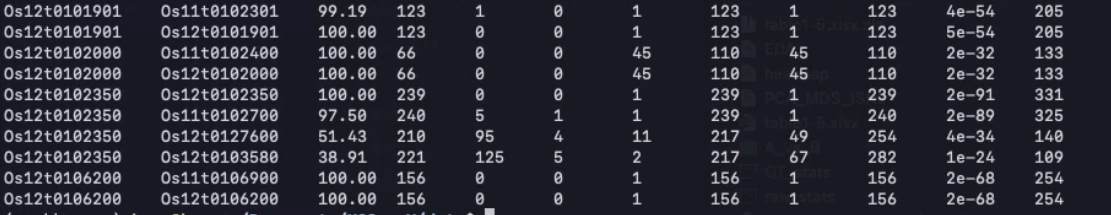
テストラン
blastの出力とGFFファイルを用意する。MCscanX/data/にあるテストデータの1組ならこの写真のようになっている。このprefixを指定してMCScanXをランする。
![]()
1、blastpの実行
blastall -i query_file -d database -p blastp -e 1e-10 -b 5 -v 5 -m 8 -o xyz.blast
2、MCScanXの実行。dataディレクトリのos_sb.blastとos_sb.gffを対象に以下のようにランする。
MCScanX os_sb
出力

> head -n 20 data/os_sb.collinearity
$ head -n 20 data/os_sb.collinearity
############### Parameters ###############
# MATCH_SCORE: 50
# MATCH_SIZE: 5
# GAP_PENALTY: -1
# OVERLAP_WINDOW: 5
# E_VALUE: 1e-05
# MAX GAPS: 25
############### Statistics ###############
# Number of collinear genes: 33937, Percentage: 50.45
# Number of all genes: 67274
##########################################
## Alignment 0: score=559.0 e_value=1.8e-34 N=13 os1&os1 plus
0- 0: Os01t0584200 Os01t0713850 7e-15
0- 1: Os01t0584900 Os01t0714800 2e-30
0- 2: Os01t0588200 Os01t0715500 9e-12
0- 3: Os01t0589200 Os01t0716500 7e-62
0- 4: Os01t0593200 Os01t0719000 7e-25
0- 5: Os01t0593700 Os01t0719300 0
0- 6: Os01t0595100 Os01t0719400 2e-09
0- 7: Os01t0595201 Os01t0719600 1e-22
blast出力以外にペアワイズのアラインメントがあればそれを使用してランすることもできる(MCScanX_hを使用)。ファイル例はat_gm_pt_vv.homologyを参照。
MCScanXによるコリニアブロックのマルチプルアラインメントを表示したHTML出力。

sb9.html
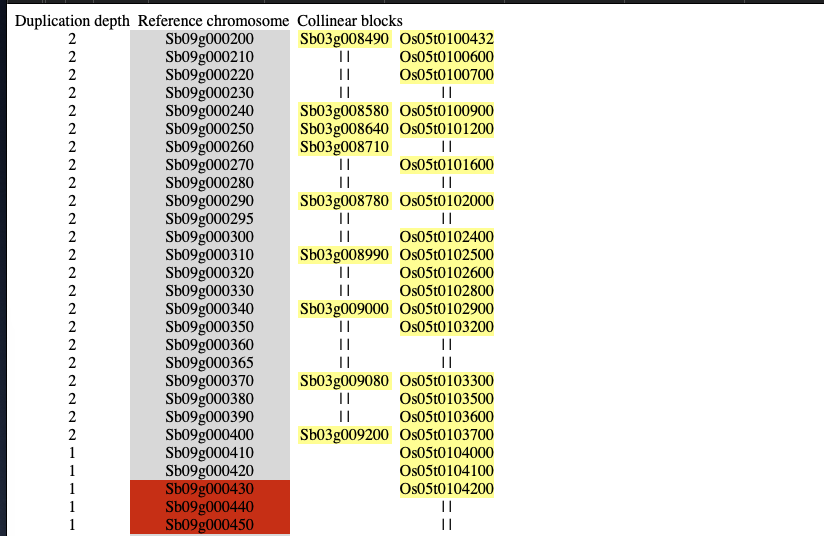
もう1つのテストデータ
./MCScanX data/at_vv
他に重複遺伝子を見つけるためのduplicate_gene_classifierがある。
./duplicate_gene_classifier data/os_sb
追記
結果はSynVisioを使って視覚化できる。
MCScanXのテストデータを視覚化した。
引用
MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity
Yupeng Wang, Haibao Tang, Jeremy D. DeBarry, Xu Tan, Jingping Li, Xiyin Wang, Tae-ho Lee, Huizhe Jin, Barry Marler, Hui Guo, Jessica C. Kissinger, Andrew H. Paterson
Nucleic Acids Res. 2012 Apr; 40(7), Published online 2012 Jan 4
