ロリポップダイアグラム は、ガンゲノミクスにおける遺伝子変異のトランスレーショナル効果を可視化し、探索するために広く用いられているグラフィカルな表現の一つである。しかし、使いやすい機能を備えたロリポップダイアグラムツールはまだ不足している。ここでは、研究者がウェブブラウザ上でロリポップダイアグラムを用いて遺伝子変異データを探索できるようにするRパッケージ、g3vizを紹介する。わずか数行のRコードで、ユーザーはデータの詳細をインタラクティブに可視化し、所見に注釈を付け、結果として得られた図を高品質の図にエクスポートすることができる。その有用性と使い勝手の良さから、g3vizは、バイオインフォマティクスのスキルやプログラミングの経験が豊富な研究者に広く利用されている。R パッケージは CRAN (http://cran.r-project.org/web/packages/g3viz) から MIT ライセンスで自由に入手できる。g3lollipop JavaScriptパッケージはGitHub (https://github.com/g3viz/g3lollipop.js)からMITライセンスで自由に利用できる。
Package ‘g3viz’
https://cran.r-project.org/web/packages/g3viz/g3viz.pdf
Tutorial
http://127.0.0.1:23058/library/g3viz/doc/chart_themes.html
インストール
macos10.14にてRstudioを使ってテストした。
依存
Depends R (>= 3.0.0)
#CRAN(link)
install.packages('g3viz', dependencies = TRUE)
library(g3viz)
help
> browseVignettes("g3viz")
実行方法
ここではGithubに用意されているテストデータ(g3viz/visual_test/tables/tp53-msk_impact_2017.tsv)を使う。
1、readMAF関数を使ってmutation.datデータフレームにバリアント情報を読み込む。
mutation.dat <- readMAF("/Users/kazuma/Downloads/g3viz-master/visual_test/tables/tp53-msk_impact_2017.tsv", sep="\t")
自分のデータを使うなら,setwd("<PATH>") で作業ディレクトリに移動して実行するか、ファイルをフルパスで記載する。
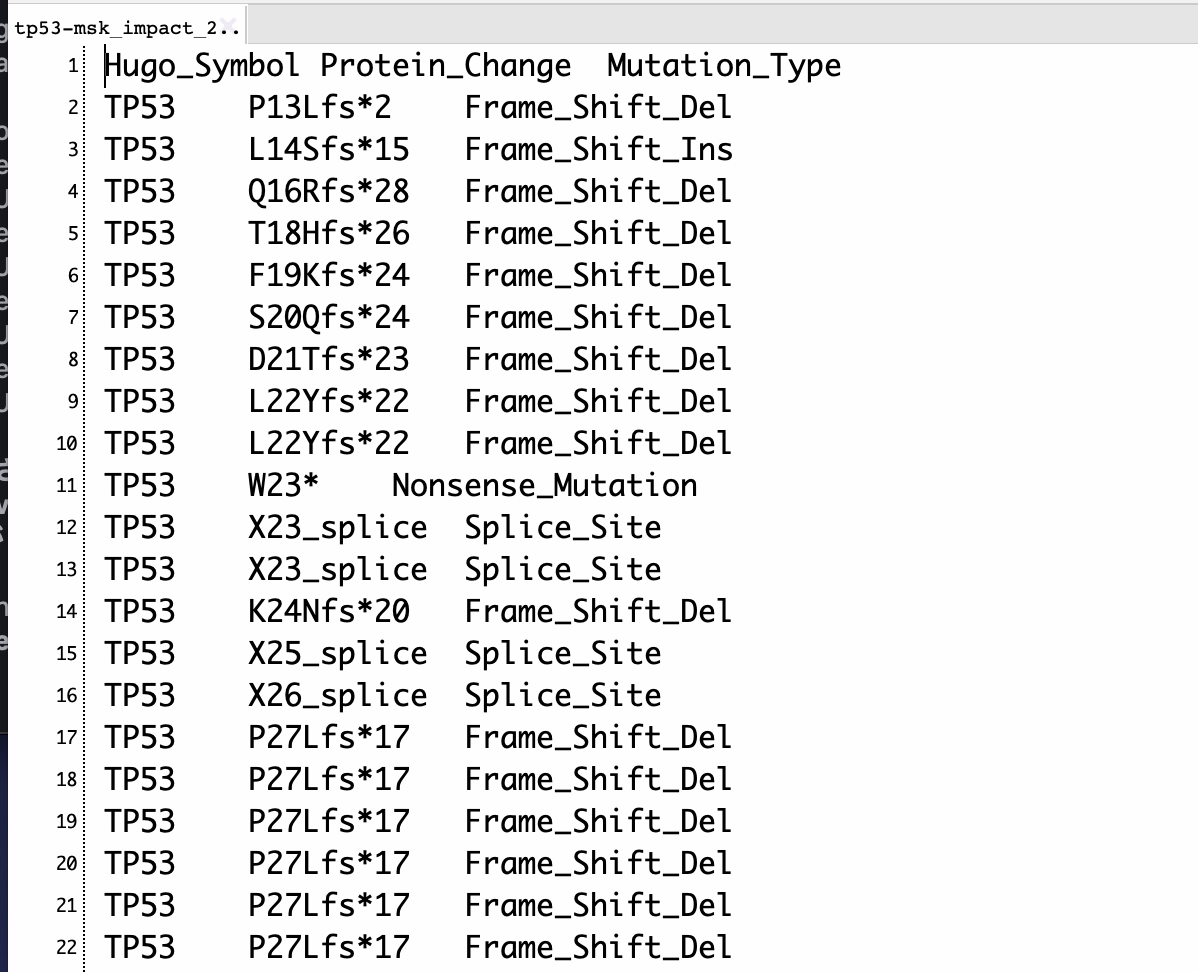
読み込んでいるデータは次のような形式になる。HUGOのgene symbol、アミノ酸変異とその検体数、変異の種類になる。
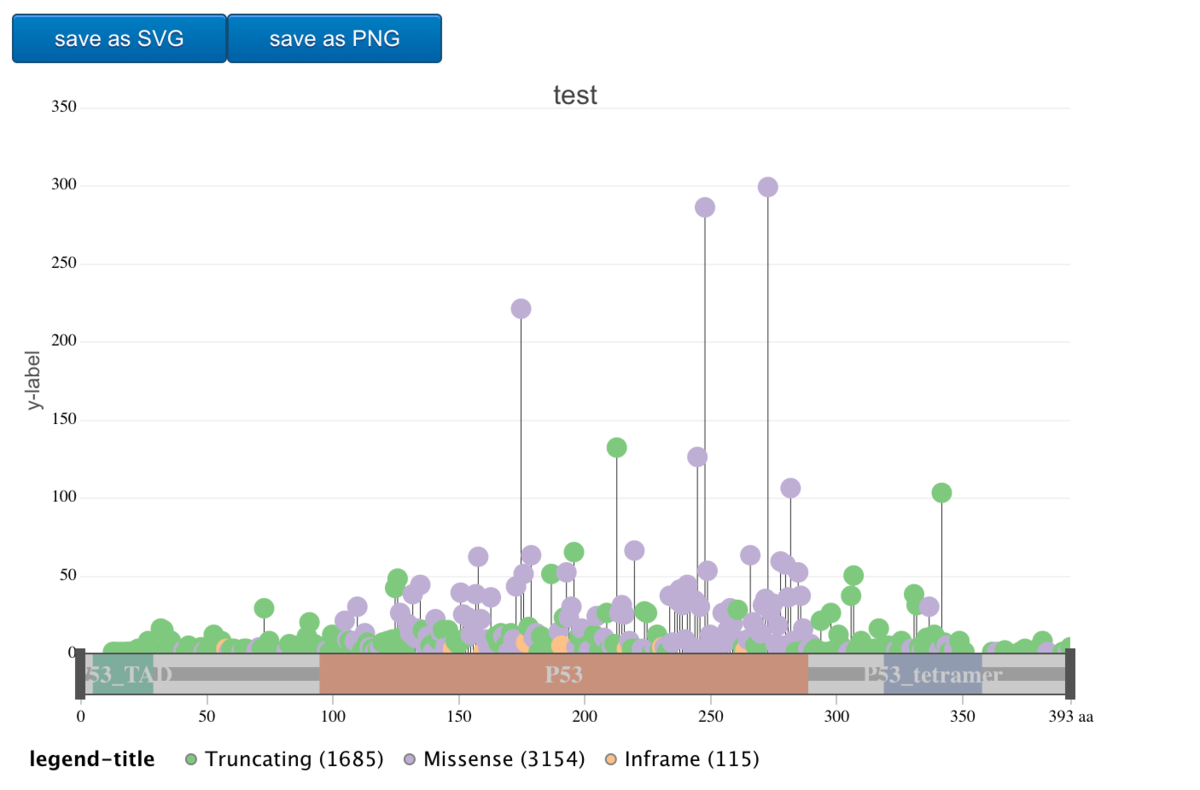
2、g3Lollipopを使ってインタラクティブな図を出力する。まずはテーマ"simple"を使ってみる。
g3Lollipop(mutation.dat,
plot.options =
g3Lollipop.theme(theme.name = "simple",
title.text = "test",
y.axis.label = "y-label",
legend.title = "legend-title"),
btn.style = "blue",
gene.symbol = "TP53")
- mutation.dat Input genomic mutation data frame
- plot.options g3lollipop diagram options in list format. Check g3Lollipop.options
- gene.symbol HGNC primary gene symbol
- btn.style button style, including browser default button style, and two built-in styles, blue or gray. Default NA, indicating browser default.
- theme.name me name, including default, cbioportal, nature, nature2, dark, blue, ggplot2, and simple. Default default.
遺伝子名はHGNCのgene symbolに対応している。
出力。 gene symbolが記載されている領域が対象になる。
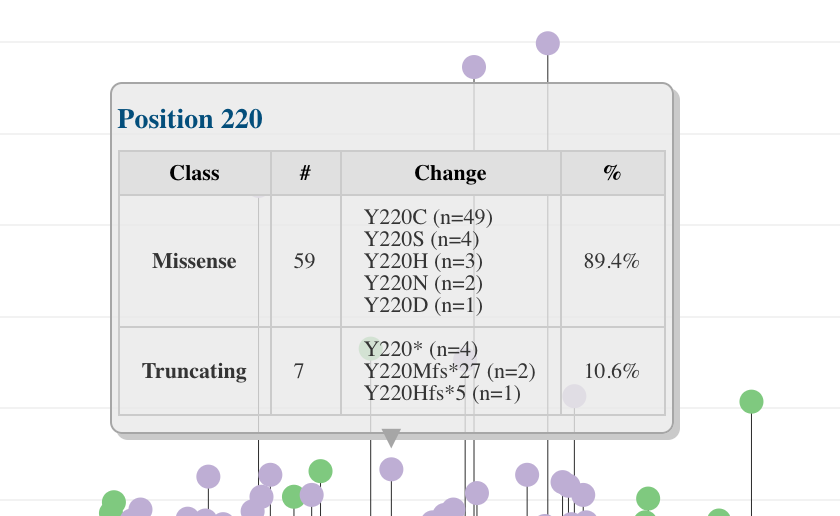
カーソルを合わせると、多型/変異のタイプや内訳を確認できる。
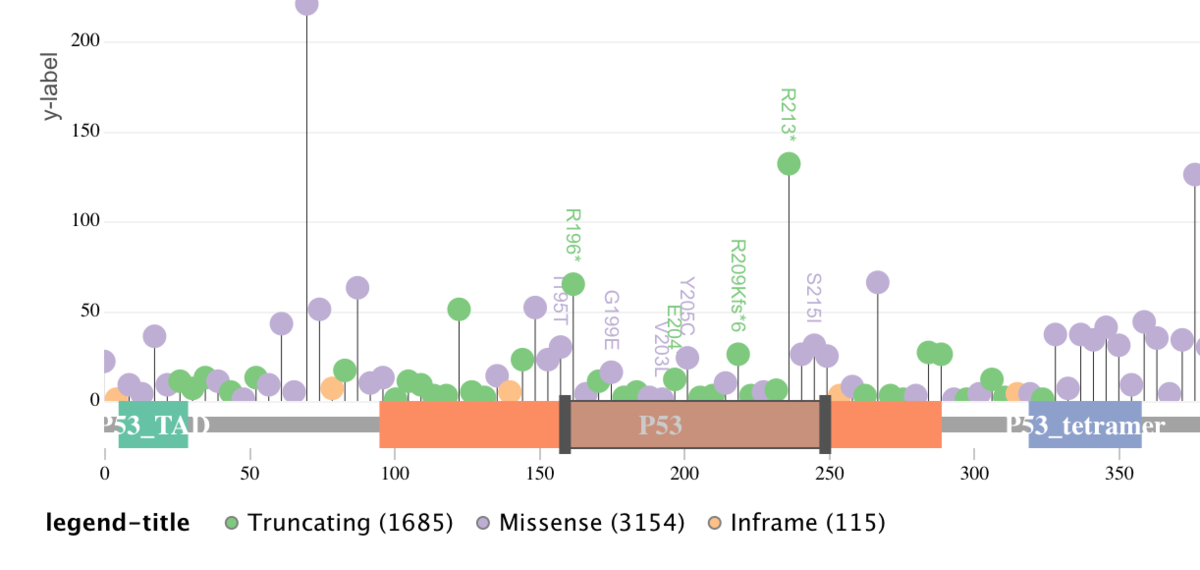
マウスホイールで拡大してアミノ酸変化の注釈をつけた。
結果はwebページとして出力できる。いつでもブラウザに読み込んでインタラクティブに操作できるようになっている。
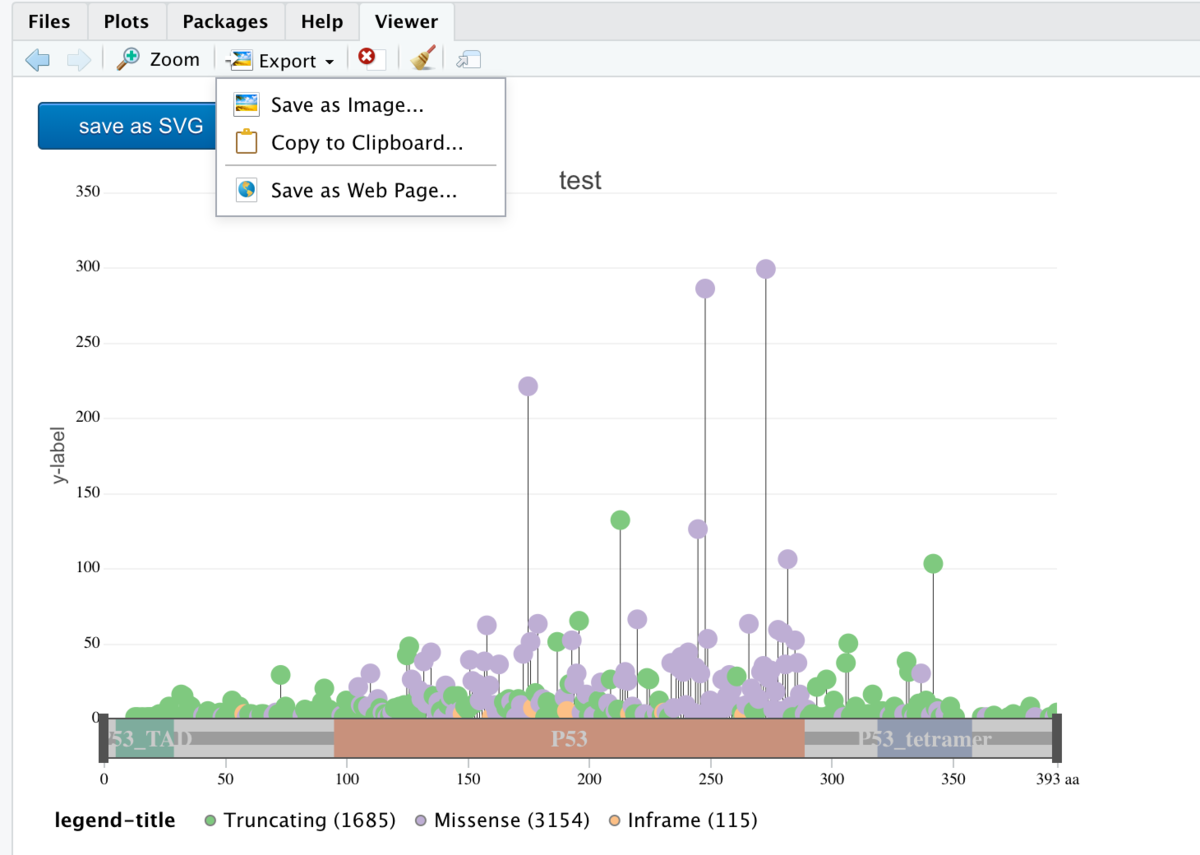
Rstudioの場合。
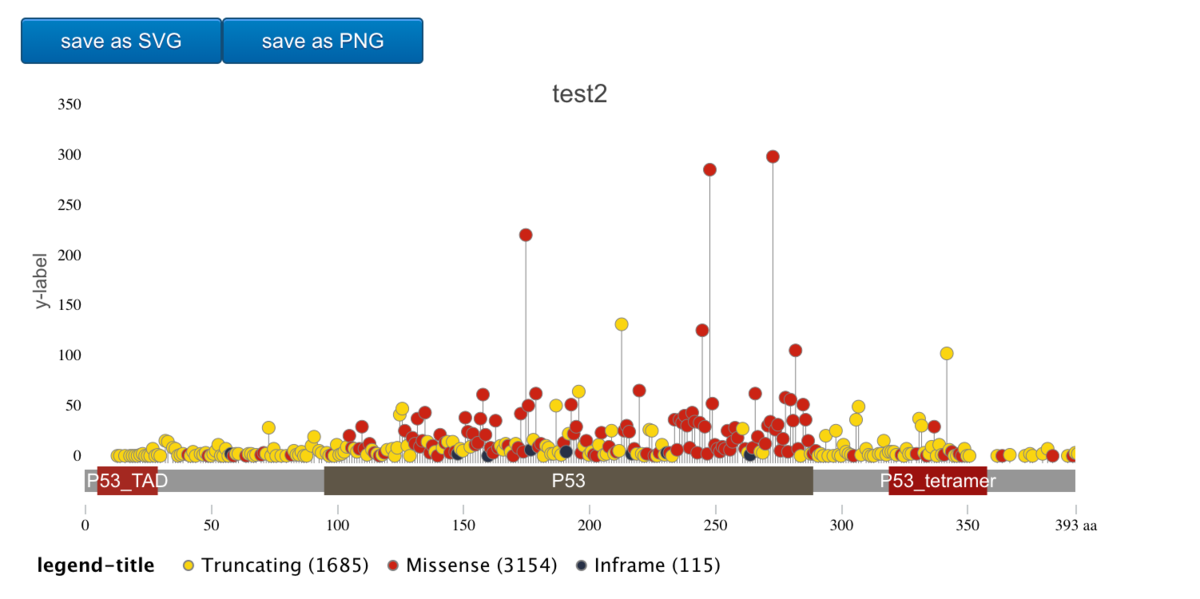
テーマcBioPortal
g3Lollipop(mutation.dat,
plot.options =
g3Lollipop.theme(theme.name = "cbioportal",
title.text = "test2",
y.axis.label = "y-label",
legend.title = "legend-title"),
btn.style = "blue",
gene.symbol = "TP53")
テーマggplot2
g3Lollipop(mutation.dat,
plot.options =
g3Lollipop.theme(theme.name = "ggplot2",
title.text = "test3",
y.axis.label = "y-label",
legend.title = "legend-title"),
btn.style = "blue",
gene.symbol = "TP53")
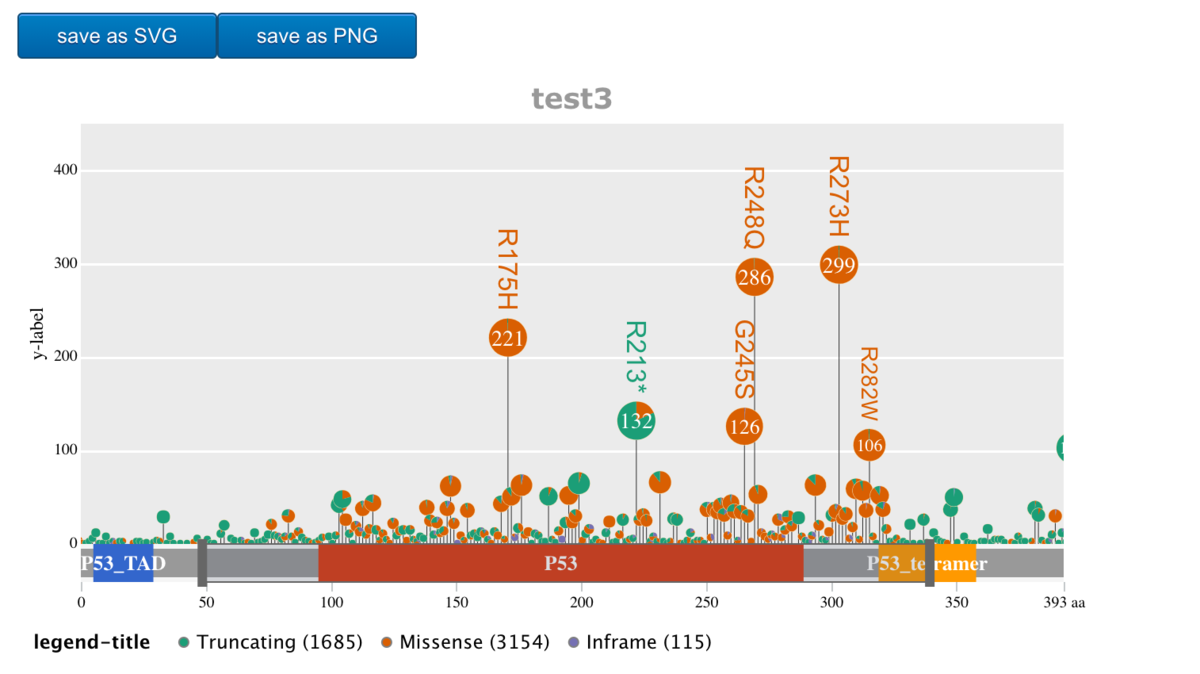
少し拡大してキャプチャした。
他の例はチュートリアルを参照して下さい。
引用
G3viz: an R package to interactively visualize genetic mutation data using a lollipop-diagram
Xin Guo, Bo Zhang, Wenqi Zeng, Shuting Zhao, Dongliang Ge
Bioinformatics, Volume 36, Issue 3, 1 February 2020, Pages 928–929