大規模なRNAシーケンス(RNA-Seq)は、ゲノムシーケンスの実用的な代替手段として、特に比較分析のために非モデル種でよく使用される(Ozsolak and Milos、2011; Todd et al、2016; Wang et al 、2009)。しかし、トランスクリプトームアッセイのショートリードからの完全長の転写配列へのアセンブリは、反復領域、発現レベル変動、選択的スプライシング、シーケンシングエラー、および組成バイアスに関連する困難な問題を提起する(Garber et al、2011)。さらに、これらの配列を遺伝子ファミリーにクラスター化すること、それらのアラインメント、および遺伝子ツリー再構築のステップはすべて、合意された基準が無い、比較ゲノミクス研究が直面する課題を表している。
近縁種のゲノムデータの存在に応じて、転写産物のアセンブリにさまざまな戦略を使用できる(Conesa et al、2016; Ockendon et al、2016)。シーケンスされたゲノムを持つ姉妹種が利用できない場合、リードは重複するシーケンスに基づいてデノボでアセンブルされる(例えばTrinity、 Grabherr et al(2011))。それ以外の場合は、ゲノムガイドアセンブリが使用できる(例:Tophat、Trapnell et al (2009)、 Cufflinks、Trapnell et al(2010))。その場合、リードはこのガイドゲノムに合わせて行われ、ローカルの転写産物アセンブリに使用されるリードのクラスターが作成される。この戦略は明らかに、非常にclosely relatedな種、すなわち種を超えてリードのマッピングが可能な場合に限定される。より遠い関係にある種では、RNA-Seqアセンブリに対するアプローチは提案されていないが、開発が続いている。特に、Johnson et al(2013)によるTarget Restricted Assembly Method (TRAM) 、自動のaTRAM(Allen et al、2015)では、BLAST(Camacho et al、2009)を使用してリファレンスゲノムとの配列類似性によってリードが繰り返しコレクションされ、その後遺伝子配列が再構築、アセンブリされる。 k-mersアプローチに基づいてKollectorで異なる実装が提案された(Kucuk et al、2017)。これらの方法は有望な結果を示しているが、RNA-Seqデータおよび一度に数千の遺伝子に使用するようには設計されていない。
アセンブリ後、転写産物には遺伝子名のアノテーションを付けるのが理想的である。一般に、トランスクリプトームアノテーションは、すでにアノテーションが付けられている種のトランスクリプトームとトランスクリプトームの間の配列類似性に基づいている。このステップは、通常、種固有のduplicationsを処理できないBLAST(Camacho et al、2009)を使用して、Reciprocal Best Hits(RBH)(Rivera et al、1998)によって処理されることがほとんどである(Altenhoff and Dessimoz、2009; Tekaia、2016)。多くの遺伝子が重複しているため、これは問題である。たとえば、Ensemblデータベース(Herrero et al、2016; Yates et al、2016)では、すべてのヒト遺伝子の10%がマウス遺伝子と1対1のorthology関係を持たない。
原則として、アノテーションをRBHではなく遺伝子系統に依存することで、複雑な相同関係を処理することができる(Chen et ak、2007; Kristensen et al、2011; Kuzniarアノテーション、2008 et al; Tekaia、2016)。このようなアプローチを取ることを勧める。アノテーション付きの転写産物からの遺伝子は、de novo(Kristensen et al、2011)または既存のファミリー[EnsemblCompara(Herrero et al、2016)、TreeFam(Finn et 、2014)、Hogenom(Penel et al、2009)、PhylomeDB(Huerta-Cepas et al、2014)]から相同な遺伝子ファミリーにクラスター化される。次に、再構築された転写産物は、配列の類似性に基づいて遺伝子ファミリーに統合される。これらの拡大された遺伝子ファミリーのアライメントとツリーが再構築される。ツリーの品質は、種のツリーによって提供される情報を使用する再構築方法を使用することで改善できる(Boussau et al、2013; Ullah et al、2015)。最後に、遺伝子ツリーを種のツリーと調整して、種分化、重複、および遺伝子の欠失(loss)にアノテーションを付ける(Kristensen et al、2011)。遺伝子進化のシナリオに基づいて、オルソロジーとパラロジーの関係が導き出され、遺伝子名がアノテーション付きの配列から新しい転写産物に伝播される(Kristensen et al、2011)。このアプローチでは、正確なアノテーションは正確な遺伝子ツリーの結果である。
ここでは、CAARSという名前の自動化ツールを紹介する。CAARSはよく似た生物、またはより距離のある生物の情報を元にガイドアセンブリしたりアノテーションすることで、非モデル生物のトランスクリプトーム全体をアセンブルし、アノテーションを付ける。CAARSはリファレンス遺伝子のアライメントに依存しており、下流の比較分析に直接使用できる高品質の系統樹とオルソロジー関係を持つ相同遺伝子セットを出力する。 CAARSは、確立されたパイプラインを使って、トランスクリプトームの完全性、トランスクリプトの正確性、およびアノテーションの正確性を改善する。その高品質の出力遺伝子ツリーのおかげで、CAARSは、回復できるオルソログの数の点でEnsemblComparaを改善している。
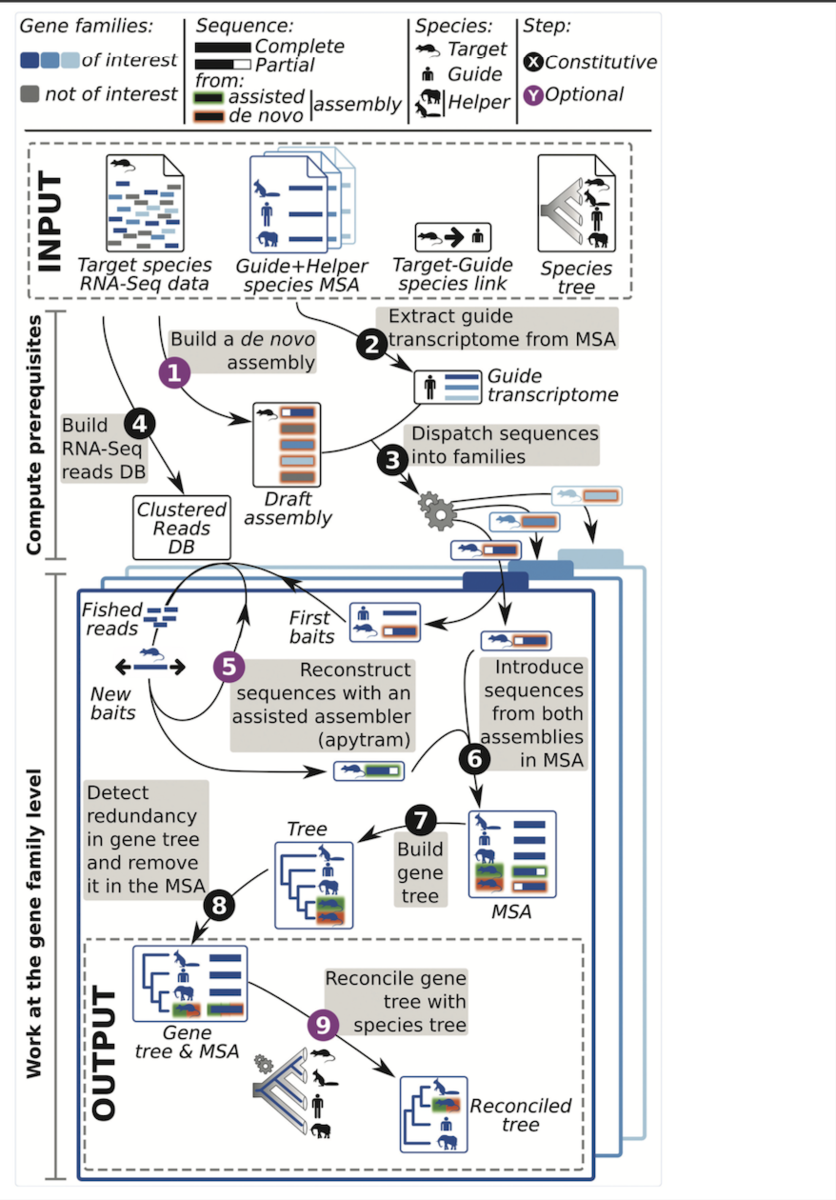
CAARS overview. 論文より転載
https://github.com/CarineRey/caars/wiki/Tutorial#2-running-caars-example-on-mouse-test-data
インストール
#dockerイメージが提供されている
docker pull carinerey/caars
実行方法
ランにはRNA seqのリードのほか、サンプルシート、ツリーファイル、遺伝子ファミリーのアラインメントファイルを使う。
caars --outdir OUTPUT_DIR --sample-sheet sample_sheet.tsv --species-tree /home/user/data/species_tree.nw --alignment-dir GENE_FAMILIES_MSA_DIR --seq2sp-dir GENE_FAMILIES_SEQ2SP_DIR --np 2 --memory 5
作成中
引用
CAARS: comparative assembly and annotation of RNA-Seq data
Rey C, Veber P, Boussau B, Sémon M
Bioinformatics. 2019 Jul 1;35(13):2199-2207
関連