2020 3/3 論文追記
過去数十年にわたって、Genlisea aureaの63 Mb [ref.1]からPinus taedaの22 Gb [ref.2]までのサイズの多数の植物ゲノムアセンブリが生成された。このようなプロジェクトから生成されたゲノムリソースは、改良された作物品種の開発に貢献し、ゲノムサイズ、構造、および複雑さの理解を高め、植物の成長と開発の基礎となるメカニズムを明らかにしてきた[ref.3, 4 ]。シーケンスのコストが低下するにつれて、ゲノムアセンブリの数が指数関数的に増加した(論文補足図1)。 NCBIアセンブリデータベース[ref.5]は現在、800を超える植物ゲノムアセンブリをホストしており、さまざまな程度の隣接性があり、種ごとに複数のゲノムアセンブリが増えている(論文補足図2)。
アセンブリと遺伝子アノテーションの数が増えているため、それらの品質を比較するために使用できるメトリックの開発が必要になった。このようなメトリックは、さまざまなアセンブリのパフォーマンスの評価も可能にする。長さに関するメトリック(N50 / NG50およびL50 / LG50値)は、アセンブリの連続性の標準的な尺度を提供する[ref.6]。最も一般的に報告されているN50 / NG50値は50%のしきい値に対して計算されるが、すべてのしきい値(1〜100%)にわたるNG(X)プロットは、より完全な図を提供する[ref.6]。アノテーションの品質指標には、遺伝子モデルの数、遺伝子モデルごとのエクソン、遺伝子の平均長、エクソンおよび転写産物が含まれる[ref.7]。このような長さとカウントのメトリックは便利だが、アセンブリの完全性を完全に把握しているわけではない。
完全性は、種の特定のクレードにオルソログとして広く分布している遺伝子のセットを使用してより適切に評価される[ref.8]。完全なシングルコピー、複製、断片化、および欠落しているベンチマークユニバーサルシングルコピーオーソログ(BUSCO)遺伝子の概要は、予想される遺伝子量に基づくゲノム完全性の定量的尺度としてよく使用される。 BUSCOは遺伝子空間の評価に限定されているが、LTR Assembly Index [LAI; ref.9]は、インタクトなLTRレトロエレメントの割合を推定することにより、より反復性の高いゲノム領域の完全性を評価することができる。 LAIは、植物ゲノムアセンブリの評価に特に有用である。植物ゲノムアセンブリは、多くの場合、リピートで構成されている。最近、ロングリードデータの改善により、植物ゲノムのリピート部分の完全性が劇的に向上した[ref.9]。
ここでは、R / Shinyパッケージ[ref.10]に基づいた使いやすいインタラクティブなWebフレームワークについて説明する。このフレームワークは、ゲノムアセンブリとアノテーションを特徴付ける一連の定量的手段を統合する。 GenomeQCという名前のアプリケーションは、これらの統計の概要を研究者に提供し、ゴールドスタンダードリファレンスアセンブリに対するベンチマークを可能にする。また、これらのメトリクスを計算し、大きな(> 2.5Gb)ゲノムの分析をサポートするGenomeQCパイプラインのDockerコンテナを開発した。
QUAST-LG [ref.11]、Icarus [ref.12]、LASER [ref.13]、REAPR [ref.14]のようなゲノムアセンブリの品質を評価および視覚化するためのツールがいくつかあるが、インストールと構成が難しく、アノテーションを構造化し遺伝子の評価をサポートしておらず、LTRレトロトランスポゾンの内容に基づいてリピート部分の完全性を決定しない。
GenomeQCは、ゲノムアセンブリとアノテーションの連続性と完全性のメトリックを計算するユーザーフレンドリーなWebフレームワークを提供する。このツールは、研究者がゲノムと遺伝子モデルの品質の包括的な評価を取得できるように、複数のパイプラインを統合するという点でユニークである。 Webアプリケーションは、上限が2.5 Gb(トウモロコシゲノムのおおよそのサイズ)の小規模から中規模のゲノムのメトリックを計算するように最適化されている。
また、GenomeQCを使用すると、研究者はゴールドスタンダードリファレンスゲノムと比較して分析をベンチマークできる。(一部略)さらに、GenomeQCでは、NCBI UniVecデータベース[ref.16]に対する汚染チェックを行って、ベクターおよびアダプター配列を特定し、NCBIまたは他のゲノム配列アーカイブに送信する前にこれらを削除またはマスクできる。
コンテナ化されたGenomeQCバージョンは、入力ゲノムアセンブリのLAI値を追加で計算するように構成されている。 LAIはリピートの完全性の非常に有用な尺度だが、
計算式のツールであるため、GenomeQCのコンテナーバージョンでのみ使用できる。
( 以下略)
FAQ
使い方
https://genomeqc.maizegdb.orgにアクセスする。
1、Compare reference genome - すでに公開されているリファレンスゲノム同士の比較
ゲノムを選択する。ここでは3つ選んだ。
メールアドレスを記載してSubmitする。
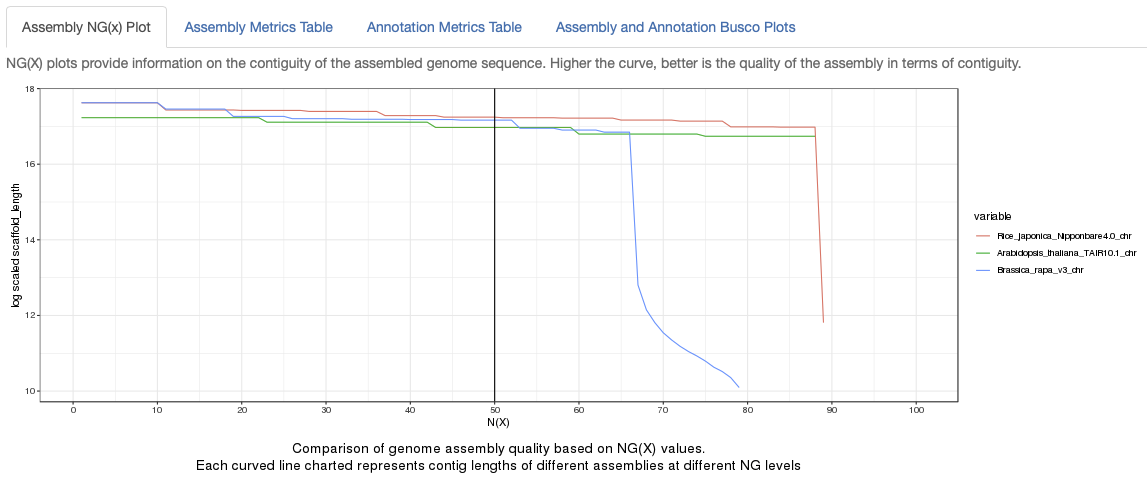
結果
Assembly NG(x) Plot
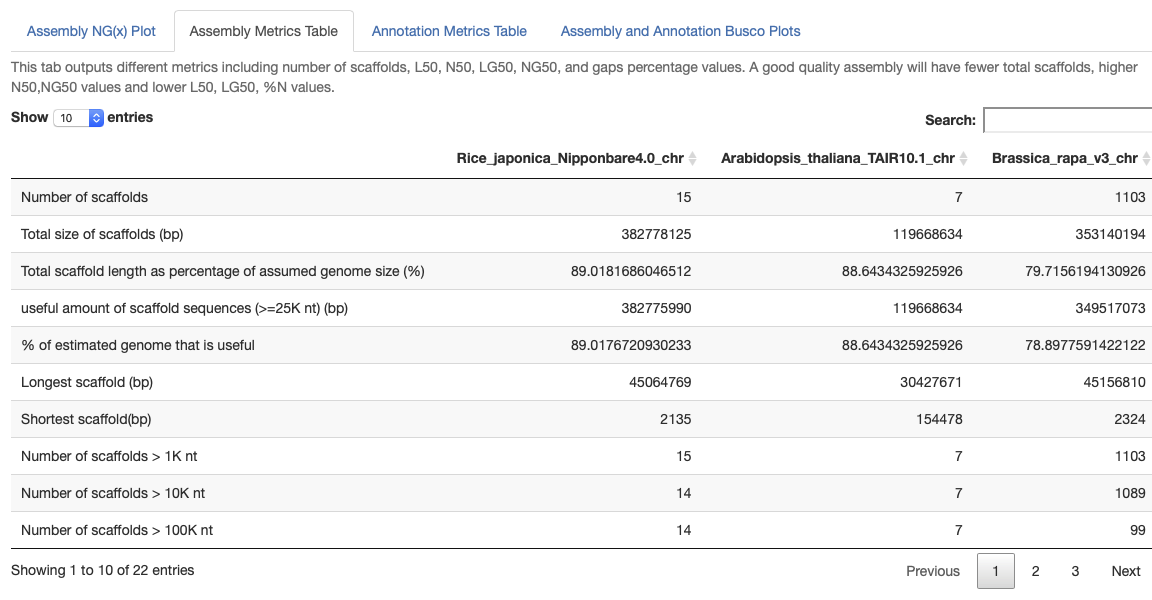
Assembly Metrics Table
長さやcontig数などの標準的なアセンブリmetricsがまとめられる。
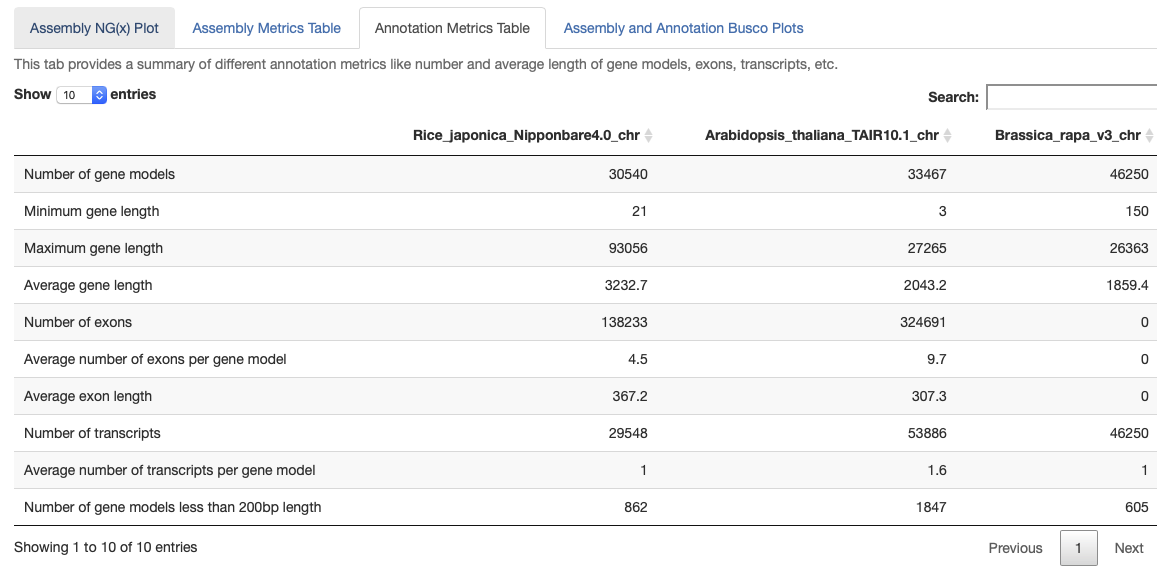
Annotation Metrics Table
アノテーションのmetrics
2、Analyze your genome assembly - ユーザー指定のゲノムアセンブリの分析
ここではこちらで公開されているIpomoea batatas(6倍体)のドラフトゲノムを使ってみる。
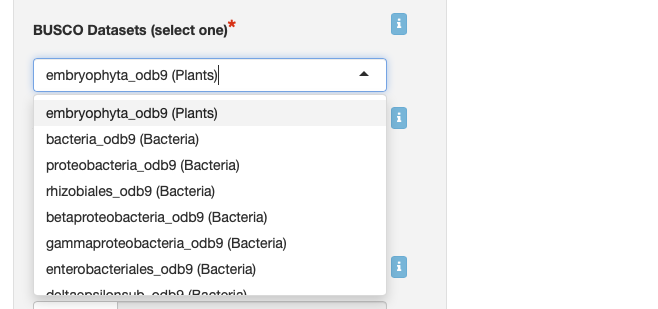
BUSCOで評価する時のデータセットを選択する。
AUGUSTUSの種を選択する。

AUGUSTUSはBUSCOパイプラインで使用されている遺伝子予測プログラムで、その種の遺伝子を予測するために訓練されている。ゲノムのBUSCOスコアを計算するためにも必要なフィールドになる。 該当する種がない場合、最も近い種を選択する。
出力の形式は1と同じになる。
3. Analyse your genome annotation
ゲノムのfastaとアノテーションファイル(gff, gff3 or gtf)をuploadする。 手順は同じになるので説明は省略する。
引用
GenomeQC: A quality assessment tool for genome assemblies and gene struc- ture annotations
Nancy Manchanda, John L. Portwood, II, Margaret R. Woodhouse, Arun S. Seetharam, Carolyn J. Lawrence-Dill, Carson M. Andorf, Matthew B. Hufford
bioRxiv preprint first posted online Oct. 7, 2019
2020 3/3 追記
GenomeQC: a quality assessment tool for genome assemblies and gene structure annotations
Nancy Manchanda, John L. Portwood II, Margaret R. Woodhouse, Arun S. Seetharam, Carolyn J. Lawrence-Dill, Carson M. Andorf, Matthew B. Hufford
BMC Genomics volume 21, Article number: 193 (2020)
関連