シーケンシング技術開発はガン研究に革命をもたらした。約20年に及ぶ発展後、次世代シーケンシング(NGS)は速くて手頃な価格になっている。それは精密医療を臨床の現実にした。 NSGは、臨床現場での治療法を個別化し、研究情報を広げるための包括的なビッグデータを提供している。この技術的進歩は治療および研究のためのより多くの機会を生み出したが、それらは非常に大きく詳細であるため、結果として得られたデータを効率的に合成および要約するという問題も生じた。ビッグデータを手動でフィルタリングすると、エラーの可能性が高まり、整理するのに時間がかかる。ビッグデータも効果的に提示するのは困難である。ソフトウェアはこれらの問題をすべて回避する。この目的のためにいくつかのツールが利用可能である。ただし、ほとんどはプログラミングの背景を持つユーザー向けに設計されている。これは病院やそのような訓練を受けていない施設利用者の大部分を締め出す。 MutplotはWebブラウザで機能する機能し、簡単なカスタマイズのための柔軟性を提供する。臨床医や研究者が自分で使用するために特別に設計された。抽象的なビッグデータを視覚的な結果に変換する。さらに、Mutplotはすべてのプラットフォームで動作するオープンソースのツールであり、セキュリティ目的のため、ファイアウォールの内側で簡単に統合できる。
Mutplotを、MutationMapper [ref.1]、Lollipops [ref.2]、Muts-needle-plot [ref.3]、Pfam [ref.4]、Plot Protein [ref.5]、trackViewer [ref.6]など、他の6つの最も一般的な突然変異プロットツールと比較した。それらのどれも技術的でないユーザーのためのすべての要件を満たさない(詳細は論文表1に示されている)。 MutationMapperを除くそれらすべては、プログラミングトレーニングを必要とするコマンドラインユーザーインターフェイスを使用する。 Muts-needle-plot、Plot Protein、TrackViewerでは、手動のドメイン入力が必要である。Lollipopsは、サンプル頻度が類似している突然変異とクラスタ化突然変異を区別することはできない。その上、手作業でデータを入力することは人的ミスを犯しがちであり、そしてそれは突然変異ハイライト機能を持たない。 Pfamは公開フォーマットではないJSONファイルを出力する。 MutationMapperはWebベースのユーザーインターフェースを使用しているので最良の選択と思われるが、それ自身の欠点もある。それは最も高い再発性の突然変異(アミノ酸の変化)を示すだけで、これは低い頻度であるがドライバー遺伝子の突然変異を排除するだろう[ref.7]。事実、腫瘍間の遺伝的異質性のために、多くのドライバー遺伝子が非常に低いvariant allele frequenciesで生じる。同じ遺伝子に複数の突然変異が発生した場合、MutationMapperは、ガン研究と個別化医療の進歩に不可欠な低発生突然変異を簡単に無視することがあり得る。さらに、2つのバリアントがMutationMapper内で非常に近くにあると、それらのうちの1つからの情報が重複する。 MutationMapperのもう1つの落とし穴は、スペースが限られている場合にドメイン名が自動的に切り捨てられることである。これらの欠点により、MutationMapperはNGS分析にはあまり理想的ではない。
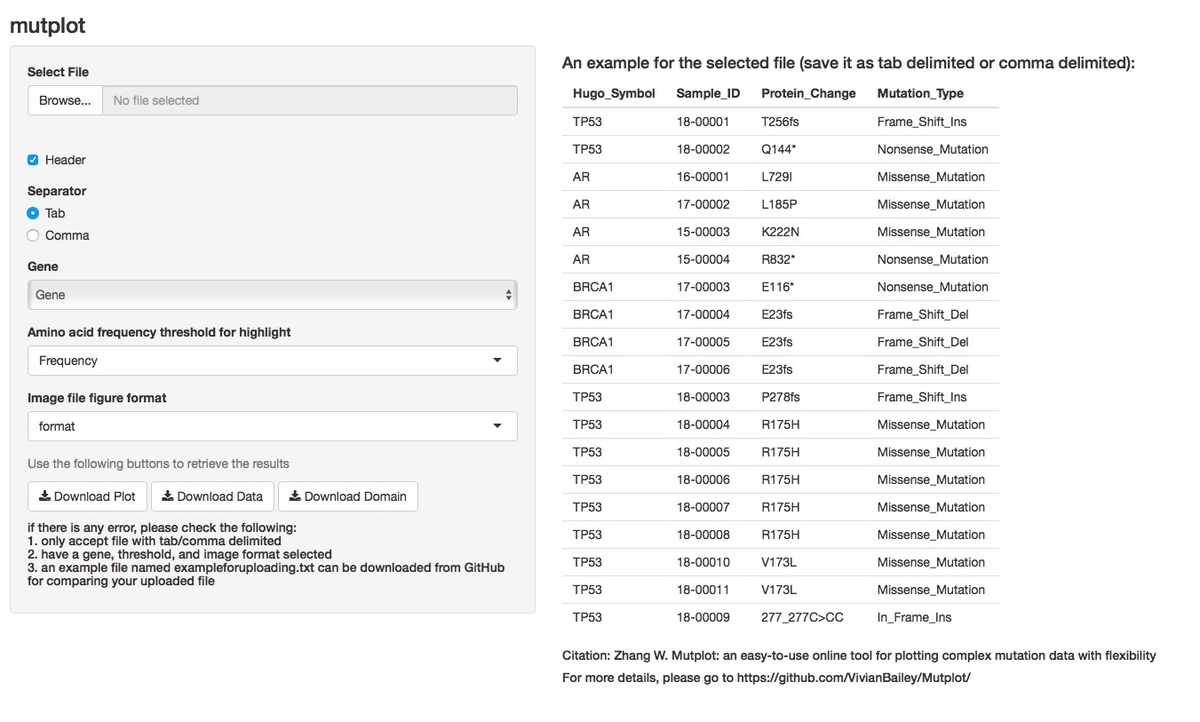
Mutplotには、さまざまなタンパク質の変異を可視化するための完全なワークフローが含まれている(論文図1)。バリアント情報(必要な4つの列はHugo_Symbol、Sample_ID、Protein_Change、およびMutaiton_Type。表S1参照)でファイル(タブ区切り形式またはカンマ区切り形式であること)を入力すると、Mutplotは自動的にUniProtから最新のタンパク質情報に接続する[ref.8]。ドロップダウンメニューを使用して、合計409のガン遺伝子および腫瘍抑制遺伝子が組み込まれている(論文表S2)。 Mutplotは選択された遺伝子のドメイン情報を取得する。アミノ酸頻度閾値のハイライトオプションは1、2、3、4、5、10、15、20、25、30に設定されている。両方の遺伝子とハイライト閾値オプションは、ソースコードをカスタマイズするだけで拡張できる。指示はGitHubにdepositされている:https://github.com/VivianBailey/Mutplot。
この情報を使って、Mutplotはドメイン情報、アミノ酸の位置、変異の頻度、アミノ酸の変化、変異の種類、そして強調表示された変異を含むタンパク質の図を作成する。アミノ酸の位置は正確な比率のために遺伝子の長さに合わせて調整されている。強調表示された変異は詳細なアミノ酸改変情報を有する。突然変異の種類と説明は、視覚化と区別を容易にするために色分けされている。複数の変異が密集している場合、Mutplotはスマートに他の変異を妨害することなく変異を標識する方法を見つけ出す。 Mutplotは出力オプションに関しても高い柔軟性を与える。 JEPG、PDF、PNG、画像ダウンロード用のSVGをサポートしている。また、入力データと更新されたUniprotデータベースから取得した対応するドメイン情報から、ダイアグラム用に選択したデータをエクスポートすることもできる。
このソースコードはGitHub: https://github.com/VivianBailey/Mutplotで利用かのであり、非営利目的で使用することができる。そして簡単にアクセスしたり、修正したり、他のパイプラインやソフトウェアに統合することができる。ソースコードを修正すると、MutplotはWebアプリケーションからファイアウォールの内側にあるパーソナルコンピュータまたはサーバに移行する可能性がある。これは、厳格なセキュリティ規制に従う機関にとって大きな選択肢となる。さらに、GitHubにはMutplotの完全なドキュメント、ソースコードのカスタマイズ方法の説明、そして将来のリリースもGitHubに説明が載っている。
WebアプリはRプログラミング言語で開発された。 shinyパッケージ、ggplot2パッケージ、plyrパッケージ、httrパッケージ、drawProteinsパッケージ、およびggrepelパッケージが使用されている。
使い方
https://bioinformaticstools.shinyapps.io/lollipop/ にアクセスする。
入力ファイルはタブ/カンマ区切りの4列からなるフォーマットで用意する必要がある。それぞれの列には、Hugo_Symbol、Sample_ID、Protein_Change、およびMutaiton_Typeが記載されている必要がある。
入力ファイル例(direct link)。
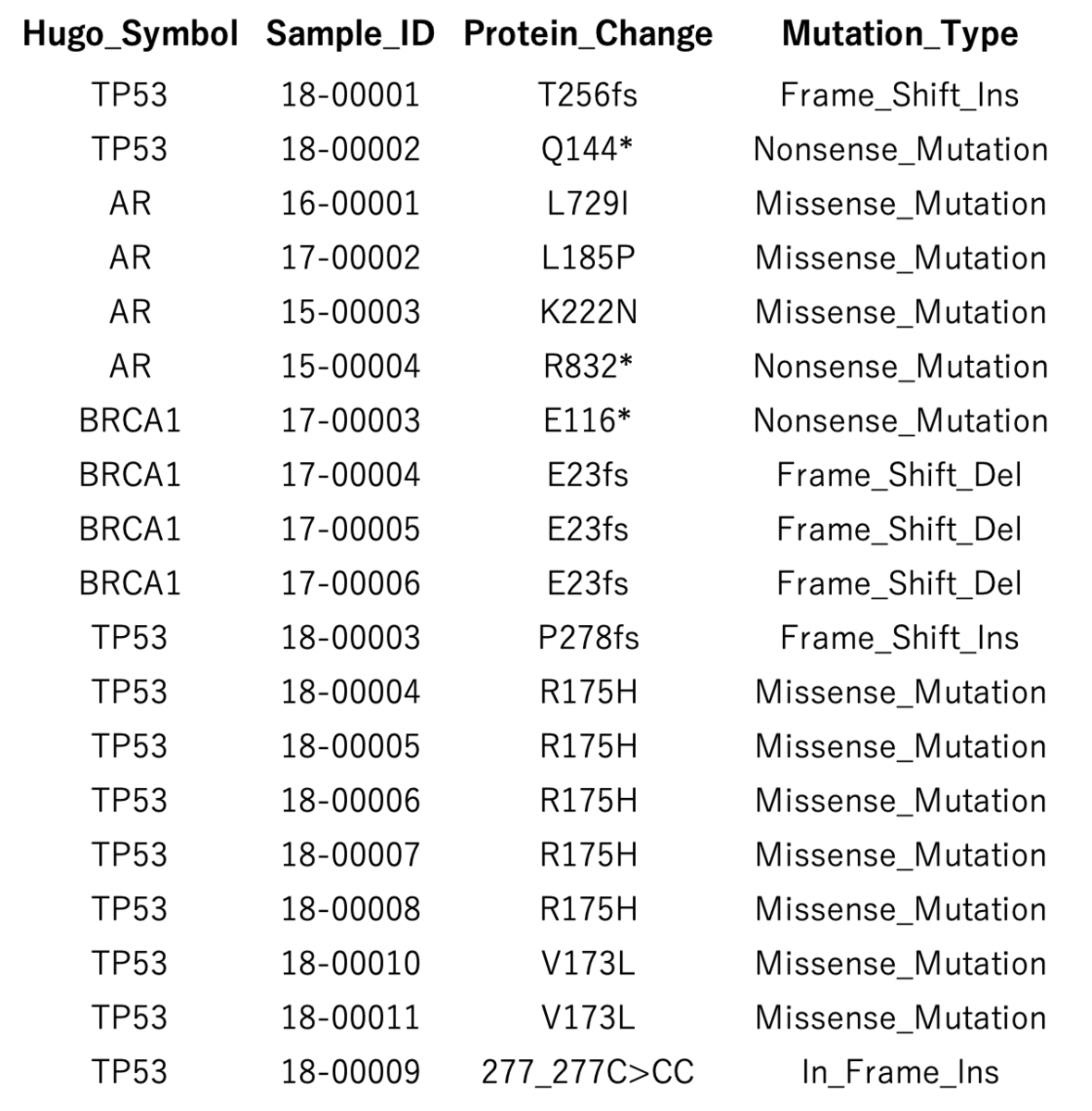
Browseからファイルをアップロードする。
ヘッダーがある場合は「ヘッダー」を選択。入力ファイルに合わせセパレータを指定する。
視覚化する遺伝子を409のリスト中から選択。ここではテストデータを使っているので、TP53遺伝子か、BRCA1を選ぶ。
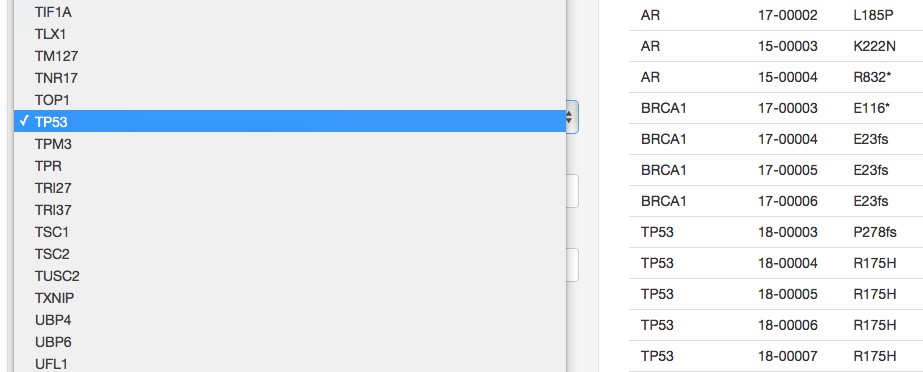
利用できる遺伝子リスト(direct link)。
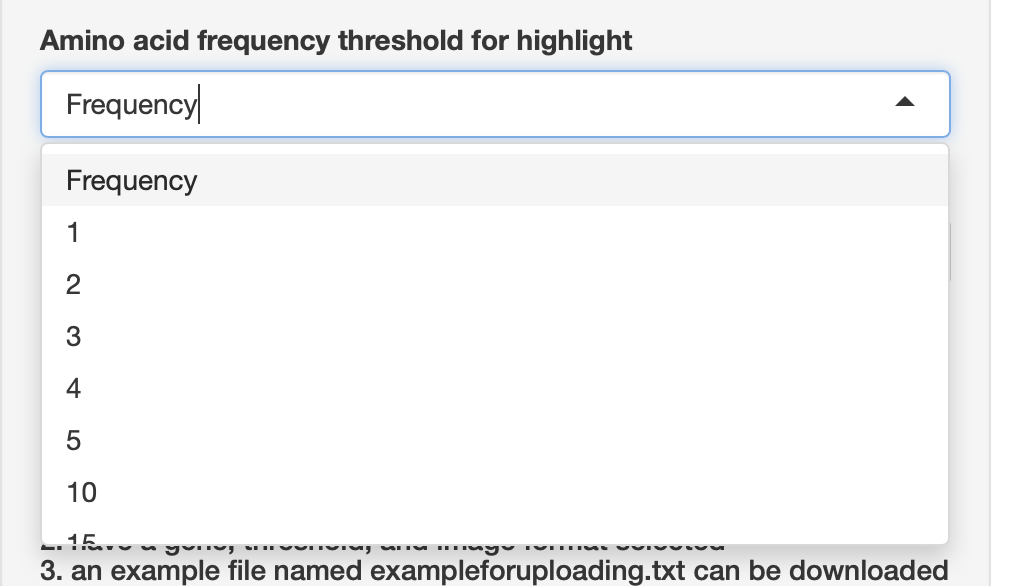
defaultでは全変異のハイライト表示は無しになっているが、数値を選ぶことで、多くのサンプルに共通する変異に絞ってハイライト表示できる。上に載せた例では、R175Hの変異が5サンプルで共通しており、他はそれぞれ1サンプルのみから由来。
ここでは1を選択、出力フォーマットにはpdfを指定。
Download Plotで図がダウンロードされる。
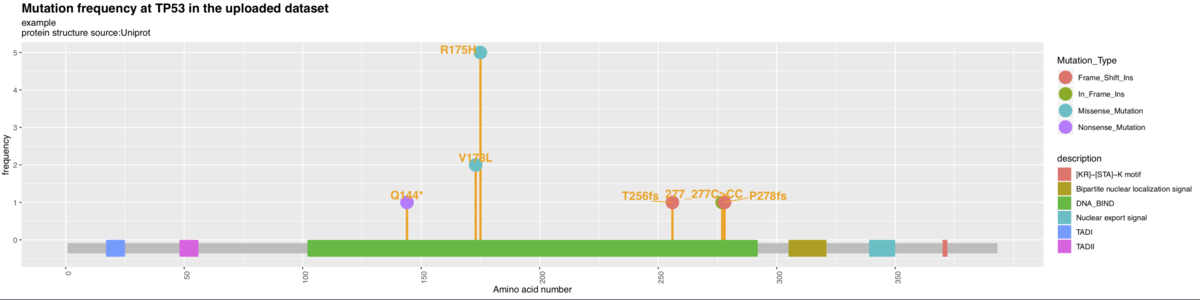
図の右端にレジェンドが表示される。変異のタイプの他、モチーフ位置も表現されている。
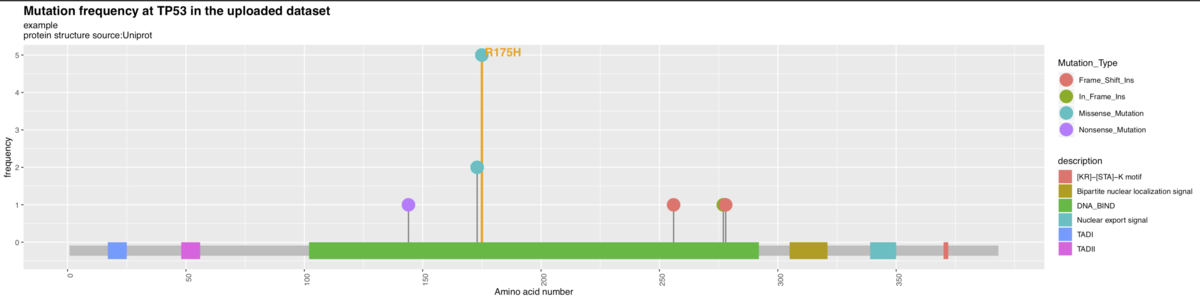
5サンプル以上で共通する変異のみハイライト表示された。
引用
Mutplot: An easy-to-use online tool for plotting complex mutation data with flexibility
Zhang W, Wang C, Zhang X
PLoS One. 2019 May 15;14(5)