ゲノムワイド研究のアウトプットは、通常、共有の発現パターンを示す遺伝子(またはそれらのタンパク質産物)のリストである。例えば、これらは、疾患の有無にかかわらずドナー群において差次的に発現される遺伝子、または生物学的サンプルの特定の画分において質量分析法によって同定されたタンパク質のリストであり得る。そのようなデータから科学的な意味を理解することは、関与する遺伝子/タンパク質およびそれらの機能に関する生物学的知識を必要とする複雑な作業である。公開されたデータが拡大するにつれて、絶えず拡大する知識と計算方法により最新情報を入手することがますます困難になっている。データベースリソースは、この知識を利用しやすくするための重要な機能である。 Gene Ontology(GO、http://geneontology.org/、[ref.1])は、molecular functions、biological processes、そしてcellular componentsを記述するための論理的定義とともに、コントロールされた階層vocabularyを維持する先駆的プロジェクトである。このコントロールされたvocabularyは、特定の遺伝子が果たす役割に関する実験的(および計算上の)知見を捉えるために、いくつかのモデル生物データベースによって利用されている。この知識は、遺伝子にアノテーションを付けるGO termを調べ、それらを機能グループに分割するために、与えられた遺伝子のリスト(遺伝子セットとも呼ばれる)に適用することができる(‘annotation’ analysis)。このアプローチは、例えばDAVIDツール[ref.2]で実装されている。もう1つの一般的な手順は、ユーザーによって送信されたエントリの一覧で大幅にover-represented しているtermのみに焦点を当てることである(‘enrichment’ analysis)。このアプローチは、Gene Ontologyアノテーションに適用されたGSEA(gene set enrichment analysis)の特定のケースである。そのような分析は、他のウェブアプリケーション(例えば、GOrilla [ref.4]、NaviGO [ref.5]、DAVID [ref.2]、AmiGO [ref.6])を使用してGOプロジェクトのウェブサイト[ref.3]から実行できる。プログラミング言語のPythonで利用可能なモジュール(例えばGOATools [ref.7]、goenrich [ref.8])およびR(例えばGOstats [ref.9]、topGO [ref.10])でも実行できる。そのようなアプローチの人気は、最初のGOC publication [ref.11]が22000以上の論文によって引用されているという事実によって強調されている(2018年10月現在のGoogle Scholar検索による)。
ただし、現在のGO分析Webアプリケーション(AmiGOやDAVIDなど)の出力は、termの階層構造を完全には伝えていない。 GOrillaやNaviGOなどのツールを使用すると、GO termの階層を視覚化できるが、分析中の遺伝子やタンパク質に対するGO termの関係は失われる。termのhierarchical structureの視覚化と遺伝子と termの関係の両方に取り組むことが、オープンソースのWebアプリケーションGOnet(https://github.com/mikpom/gonet)を作成する主な動機であった。それは遺伝子とtermのノードを持つ完全にインタラクティブなグラフを生成することによって達成される。グラフはさまざまなレイアウトをサポートしているため、グラフトポロジに基づいて分析を拡張することができる。
研究者は、時折、より詳細な情報を得るために、調査した各遺伝子産物の機能を調べる必要がある。そのようなエントリーごとの分析のために、研究者は様々な公的リソースから情報を検索する必要があり得る。 GOnetはこのアプローチに準拠し、結果として得られるビューに外部データベース(UniProt [ref.12]、Ensembl [ref.13]、DICE-DB [ref.14]、Genecards [ref.15])への便利なリンクを提供する。さらに、外部情報源からの発現データを使用して、遺伝子を色付けし、調査されたシグネチャへのさらなる洞察を提供することができる。全体的にこれらの特徴はGOnetを実験生物学者および計算生物学者のためのオミックスデータの生物学的解釈を容易にするための重要な道具にしている。
GOnetアプリケーションは入力として遺伝子シンボル、タンパク質シンボル、またはタンパク質ID(UniProt ID)のリストを受け取り、グラフを出力する(論文図1の例)。視覚化されたグラフの実際の構造とその外観に影響を与えるさまざまな入力パラメータがある。最初の主なユーザーの選択は、遺伝子に注釈が付けられているGO termである。
1、submitされた遺伝子リストにおいて統計的に有意に過剰に示されたGO term。
2、
事前定義されたサブセット(「GO slim」とも呼ばれる)、またはユーザー指定のtermリスト。
(以下略)
使い方
https://tools.dice-database.org/GOnet/ にアクセスする。
入力遺伝子名をプレーンテキスト形式で貼り付ける。 形式は一般的な遺伝子名(ABCB1, CCR7, Klf4, etc), Uniprot IDs (e.g. P32248) 、MGI IDs (e.g. MGI:1342287, mouse only)になる。Tab区切りにして2カラム目にlog2FCの値をつけても動作する。

ヒューマンデータにはUniprot IDし、マウスデータにはMGI ID使用が推奨されてる。
2種類の解析、GO term enrichment解析とGO term annotation解析がサポートされている。

defaultのGO term enrichment解析では標準的なGOenrichment解析を実行し、submitされた遺伝子リストと過剰に表されたGO termとの関係のみが示される。GO term annotation解析では、GOnetは入力遺伝子リストとGOサブセットパラメータで指定されたGO termとの関係を再構築する。
GO term annotation解析を行うにはGOサブセットを指定する。defaultはGO slim genericになっている。これはGOCによって維持されている遺伝子オントロジーのサブセットで、大部分の研究に適したカテゴリーを含んでいる。

カスタムオプションを選択し、ユーザーがGO termを指定することも可能。
GO term enrichment解析ではバッググラウンドを指定できる。defaulではAll GO annotated genesになっている。

結果は十分に具体的ではない場合、定義済みのバッググラウンドを指定することで改善される場合がある。GOnetでは、いくつかのcell type/tissueがバッググラウンドとして指定できる。

ユーザのデータセットに由来する細胞型で有意に発現される遺伝子をバッググラウンドとして使う。例えば、オプション(DICE-DB)TH1が選択された場合、Th1細胞中で1 TPMより高い発現を有する全ての遺伝子がバックグラウンドとして使用される。
エンリッチ解析のp値の計算は、Python goenrichパッケージ(link)の手順に従う。すべてのGOに対してフィッシャーの直接確率検定 (wiki)のp値が計算され、すべての遺伝子リストのGO termについて、過剰表現されたGO termはないという帰無仮説を立てて検定が実施される。
条件を決定したらsubmitする。

結果
エッジとノードからなる一種の有向グラフとして表現される。図はインタラクティブに編集可能になっている。ノードをクリックして移動したり、右のメニューから条件を変更できる。
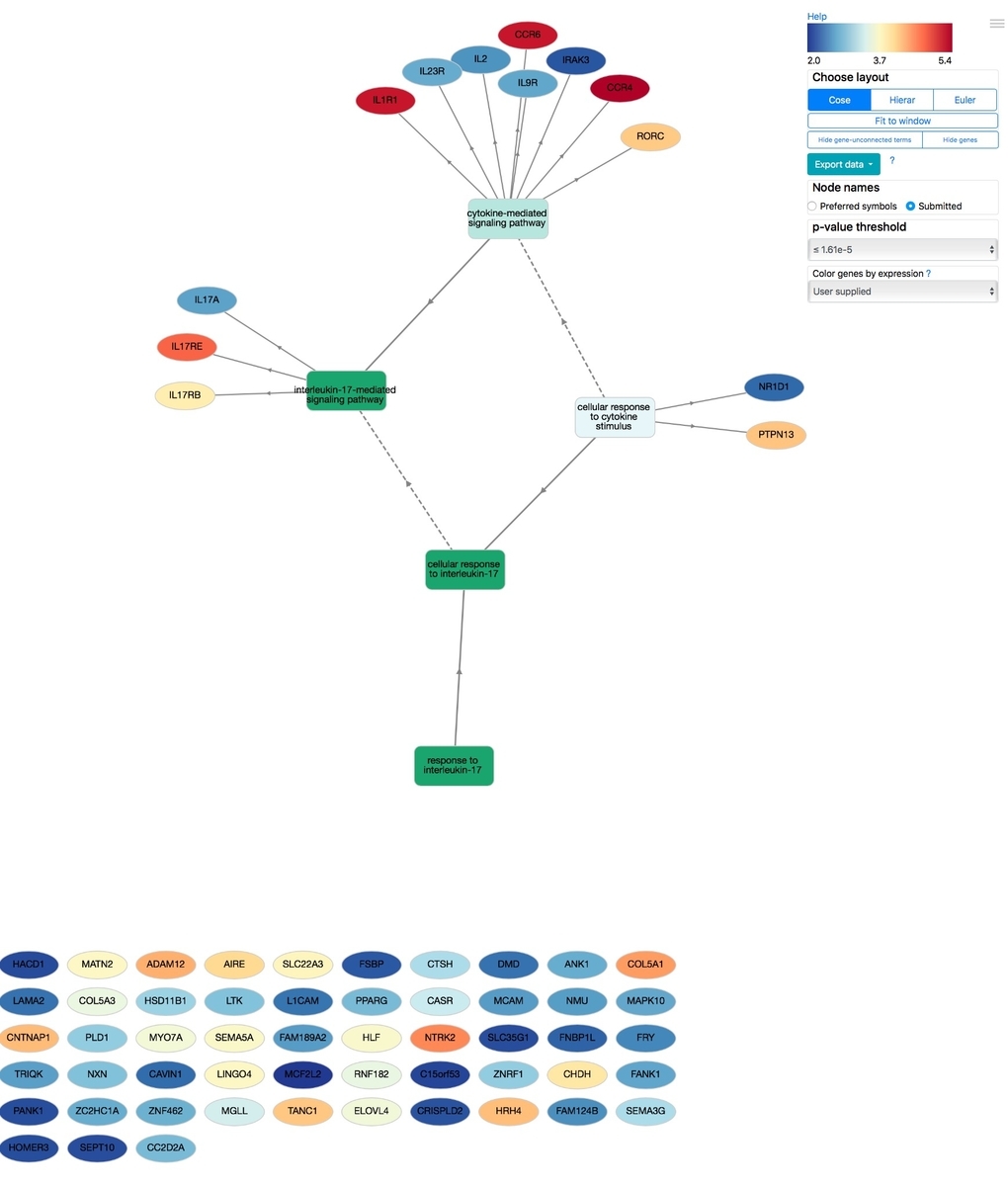
四角形のノードはGO term、楕円は遺伝子を表す。p値が小さいほどノードの色が濃くなる(GO term enrichment解析の場合)。

ノードをクリックすると詳細が表示される。

リンクをクリックするとAmiGO2にジャンプする。

レイアウトは3種類から指定できる。上のレイアウトはdefaultの"Cose"で、サンプル数が少なめの時に推奨される。サンプル数が増えると、階層構造の”Hierarchical”が推奨される。
Hierarchical

このレイアウトでは、ノードが階層内に表示される。特異性の低いGO termはネットワークの上部に配置され、特異性の高いGO termは下部に配置される。遺伝子はグラフの一番下に配置される。
もう1つ、Eulerというレイアウントも指定できる。
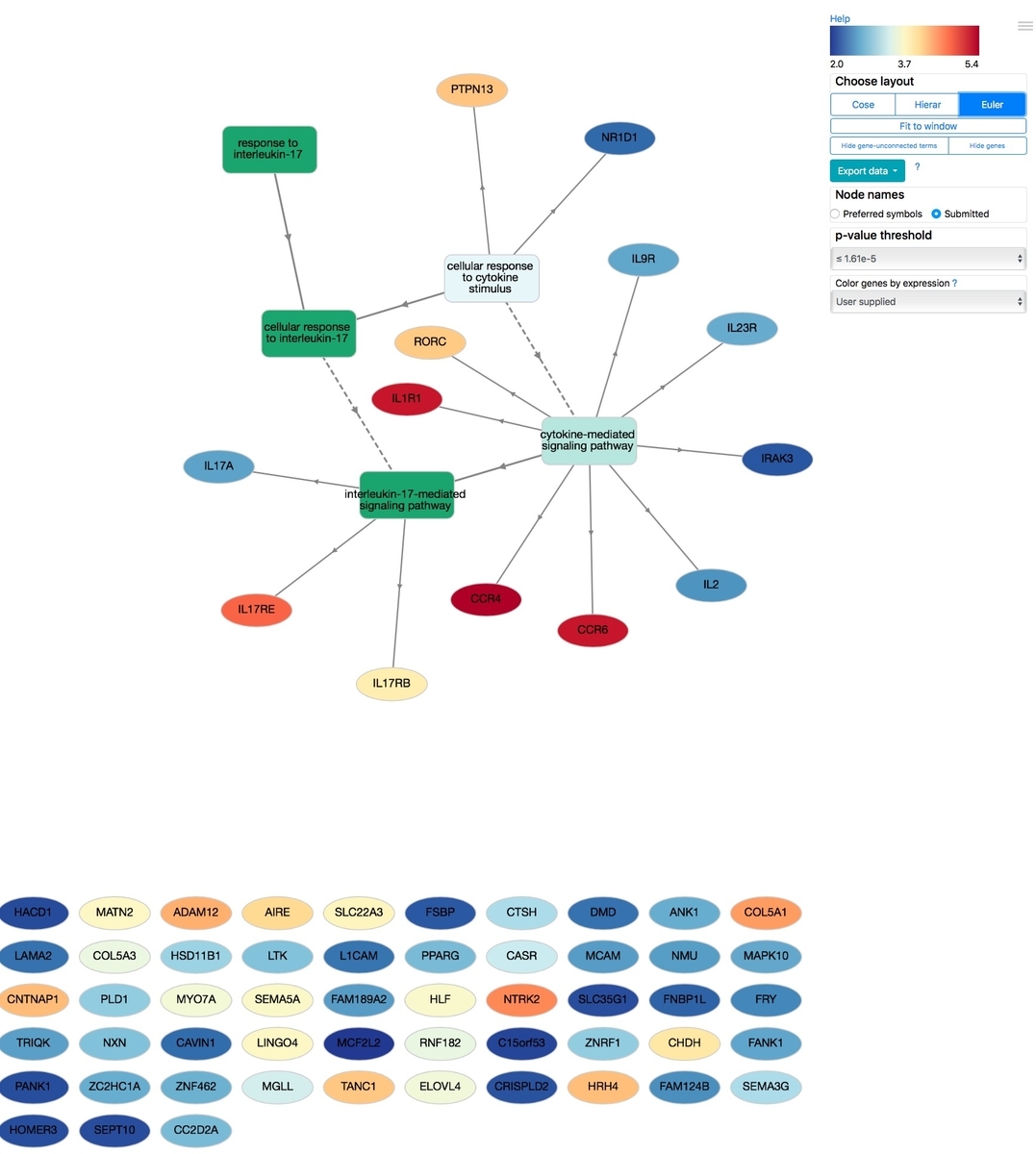
これも大規模ネットワークに適しているとされる。
メニューからはレイアウントの他にもp valueを変更できる。
threshould p-valueを減らして厳しくした。
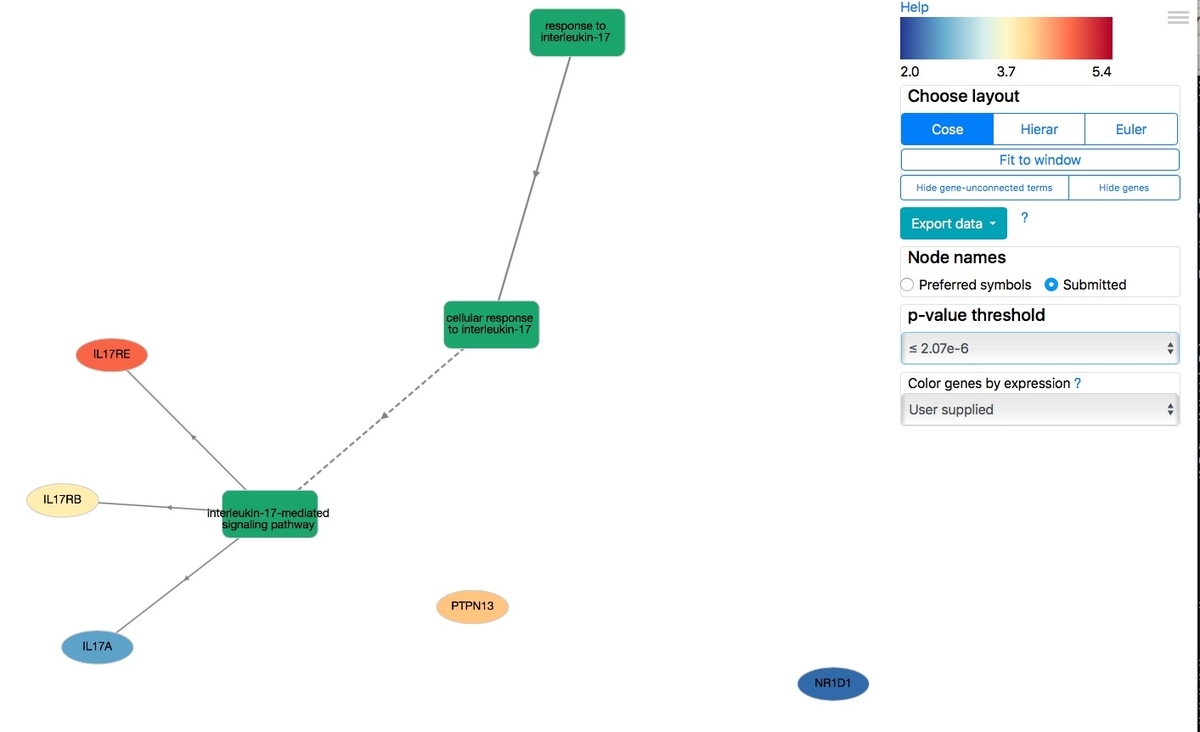
Threshould p-valueをより厳しくすると、GO termのノードの一部が見えなくなった。
引用
GOnet: a tool for interactive Gene Ontology analysis
Pomaznoy M, Ha B, Peters B
BMC Bioinformatics. 2018 Dec 7;19(1):470
