Oxford Nanopore Technologies(ONT)の小型で携帯可能な機器MinIONは、DNAシークエンシングに革命をもたらした。それはユーザーがサンプルから数時間でシーケンスまで進めることを可能にし、また非常に長いDNA分子をシーケンスすることができ、そして各フローセルからmany giga basesのデータを提供する。このため、多くの研究グループや企業が社内および現場でのシーケンスのためにこの装置を採用している。
ここではMinIONQCを紹介する。MinIONのシーケンスデータのクオリティ管理および診断分析を行うための高速、軽量、および診断分析型スクリプトである。 MinIONQCは、主に大量のシークエンシングの迅速で複製可能な比較に焦点が当てられているという点で、関連するツール(De Coster et al、2018; Loman and Quinlan、2014; Stewart and Watson、2017; Watsonら、2015)と異なる。 MinIONQCは、新しいユースケース(新しい設定など)でのMinIONシーケンスの適用や、集計が必要な大規模ゲノムプロジェクトなど、複数のフローセルからのデータ、多くのフローセルからのデータの分析(Austin et al、2017; Jain et al、2017; Jansen et al、2017; Schmidt et al、2017; Tan、2018)を迅速かつ繰り返し比較する必要がある場合に役立つ。
インストール
mac os10.14のR 3.5.1環境でテストした。
依存のインストール
#Rにて
install.packages(c("data.table",
"futile.logger",
"ggplot2",
"optparse",
"plyr",
"readr",
"reshape2",
"scales",
"viridis",
"yaml"))
本体 Github
git clone https://github.com/roblanf/minion_qc.git
cd minion_qc/
> Rscript MinIONQC.R -h
$ Rscript MinIONQC.R -h
Usage: MinIONQC.R [options]
Options:
-h, --help
Show this help message and exit
-i INPUT, --input=INPUT
Input file or directory (required). Either a full path to a sequence_summary.txt file, or a full path to a directory containing one or more such files. In the latter case the directory is searched recursively.
-o OUTPUTDIRECTORY, --outputdirectory=OUTPUTDIRECTORY
Output directory (optional, default is the same as the input directory). If a single sequencing_summary.txt file is passed as input, then the output directory will contain just the plots associated with that file. If a directory containing more than one sequencing_summary.txt files is passed as input, then the plots will be put into sub-directories that have the same names as the parent directories of each sequencing_summary.txt file
-q QSCORE_CUTOFF, --qscore_cutoff=QSCORE_CUTOFF
The cutoff value for the mean Q score of a read (default 7). Used to create separate plots for reads above and below this threshold
-p PROCESSORS, --processors=PROCESSORS
Number of processors to use for the anlaysis (default 1). Only helps when you are analysing more than one sequencing_summary.txt file at a time
-s SMALLFIGURES, --smallfigures=SMALLFIGURES
TRUE or FALSE (the default). When true, MinIONQC will output smaller figures, e.g. suitable for publications or presentations. The default is to produce larger figures optimised for display on screen. Some figures just require small text, and cannot be effectively resized.
テストラン
入力はナノポア のAlbacoreでbase callしたときにできるsequencing_summary.txtファイル

cd minion_qc/
Rscript MinIONQC.R -i example_input_minion -o my_example_output_minion -p 2
出力
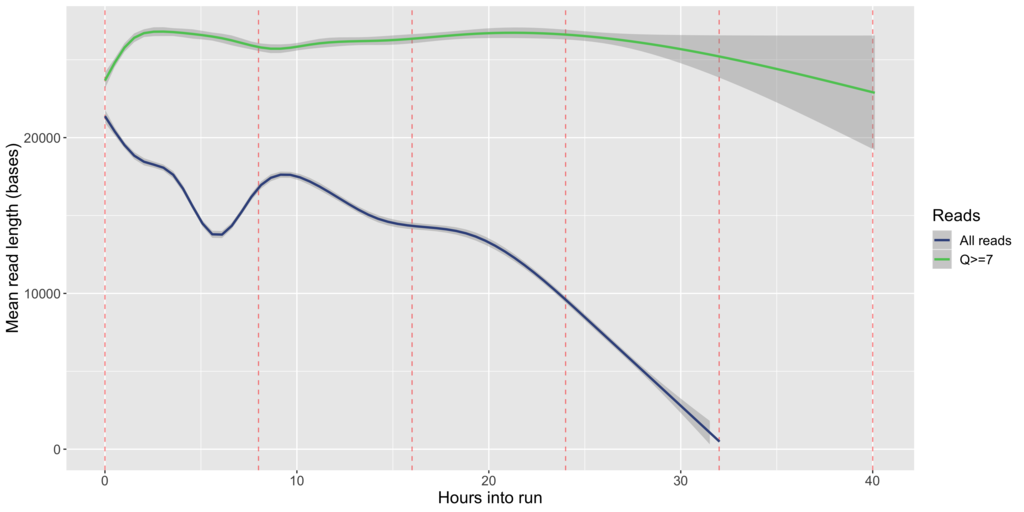
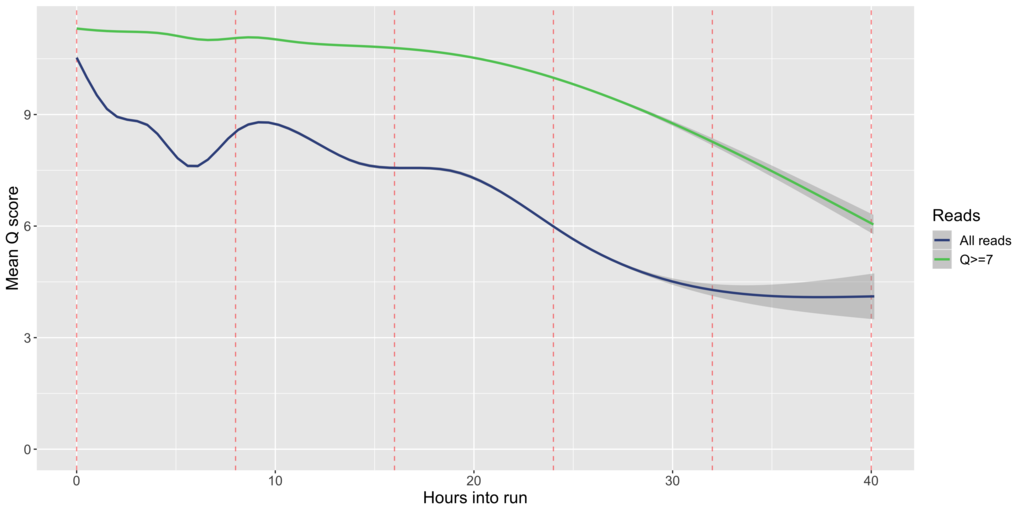
RB7_D3








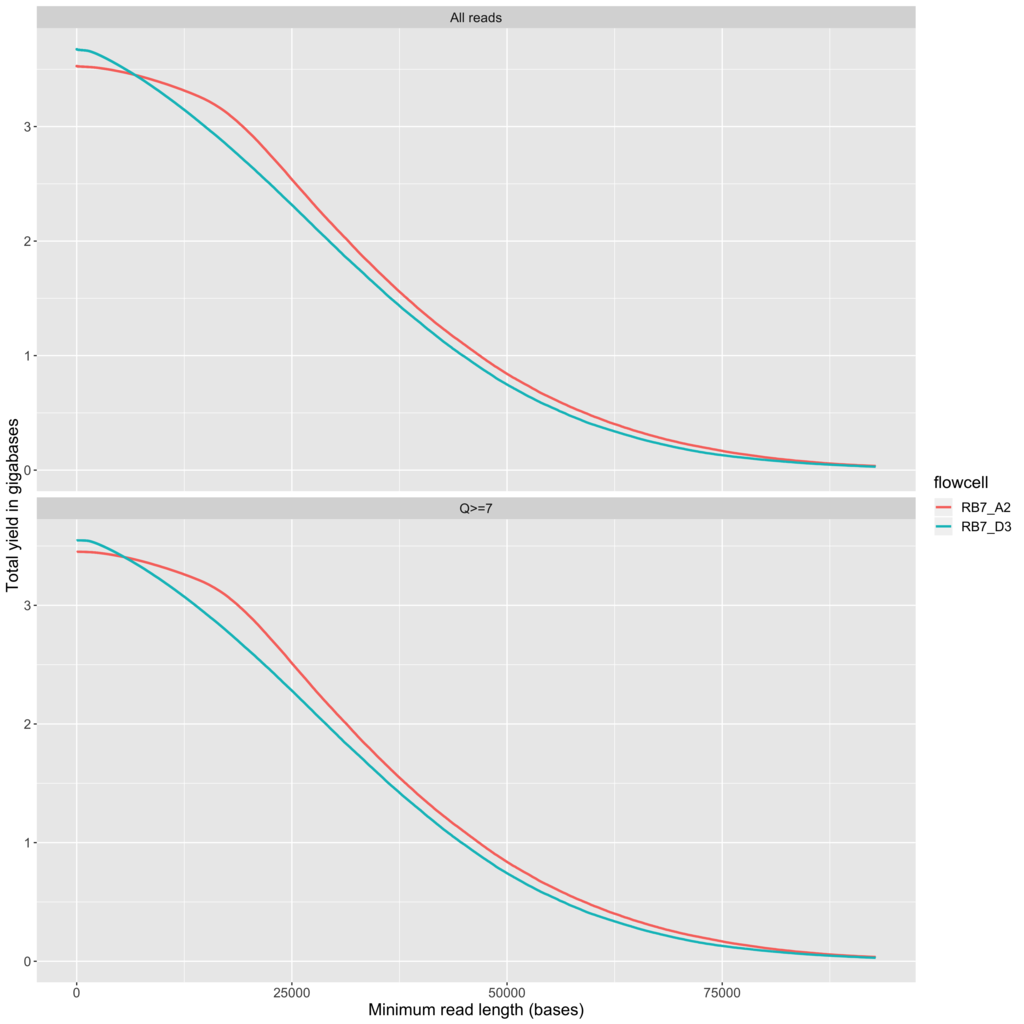
combinedQC




引用
MinIONQC: fast and simple quality control for MinION sequencing data
R Lanfear M Schalamun D Kainer W Wang B Schwessinger
Bioinformatics. 2018 Jul 23