追記 bedgraph出力
2019 9/4 インストール、twitterリンク追加
BEDファイルのオーバーラップ領域を抽出したり、マージしたりできるツール。BED以外にGFF、VCFも扱うことができる。bedtools <command> -a .bed -b .bedという使い方が基本。-aで指定したbedを-bで指定したbedと比較する。出力はリダイレクト(>)で指定しないと標準出力となる。中にはGC/AT含量を調べるコマンドまであり、多岐に渡っている。使えそうなコマンドを紹介してみます。
公式より転載。
We just dropped version 2.29.0 of bedtools. It includes a couple of nice new features and important bug fixes, so I would recommend updating. More fixes and requested features to come in the 2.30 release.#bedtoolshttps://t.co/ZDzJDjCe85
— Aaron Quinlan (@aaronquinlan) September 3, 2019
インストール
#bioconda (link)
mamba install -c bioconda -y bedtools
コマンド
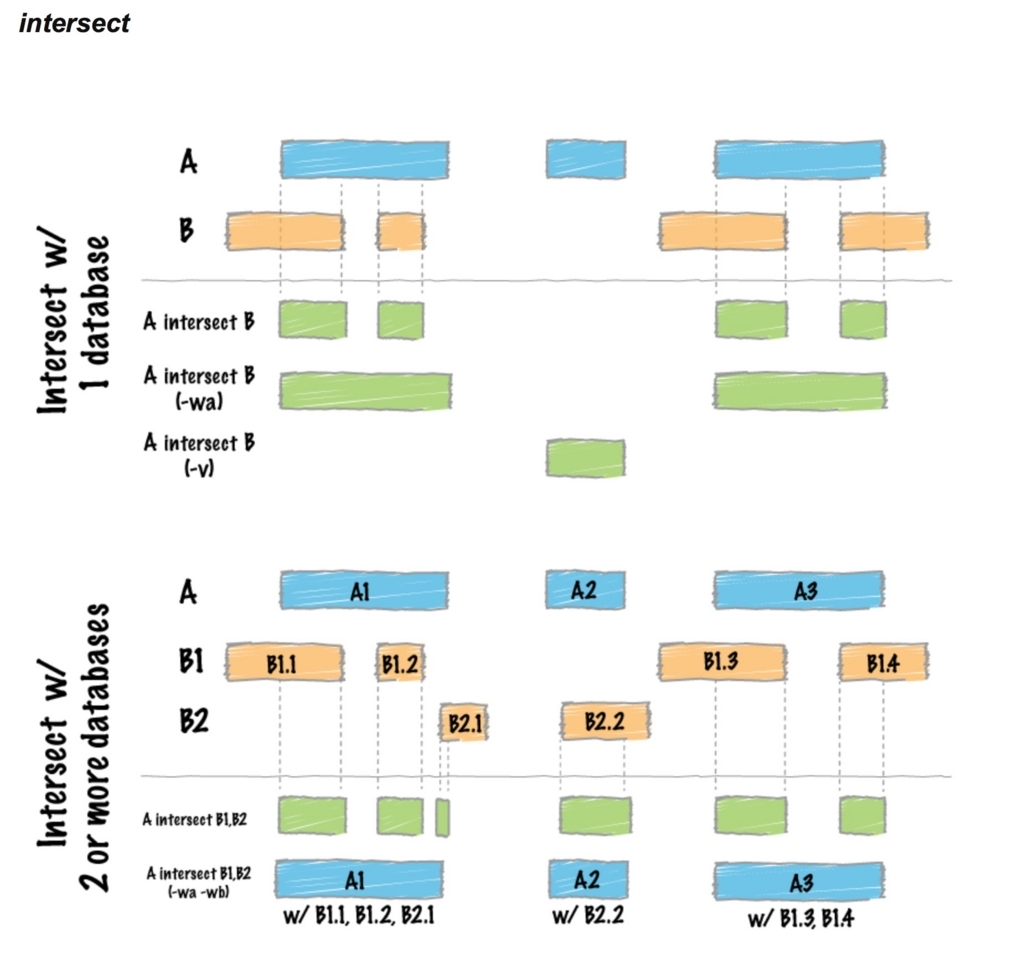
intersect
オーバーラップする領域の抽出。
aのfeatureのうちbの遺伝子とオーバーラップする領域だけをコールする。
bedtools intersect -a reads.bed -b genes.bed -sorted > output.bed
- -wa Write the original entry in A for each overlap.
- -wo Write the original A and B entries plus the number of base pairs of overlap between the two features.
- -c For each entry in A, report the number of overlaps with B.
- -f Minimum overlap required as a fraction of A. - Default is 1E-9 (i.e., 1bp). - FLOAT (e.g. 0.50)
-waをつけるとbとオーバーラップするaの領域がそのまま出力され、-waをつけないとaとbでオーバーラップする領域が出力される(参考)。-woをつけると右端カラムにオーバーラップする領域のサイズ付きで出力される。-cをつけると右端カラムにオーバーラップする領域数付きで出力される。デフォルトでは1bp以上のオーバーラップを重複とみなすが、-fで指定することでその閾値をコントロールできる。厳しくするなら、例えば-f 0.5にすれば50%以上オーバーラップを重複に設定できる。
(複数ファイル同時)オーバーラップする領域の抽出。
aのfeatureのうち3つのファイルのオーバーラップする領域をコールする。
bedtools intersect -a reads.bed -b genes1.bed genes2.bed genes3.bed -sorted > output.bed
-wa -wb をつけると重複したfeature数がわかる。-namesをつけると、どのデータ由来かわかる(-names gene1 gene2 gene3 )。
オーバーラップしない領域の抽出。
aのfeatureのうちbの遺伝子領域にない領域だけをコールする。
bedtools intersect -a reads.bed -b genes.bed -v -sorted > output.bed
Aとオーバーラップし、Bとオーバーラップしない領域の抽出。
aのfeatureのうちLINEとオーバーラップしており、かつSINEとはオーバーラップしていないfeatureを抽出。
bedtools intersect -a genes.bed -b LINES.bed |
bedtools intersect -a stdin -b SINEs.bed -v > output.bed
パイプと標準入力(stdin)を使い2回連続比較している。順次実行してもよい。
merge
オーバーラップしているfeatureを統合して返す。
bedtools merge -i reads.bed > output.bed
- -d Maximum distance between features allowed for features to be merged. - Def. 0. That is, overlapping & book-ended features are merged.
-dはdefaultでは0だが、1bp以上に指定することでオーバーラップしてないが-d指定値以内にあるfeatureを統合できる(-d 1000なら1000bp以内のfeatureを統合できる)。 -c 4 -o count,collapseをつければ、どのfeatureがどのfeatureと何回オーバーラップしたのかも出力される。
マージコマンドは、chr順、スタートポジション順にソートされていないとエラーを起こす。unixのsortコマンドでソートしてから使う(sort -k1,1 -k2,2n リンク)。似たコマンドにclusterがある。clusterはマージを実行せず、そのfeatureについてタグIDをつけて返す。
complement
featureされている領域以外の部位を返す。exon配列を入力すれば、intronとintergenicの領域が出力される。使うにはgenome配列情報が必要。
bedtools complement -i exon.bed -g genome.txt > non-exonic.bed
genomeはタブで仕切られた以下のような長さが記載してあるファイル。
User$ head -5 genome.txt
chr1 249250621
chr10 135534747
chr11 135006516
chr11_gl000202_random 40103
chr12 133851895
samtools faidxで簡単に作れる。
samtools faidx reference.fa
cut -f 1,2 reference.fa.fai > genome.txt
complementと似たコマンドにsubstractがある。
substract
featureされている領域以外の部位を返す。exon配列を入力すれば、intronとintergenicの領域が出力される。complementコマンドと異なり、bはbedファイルを指定する。
bedtools complement -a genes.bed -b introns.bed > exons.bed
例えばintron (/exon) を-bで指定して、exon (/intron) を抽出するような用途に使う。
flank
featureされている領域に隣接した領域を返す。
bedtools flank -i gens.bed -g genome.txt -b 10 > intergenic.bed
-bで抽出する配列の長さを指定する。-b 10ならfeatureの上流、下流10-bpずつが出力される。-l 2 -r 3ならfeatureの上流2-bp、下流3-bpが出力される。
slop
featureされている領域を指定値だけ増減させて返す。
bedtools slop -i reads.bed -g genome.txt -b 5 > output.bed
- -b Increase the BED/GFF/VCF entry -b base pairs in each direction. - (Integer) or (Float, e.g. 0.1) if used with -pct.
- -l The number of base pairs to subtract from the start coordinate. - (Integer) or (Float, e.g. 0.1) if used with -pct.
- -r The number of base pairs to add to the end coordinate. - (Integer) or (Float, e.g. 0.1) if used with -pct.
- -s Define -l and -r based on strand. E.g. if used, -l 500 for a negative-stranded feature, it will add 500 bp downstream. Default = false.
- -pct Define -l and -r as a fraction of the feature's length. E.g. if used on a 1000bp feature, -l 0.50, will add 500 bp "upstream". Default = false.
-bで抽出する配列の長さを指定する。-b 5ならfeatureの上流末端と下流末端がそれぞれ5-bpずつ伸びて出力される。”5 100”なら”0 105”になる。-l 2 -r 3ならfeatureの上流2-bp、下流3-bpずつ伸びて出力される。-sもつけると、-l と-rで伸びる領域がリードの方向性を考慮して決められる。
genomecov
genome全体に対するfeatureのカバレッジを返す。mergeコマンドと同様、bedはあらかじめソートされている必要がある。"-i"でbedを指定する場合はgenome配列情報も必要。"-ibam"でbamを指定する場合、genome配列情報は不要。
bedtools genomecov -i exons.bed -g genome.txt > intergenic.bed
#0カバレッジの領域だけ出力(awkフィールド4が1以下か判定)
bedtools genomecov -bga -ibam sort.bam | awk '$4 < 1' > zero-coverage
#0カバレッジの領域だけ出力。awkの代わりにgrepを使う
bedtools genomecov -bga -ibam sort.bam | grep -w 0$ > zero-coverage
このコマンドは他より時間がかかる。-bgをつけるとfeatureのカバレッジを返す。-bgをつけないとゲノム全体のカバレッジを一定の間隔で区切って計算した値が返される。
- -ibam The input file is in BAM format. Note: BAM _must_ be sorted by position
- -bg Report depth in BedGraph format. For details, see: genome.ucsc.edu/goldenPath/help/bedgraph.html
- -bga Report depth in BedGraph format, as above (-bg). However with this option, regions with zero coverage are also reported. This allows one to quickly extract all regions of a genome with 0 coverage by applying: "grep -w 0$" to the output.
-
-strand Calculate coverage of intervals from a specific strand. With BED files, requires at least 6 columns (strand is column 6). - (STRING): can be + or -
-
-pc Calculate coverage of pair-end fragments. Works for BAM files only
似た名前のコマンドにcoverageがあるが、coverageコマンドはbed同士を比較して被っている割合、個数などを出す。
bedgraph出力
bedtools genomecov -i exons.bed -ibam input.bam -bg > bedgraph_output
coverage
aのfeatureについてbのfeatureとオーバーラップしている領域の割合、個数などの情報を返す。
bedtools coverage -a A.bed -b B.bed > coverage.bed
公式マニュアルの図がわかりやすいのでそのまま引用する。
上のaのfeatureについて、bのfeatureを使いcoverageを分析した結果はresultのようになる。-sをつけるとfeature同士の向きが同じものだけカウントされる。
closest
aのそれぞれのfeatureについて、最も近いbのfeatureをコール。
bedtools closest -a genes.bed -b ALUs.bed > output.bed
-t lastや-t firstなどをつけると、同じ距離に複数featureがあった場合の選択をコントロールできる。
- -t How ties for closest feature are handled. This occurs when two features in B have exactly the same "closeness" with A. By default, all such features in B are reported. Here are all the options:
- "all" Report all ties (default).
- "first" Report the first tie that occurred in the B file. -
- "last" Report the last tie that occurred in the B file.
bamtobed
bamをbedに変換する(bed)。
bedtools bamtobed -i reads.bam > reads.bed
bamをbedpeに変換する(bedpe)。
bedtools bamtobed -i reads.bam -bedpe > reads.bedpe
似たコマンドにBEDOPSのgtf2bedがある。gtf2bedの方がより様々なフォーマットからBEDを作成可能である。
フォーマット変換 GTF => BED - macでインフォマティクス
bamtofastq
bamをfastqに変換する。
bedtools bamtofastq -i reads.bam -fq single.fastq
- -fq2 FASTQ for second end. Used if BAM contains paired-end data. BAM should be sorted by query name (samtools sort -n aln.bam aln.qsort) if creating paired FASTQ with this option.
ペアリードのbamなら-fq2で相方の出力も指定する。
bedtobam
bedをbamに変換する。
bedToBam [OPTIONS] -i reads.bed -g <GENOME> > reads.bam
- -mapq Set a mapping quality (SAM MAPQ field) value for all BED entries. Default: 255
似たコマンドにbedpetobam(リンク)がある。
getfasta
bedからfasta配列を出力する。
bedtools getfasta -fi ref.fasta -bed reads.bed > reads.fasta
- -fi Input FASTA file
- -s Force strandedness. If the feature occupies the antisense strand, the sequence will be reverse complemented. Default: strand information is ignored.
- -bedOut Report extract sequences in a tab-delimited BED format instead of in FASTA format.
- -name Use the “name” column in the BED file for the FASTA headers in the output FASTA file.
- -fo output file (リダイレクトでも同じ)
bedで指定した領域の配列が出力される。配列はfeatureの向きを考慮して出力されるので、リファンレスと同じ配列の強制出力には-sをつける。
コマンドを使う場合、リファレンスのfastaのヘッダーはbedと同じものである必要がある。また、出力されるfastaは改行されない。長くて見にくい時はfoldコマンドで折り返す。( | fold -w 60 > reads.fasta)
window
CNVの上流/下流10000bp以内にあるgeneを調べる。
bedtools window -a CNVs.bed -b genes.bed -w 10000
-l -rをつけることで、上流と下流の数値を個別に指定できる。例えば-l 10000 -r 5000なら上流10000bp以内、下流5000bp以内となる。defaultは1000bp。smとSmをつけると向きも考慮できる。
- -sm Only report hits in B that overlap A on the _same_ strand. - By default, overlaps are reported without respect to strand.
- -Sm Only report hits in B that overlap A on the _opposite_ strand. - By default, overlaps are reported without respect to strand.
annotate
オーバーラップしているfeatureの個数、名前などの情報を返す。
bedtools annotate -both -i variants.bed -files genes.bed conserve.bed known_var.bed
- -both Report the counts followed by the % coverage. - Default is to report the fraction of -i covered by each file.
- -s Require same strandedness. That is, only counts overlaps on the _same_ strand. - By default, overlaps are counted without respect to strand.
- -S Require different strandedness. That is, only count overlaps on the _opposite_ strand. - By default, overlaps are counted without respect to strand.
例えば変異をコールしたファイルのfeatureをgene.bedと比較して、コールされた部位のアノテーションをつけることに使える。上の例では3つのbedと比較し、同時に3つのアノテーションをつけている。-sや-Sを指定することでリードの向きを限定できる。
groupby
featureをグループ分けして、様々な情報を返す。
bedtools groupby [OPTIONS] -i <input> -g <group columns> -c <op. column> -o <operation>
タブ仕分けされたファイルならなんでも使えるため、 公式ページではbed以外のテキストファイルの分析ツールとしての利用価値もあるとしている。
作成途中
maskfasta
指定した領域以外をNにしてfastaを返す。
bedtools maskfasta -fi ref.fasta -bed genes.bed -fo output.fasta
- -fi Input FASTA file
- -bed BED/GFF/VCF file of ranges to mask in-fi
- -fo Output FASTA file
- -soft Enforce "soft" masking. That is, instead of masking with Ns, mask with lower-case bases.
-softをつけると、指定した領域以外をNでなく小文字にして返す。
shift
featureを指定した数値だけ移動させる。
bedtools shift -i reads.bed -g genome.txt -s 5 > reads_shifted.bed
- -s Shift the BED/GFF/VCF entry -s base pairs. Integer or Float (e.g. 0.1) if used with -pct.
- -m Shift entries on the - strand -m base pairs. Integer or Float (e.g. 0.1) if used with -pct
- -p Shift entries on the + strand -p base pairs. Integer or Float (e.g. 0.1) if used with -pct
- -pct Define -s, -m and -p as a fraction of the feature’s length. E.g. if used on a 1000bp feature, -s 0.50, will shift the feature 500 bp “upstream”. Default = false.
-s 5なら5bpポジションが下流にシフトする。-p 2 -m -3なら+のfeatureは2-bp下流、-のfeatureは3-bp上流にそれぞれシフトする。-s 0.5 -pctなら100-bpのfeatureが50-bp下流にシフトする。
nuc
featureの塩基組成を分析して返す。
bedtools nuc -fi input.fasta -bed genes.bed > oputput.bed
以下の情報が出力される。
- 1) %AT content
- 2) %GC content
- 3) Number of As observed
- 4) Number of Cs observed
- 5) Number of Gs observed
- 6) Number of Ts observed
- 7) Number of Ns observed
- 8) Number of other bases observed
- 9) The length of the explored sequence/interval.
- 10) The seq. extracted from the FASTA file. (opt., if -seq is used)
- 11) The number of times a user's pattern was observed.
GC/AT含量の分析に広く活用できると思われる。
random
指定された配列からrandomにfeatureを発生させる。
bedtools random -g genome.txt |sort -k1,1 -k2,2n > randam.bed
- -l The length of the intervals to generate. Default = 100
- -n The number of intervals to generate. Default = 1,000,000
部位がランダムに選ばれるので、sortコマンドに渡し、名前とポジションでソートして出力した方が見やすくなる(sort -k1,1 -k2,2n リンク)。
デフォルトではfeature間の間隔が 1,000,000なので、ゲノムが小さいとたくさんfeatureが出来すぎてしまう。-nを使いゲノムサイズに合わせて調節する(featureのサイズは-lで変えられるが、サイズをランダムにすることは出来ないらしい。-nで少しオーバーラップするくらいにfeatureを発生させて、bedtools merge -dすれば少しランダムなサイズのfeatureになる。しかし真のランダムとは程遠い...)。
他にも様々なコマンドがあります。fisher検定して有意なのか偶然なのか判定させたり、オーバーラップの度合いを計算したりするなど多種多様なコマンドがあります。興味があれば公式ページから探してみてください。
The BEDTools suite — bedtools 2.26.0 documentation
その他、公式ページではbedtoolsを使った応用例がいくつか記載されています。exome解析やRNA seq解析のカバレッジを調べる方法などを知ってると便利です。
http://bedtools.readthedocs.io/en/latest/
時間があればここでもまとめてみます。
BEDフォーマットは別に紹介しています。
BEDがない場合、GTFやGFFから変換して作ります。
追記
bedutils
こちらも確認してください(主にヒト)。
引用
BEDTools: a flexible suite of utilities for comparing genomic features.
Quinlan AR1, Hall IM.
Bioinformatics. 2010 Mar 15;26(6):841-2.
https://www.ncbi.nlm.nih.gov/pubmed/20110278
Example usage
http://bedtools.readthedocs.io/en/latest/content/example-usage.html