2019/12/28 インストール追記
IGVはいくつかのバージョンが提供されいる。デスクトップにショートカットを作ってjavaのwebスタート版を使っている人もいるかもしれないが、コマンドラインから叩くやり方も知っておくと便利である。
まずigvを導入。igvtoolsも入れておくとよい。
#igv
conda install -c bioconda -y igv
conda install -c bioconda -y igvtools
#homebrew
brew install igv
brew install igvtools
リファンレスファイルを指定してigvを立ち上げる。
fastaを指定。
igv -g input.fa-g リファレンス配列の指定

IGVのウィンドウが出現する。
genbankファイルを指定してigvを立ち上げる。
igv -g input.gbk
終了はIGVのウィンドウを閉じるだけでよい。
すでにゲノムが登録してある時。
igv -g <name>すでにIGVに登録済みのゲノムであれば、登録名でも配列の呼び出しはできる。
例えば、下の様に登録済みの7002という配列を呼び出すとする。
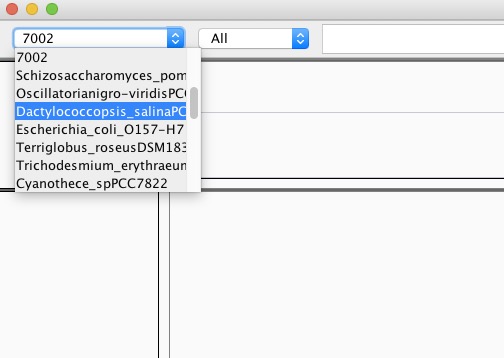
以下の様に打つ。
igv -g 70027002の配列を呼び込んだ状態でigvが起動する。
リファンレスとそこから作ったbamファイルを指定してIGVを起動する。
igv -g input.fa sample1.bam
複数のbamファイルを指定してIGVを起動する。
igv -g input.fa sample1.bam,sample2.bam,sample3.bam
複数のbamファイルとgtfファイルを指定して起動する。
igv -g input.fa sample1.bam,sample2.bam,sample3.bam,input.gtf
複数のbamファイルと変異コール結果のVCFファイルを指定して起動する。
igv -g input.fa gatk.vcf,sample1.bam,sample2.bam,sample3.bam,input.gtf
さらにgtfとbedファイルも指定して起動する。
igv -g input.fa sample1.bam,sample2.bam,input.gtf,input.bed
gtfとbedの両方の表示は、例えばRNAの解析でfusion transcriptのアノテーションファイルを作り、オリジナルのアノテーションと比較したいときなど便利である。
注意点
ファイル間にズレがあったり、ヘッダ名が違うとエラーが起こります。こういった時は、genbankやfastaなど起点となるファイルを決め、そこから全てのファイルを作り出すとズレが起こりにくくなります。ゲノム解析が進んでいる種なら、もう一度全てのデータを同じデータベースからダウンロードしてみましょう。例えばEnsembl plantだと、各種フォーマットを選んでダウンロードできるようになっています。Ensemblのリンクを貼っておきます。
plantはGTFやGFF3もダウンロードできるようです。
IGVが重い場合は以下を参考にしてください。
wiggle形式のcoverage trackを追加する流れは以下にまとめています。