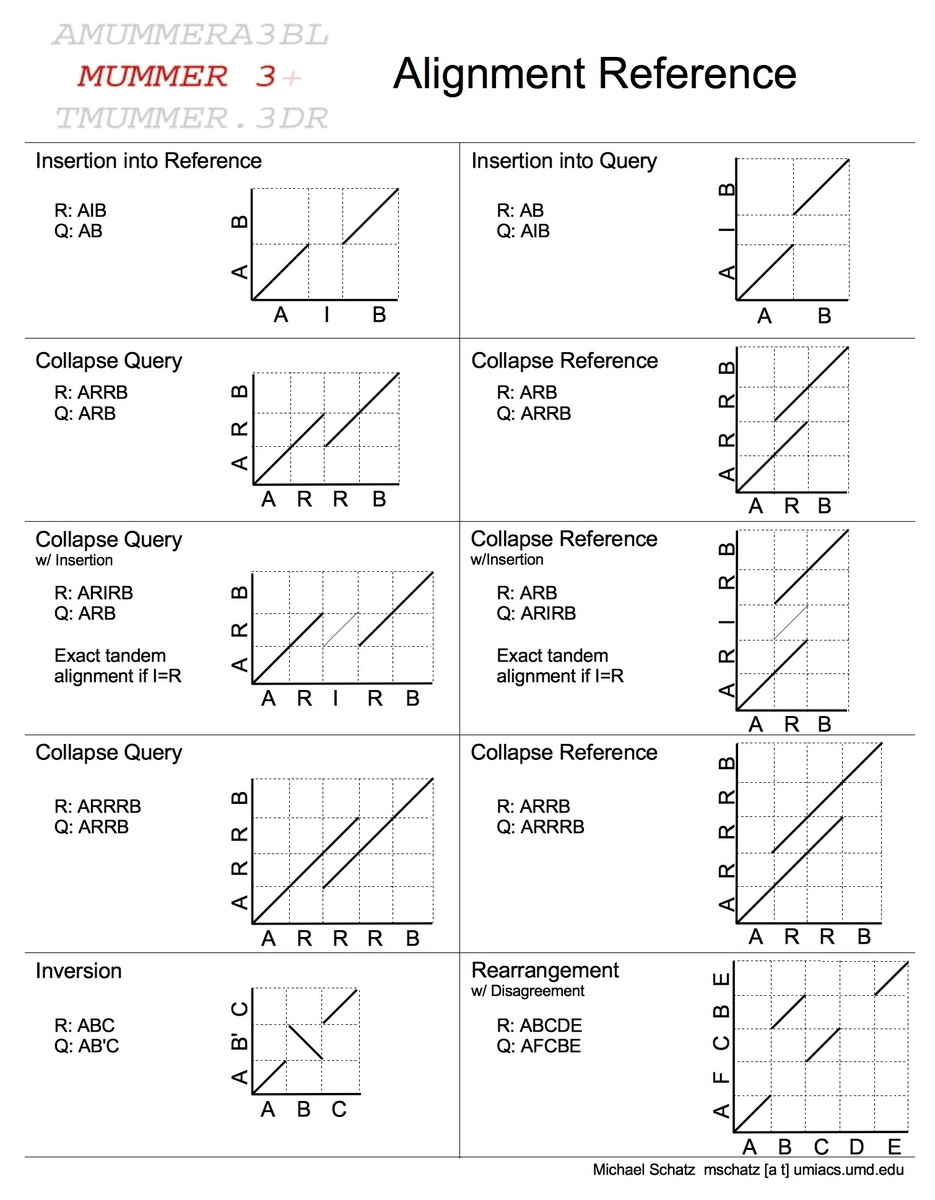
2018 9/1-9/6 アライメントワークフロー
2018 11/25 誤字修正
2019 6/9 show-tiling help追加
2019 6/12 dot plot表追加
2019 8/5 インストール追記
2019 11/12 bioconda link追加
MUMmer3 シーケンスアライメントパッケージ[mummer4論文より ref.1]の2004年のpublish以来、バイオインフォマティクスのランドスケープは劇的に変化した。シーケンスデータを生成するコストは急速に低下し、組み立てられたゲノムの数が急激に増加し、配列決定に基づくアッセイが急増した。これらの増加に伴い、効率的なシーケンスアラインメントアルゴリズムの要求が相応に増加した。アラインメントの応用には、一塩基多型(SNP)の発見、進化的に保存された要素の検出のための異なる種のシーケンス決定および比較、大規模な染色体再編成を検出するためのアラインメントなどが含まれる。アライメントアルゴリズムは、ゲノムアセンブリを作成して検証し、それらをゲノムのあるバージョンから次のバージョンに比較するためにも使用される。これらおよび他の用途は、大きなゲノムおよび大量のシーケンスデータを処理することができる迅速で信頼性の高い配列アライメント技術の必要性を動機付けている。コンピューティング速度はシーケンシング効率の向上に追いついていないが、メモリ容量の改善と並列処理を使用してカバーすることができる。特にアルゴリズムは、より大きなゲノムおよびデータセットの課題を処理するために、より大きな量のランダムアクセスメモリ(RAM)および複数のコアを必要とする可能性がある。
BLAST [ref.2]、Bowtie [ref.3]、BWA [ref.4]、Blat [ref.5]、Mauve [ref.6]、LASTZ [ref.7]およびBLASR [ref.8]を含む多くのDNAおよびタンパク質配列アライメントソフトウェアパッケージが今日利用可能である。これらのシステムのいくつかは、BWAおよびBowtie2が多数の比較的短い配列(50~300bp)をリファレンスゲノムにアライメントさせるのに最も適しているなど、特定のタイプのアライメント問題を標的とする。 BLASRは、長い高いエラー率(15-20%)のシーケンスを基準に合わせるように設計されている。 MUMmerとその関連コマンドは元々、バクテリアゲノム全体を他のゲノムにアラインメントさせるために開発されたが、広く普及している汎用アライナーに進化した。全ゲノムをアライメントさせることに加えて、MUMmer3は、可変のエラー率を有するショートおよびロングリードをリファレンスゲノムにアライメントさせることができるが、それには非効率的である。MUMmerはDNAに限定せず、タンパク質配列をアライメントさせることもできる。 MUMmerはペアワイズのゲノムアラインメントのみを生成する。すなわち、多くのゲノム配列のマルチアラインメントとは対照的にDNA配列のペアのアライメントを計算するように設計されている。しかし、現在のシーケンシング技術によって生成される非常に大きなデータセットは、入力シーケンスの最大長に対するMUMmer3の制限を超えることがある。また、これらのデータの範囲にはさらに長い実行時間が必要になる。実行時の課題に対処するために、ラッパースクリプトを使用して、長いシーケンスをより小さなものに分割し、複数の並列MUMmer3ジョブをバッチで実行することができる。しかし、このようなアドホックな並列化は、効率的で不便であり、結果として生じる複数の出力を結合して処理するための追加のステップが必要になる。
この論文(MUMmer4)では、MUMmer3をベースにして再設計され、拡張された主要な新リリースであるMUMmer4について説明する。最も大きな変化は、MUMmer4に含まれるnucmerアライナーである。 MUMmer3とMUMmer4の両方の実行可能ファイルは「nucmer」と呼ばれるがが、わかりやすくするために、ここではnucmer3とnnucmer4と呼ぶ。リファレンスのサイズ約500Mb、クエリのサイズ約4Gbの制限があるnucmer3とは異なり、nucmer4は、理論的限界である141兆塩基(Tbp)までのサイズの配列を処理することができるよう、32ビットのサフィックスツリーではなく、新しい48ビットのサフィックス配列を使用している。既知の最大のゲノムよりも1000倍大きいこの制限は、現実的なシナリオでは超過することはありそうにない。さらに、nucmer4は同じコンピュータ上で複数のコアを使用できるため、実行時間が大幅に短縮される。さらに、nucmer3はプログラムが呼び出されたときにon the flyでsuffix treeを計算し、使用後に破棄するが、nucmer4はショートリードアライナと同様に、より効率的な2ステップ動作モードを提供する。prefix配列参照を作成して保存し、クエリシーケンスのセットをアライメントさせるために繰り返しロードすることができる。 Nucmer4は、アライメントが出力される順序を除いて、nucmer3と同じ出力を生成する。以下の結果では、nucmer4とnucmer3とを比較することで、nucmer4の速度向上を実証した。この多目的シーケンスアライナーは、より特殊なアライメントタスクを処理するために最適化されたアライナーと比較しても優れている。
nucmer4の入出力フォーマットも変更され、次世代シーケンシング(NGS)用に設計されたソフトウェアパイプラインと互換性がある(オプション)。入力側では、nucmer4はFASTAとFASTQの両方のシーケンスフォーマットを受け入れるようになっている。Nucmer4は現在、ショートリードリードアライナの最も一般的な出力形式であるデルタファイルまたはSAMファイルを生成できる。 SAM出力は、SAMtools [10]を含む多くの他のパッケージと互換性がある。
オンラインマニュアル
MUMmerはゲノムを迅速にアライメントさせるためのツールである。 最初の論文が発表されたのは1999年だが、現在でも開発が続けられており、 2018年にMUMmer4のペーパーもpublishされた。 MUMmer4では、典型的な1.8 GHz Linuxコンピュータで、90 MBのメモリを使用して、5-Mbpの2つのバクテリアゲノム間の20-bpの最大完全一致を20秒で見つけることができる。 MUMmerは不完全なゲノムをアライメントさせることもでき、例えば ショットガンシーケンシングからの100~1000のコンティグをシステムに含まれるnucmerユーティリティを使用して別のコンティグまたはゲノムセットにアライメントすることができる。 promerユーティリティは、両方の入力シーケンスを6フレームで翻訳してアラインメントを行うことで、類似性を検出するには配列が違いすぎるゲノムの、タンパク質相同性による比較を可能にする。
インストール
mac os 10.12で実行した。
#bioconda (link)
#vversion4
conda install -c bioconda mummer4
#version3 bioconda (link)
conda install -c bioconda mummer==3.23-0
#高速なmambaを使う
mamba install -c bioconda mummer==3.23-0
mamba install gnuplot
#version3はbrewでも導入できる。
brew install mummer
version4はGithub からダウンロードしてビルドする(linux)。
Githubリリース
Releases · mummer4/mummer · GitHub
解凍して中に入る。
./configure --prefix=/path/to/installation #mummerのパスを指定する
make
make install
> ./nucmer -V
> ./nucmer -V
4.0.0beta2
> mummer
$ ./mummer
Usage: /home/uesaka/mummer-4.0.0beta2/.libs/lt-mummer [options] <reference-file> <query file1> . . . [query file32]
Implemented MUMmer v3 options:
-mum compute maximal matches that are unique in both sequences
-mumreference compute maximal matches that are unique in the reference-
sequence but not necessarily in the query-sequence (default)
-mumcand same as -mumreference
-maxmatch compute all maximal matches regardless of their uniqueness
-l set the minimum length of a match
if not set, the default value is 20
-b compute forward and reverse complement matches
-F force 4 column output format regardless of the number of
reference sequence inputs
-n match only the characters a, c, g, or t
-L print length of query sequence in header of matches
-r compute only reverse complement matches
-s print first 53 characters of the matching substring
-c Report the query position of a reverse complement match relative to the forward strand of the query sequence
Additional options:
-k sampled suffix positions (one by default)
-threads number of threads to use for -maxmatch, only valid k > 1
-qthreads number of threads to use for queries
-suflink use suffix links (1=yes or 0=no) in the index and during search [auto]
-child use child table (1=yes or 0=no) in the index and during search [auto]
-skip sparsify the MEM-finding algorithm even more, performing jumps of skip*k [auto (l-10)/k]
this is a performance parameter that trade-offs SA traversal with checking of right-maximal MEMs
-kmer use kmer table containing sa-intervals (speeds up searching first k characters) in the index and during search [int value, auto]
-save (string) save index to file to use again later (string)
-load (string) load index from file
Example usage:
./mummer -maxmatch -l 20 -b -n -k 3 -threads 3 ref.fa query.fa
Find all maximal matches on forward and reverse strands
of length 20 or greater, matching only a, c, t, or g.
Index every 3rd position in the ref.fa and use 3 threads to find MEMs.
Fastest method for one long query sequence.
./mummer -maxmatch -l 20 -b -n -k 3 -qthreads 3 ref.fa query.fa
Same as above, but now use a single thread for every query sequence in
query.fa. Fastest for many small query sequences.
ここでは、マニュアルがしっかりしたMUMmerのバージョン3について、代表的なコマンドmummer、nucmer、promer、run-mummer1、run-mummer3を紹介する。
ラン
mummer
サフィックスツリーデータ構造を使用して、2つのシーケンス間の最大ユニークマッチを探す。 ドットプロット表示できるマッチリストを生成するのに最も適している(リンク)。MUMmerで最も基本のコマンド。DNAもアミノ酸も比較可能。
$ mummer
Usage: mummer [options] <reference-file> <query-files>
Find and output (to stdout) the positions and length of all
sufficiently long maximal matches of a substring in
<query-file> and <reference-file>
Options:
-mum compute maximal matches that are unique in both sequences
-mumcand same as -mumreference
-mumreference compute maximal matches that are unique in the reference-
sequence but not necessarily in the query-sequence (default)
-maxmatch compute all maximal matches regardless of their uniqueness
-n match only the characters a, c, g, or t
they can be in upper or in lower case
-l set the minimum length of a match
if not set, the default value is 20
-b compute forward and reverse complement matches
-r only compute reverse complement matches
-s show the matching substrings
-c report the query-position of a reverse complement match
relative to the original query sequence
-F force 4 column output format regardless of the number of
reference sequence inputs
-L show the length of the query sequences on the header line
-h show possible options
-help show possible options
Finish genome2つの配列を比較する(3つ以上も可能)。
mummer -mum -b -c ref.fasta query.fasta > output.mums
出力は最大マッチのリストになる。

リストをもとにHarr plotを描画できる(gnuplotがなければyumやapt-getで導入しておく)。
mummerplot --postscript -p mapping output.mums
gnuplot mapping.gp
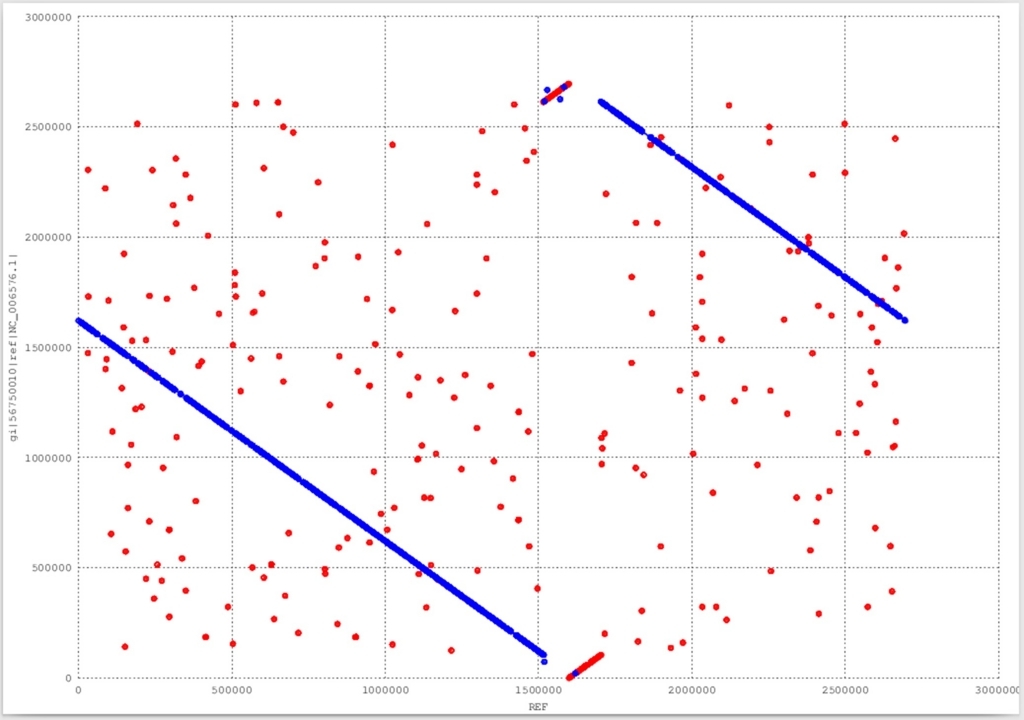
Harr plot出力。ほぼ同一だが大きなinversionが起きているゲノムの比較結果。
こちらのチートシートが役に立つ。
run-mummer1
非常によく似た2つのゲノムの比較。とSNPsやindelは考慮するが、リアレンジメントは考えない。逆向き(-r)も考慮する。nucmerとほぼ同じ。
$ run-mummer1 -help
USAGE: /Users/kazumaxneo/local/MUMmer3.23/run-mummer1 <fasta reference> <fasta query> <prefix> [-r]
run-mummer1 ref.fasta query.fasta output -r
run-mummer3
非常によく似たゲノムの比較。SNPsやindelのほか、リアレンジメントも考慮する。
$ run-mummer3
USAGE: /Users/kazumaxneo/local/MUMmer3.23/run-mummer3 <fasta reference> <multi-fasta query> <prefix>
run-mummer3 ref.fasta query.fasta output
nucmer
それなりによく似たゲノムを比較。異なる配列から似た領域を探し出す。
$ nucmer -h
USAGE: nucmer [options] <Reference> <Query>
DESCRIPTION:
nucmer generates nucleotide alignments between two mutli-FASTA input
files. The out.delta output file lists the distance between insertions
and deletions that produce maximal scoring alignments between each
sequence. The show-* utilities know how to read this format.
MANDATORY:
Reference Set the input reference multi-FASTA filename
Query Set the input query multi-FASTA filename
OPTIONS:
--mum Use anchor matches that are unique in both the reference
and query
--mumcand Same as --mumreference
--mumreference Use anchor matches that are unique in in the reference
but not necessarily unique in the query (default behavior)
--maxmatch Use all anchor matches regardless of their uniqueness
-b|breaklen Set the distance an alignment extension will attempt to
extend poor scoring regions before giving up (default 200)
--[no]banded Enforce absolute banding of dynamic programming matrix
based on diagdiff parameter EXPERIMENTAL (default no)
-c|mincluster Sets the minimum length of a cluster of matches (default 65)
--[no]delta Toggle the creation of the delta file (default --delta)
--depend Print the dependency information and exit
-D|diagdiff Set the maximum diagonal difference between two adjacent
anchors in a cluster (default 5)
-d|diagfactor Set the maximum diagonal difference between two adjacent
anchors in a cluster as a differential fraction of the gap
length (default 0.12)
--[no]extend Toggle the cluster extension step (default --extend)
-f
--forward Use only the forward strand of the Query sequences
-g|maxgap Set the maximum gap between two adjacent matches in a
cluster (default 90)
-h
--help Display help information and exit
-l|minmatch Set the minimum length of a single match (default 20)
-o
--coords Automatically generate the original NUCmer1.1 coords
output file using the 'show-coords' program
--[no]optimize Toggle alignment score optimization, i.e. if an alignment
extension reaches the end of a sequence, it will backtrack
to optimize the alignment score instead of terminating the
alignment at the end of the sequence (default --optimize)
-p|prefix Set the prefix of the output files (default "out")
-r
--reverse Use only the reverse complement of the Query sequences
--[no]simplify Simplify alignments by removing shadowed clusters. Turn
this option off if aligning a sequence to itself to look
for repeats (default --simplify)
-V
--version Display the version information and exit
nucmer --maxgap=5001 --mincluster=100 --prefix=output ref.fasta qry.fasta
#deltaファイルを変換。
show-coords -r output.delta > ref_qry.coords
#refnameとqeynameにはFASTAのヘッダー名を指定-例えばchr1とchr1を比較。
show-aligns ref_qry.delta chr1 chr1 > ref_qry.aligns #refname
nucmerの出力も、mummerplotを使ってグラフに描画できる。 データが肥大化しがちなので、マニュアルではdelta-filterコマンドを使ってはじめにone to oneのアライメント領域だけ抽出してからplotする流れが記載されている。
delta-filter -q -r ref_qry.delta > ref_qry.filter
mummerplot ref_qry.filter -R ref.fasta -Q qry.fasta
promer
かなり異なる配列の比較。 DNAレベルでは類似性が低い2つ配列も、タンパク質配列はより保存される。 promerはアライメント前にDNA配列をアミノ酸に翻訳して比較する。
promer --prefix=output ref.fasta qry.fasta
show-coords -r output.delta > output.coords #リストファイルに変換
#refnameとqeynameにはFASTAのヘッダー名を指定
show-aligns -r ref_qry.delta refname qryname > ref_qry.aligns #アライメントファイル
> head output.coords
$ head output.coords
/Users/kazumaxneo/test/A.fasta /Users/kazumaxneo/test/B.fasta
PROMER
[S1] [E1] | [S2] [E2] | [LEN 1] [LEN 2] | [% IDY] [% SIM] [% STP] | [FRM] [TAGS]
==============================================================================================================
5 772 | 1888040 1887273 | 768 768 | 68.75 80.47 0.39 | 2 -1 chr chr2
771 1 | 1887274 1888044 | 771 771 | 56.42 62.65 3.70 | -3 1 chr chr2
832 1440 | 2661990 2662556 | 609 567 | 54.19 69.95 0.00 | 1 3 chr chr2
3175 4284 | 2333649 2332540 | 1110 1110 | 58.11 67.57 4.73 | 1 -3 chr chr2
4277 3174 | 2332547 2333650 | 1104 1104 | 69.57 84.51 0.27 | -1 2 chr chr2
> head -n 20 output.aligns
$ head -n 20 /Users/kazumaxneo/test/output.aligns
/Users/kazumaxneo/test/A.fasta /Users/kazumaxneo/test/B.fasta
============================================================
-- Alignments between chr and chr2
-- BEGIN alignment [ +2 5 - 772 | -1 1888040 - 1887273 ]
5 RHRRLAEITEMIHTASLVHDDVVDEADLRRNVPTVNSLFDNRVAVLAGD
RHRRLAEITEMIHTASLVHDDVVDE+ LRR +PTV+S F NRVAVLAGD
1888040 RHRRLAEITEMIHTASLVHDDVVDESSLRRGIPTVHSSFSNRVAVLAGD
152 FLFAQSSWYLANLDNLEVVKLLSEVIRDFAEGEILQSINRFDTDTDLET
FLFAQ+SW+LA+LD+L VVKLLS+VI D AEGEILQ +NRFD+ +E
1887893 FLFAQASWHLAHLDSLTVVKLLSQVIMDLAEGEILQGLNRFDSSLSIEV
299 YLEKSYFKTASLIANSAKAAGVLSDAPRDVCDHLYEYGKHLGLAFQIVD
YL+KSY+KTASL+ANSA+AA VLS + VCD LY+YG+ LGLAFQIVD
1887746 YLDKSYYKTASLLANSARAASVLSGSSETVCDALYDYGRSLGLAFQIVD
追記1 SNPsとindel検出
#1 nucmerのラン
nucmer --prefix=test ref.fa query.fa
show-snps -C -l -r test.delta > ref_qry.snps
- -C Do not report SNPs from alignments with an ambiguous mapping, i.e. only report SNPs where the [R] and [Q] columns equal 0 and do not output these columns
- -l Include sequence length information in the output(エル)
- -r Sort output lines by reference IDs and SNP positions
-I(アイ) をつけるとindelは出力されなくなる。
追記2
ドラフトゲノムと完全なリファレンスとの比較。
#1 nucmerのラン (nucmerでも遠すぎるならpromerを使う)
nucmer --prefix=test ref.fa query.fa
#デルタファイル "test.delta" が出力される
#2 delta-filterでマルチマッピングなど不要な情報を除く(one to oneのアライメントだけ残る)。不要ならstep2は飛ばす
delta-filter -q -r test.delta > filtered.delta
#3 画像出力1
mummerplot --postscript -p mapping filtered.delta
#mapping.gpなどが出力される。これをgnuplotの入力にしてps出力
gnuplot mapping.gp
#mapping.psが出力される
#4 デルタファイルを分析するため、デルタ出力から各アライメント位置、同一性(%)などの要約情報を表示するshow-coordsコマンドを走らせる
show-coords -rcl filtered.delta > filtered.coords
#1行に1領域の結果をまとめたcoordinatesファイルtest.coordsが出力される
#5 画像出力2。step4で得たcoordinatesファイルを使う。"-f ps"も可能。
mapview filtered.coords -f pdf -p nucmer_graph
#---------------------------------------------------------------------
#おまけ 塩基レベルのアライメントファイルが必要なら、show-alignsコマンドでalingment形式に変換する (deltaファイルさえあれば実行可)。
show-aligns test.delta chromosome1 chromosome1 > test.aligns
以下はK-12とO157のゲノムを比較した結果。
#step3出力(1-> 3) delta-filterなし

#step3出力(1-> 2 -> 3) delta-filterあり

#step5出力PDF (横長になるので"-f ps"の方がいいかも)。

上で紹介したサブコマンドのヘルプ。
> show-coords -h #show-coords
$ show-coords -h
USAGE: show-coords [options] <deltafile>
-b Merges overlapping alignments regardless of match dir
or frame and does not display any idenitity information.
-B Switch output to btab format
-c Include percent coverage information in the output
-d Display the alignment direction in the additional
FRM columns (default for promer)
-g Deprecated option. Please use 'delta-filter' instead
-h Display help information
-H Do not print the output header
-I float Set minimum percent identity to display
-k Knockout (do not display) alignments that overlap
another alignment in a different frame by more than 50%
of their length, AND have a smaller percent similarity
or are less than 75% of the size of the other alignment
(promer only)
-l Include the sequence length information in the output
-L long Set minimum alignment length to display
-o Annotate maximal alignments between two sequences, i.e.
overlaps between reference and query sequences
-q Sort output lines by query IDs and coordinates
-r Sort output lines by reference IDs and coordinates
-T Switch output to tab-delimited format
Input is the .delta output of either the "nucmer" or the
"promer" program passed on the command line.
Output is to stdout, and consists of a list of coordinates,
percent identity, and other useful information regarding the
alignment data contained in the .delta file used as input.
NOTE: No sorting is done by default, therefore the alignments
will be ordered as found in the <deltafile> input.
> delta-filter -h #delta-filter
$ delta-filter -h
USAGE: delta-filter [options] <deltafile>
-1 1-to-1 alignment allowing for rearrangements
(intersection of -r and -q alignments)
-g 1-to-1 global alignment not allowing rearrangements
-h Display help information
-i float Set the minimum alignment identity [0, 100], default 0
-l int Set the minimum alignment length, default 0
-m Many-to-many alignment allowing for rearrangements
(union of -r and -q alignments)
-q Maps each position of each query to its best hit in
the reference, allowing for reference overlaps
-r Maps each position of each reference to its best hit
in the query, allowing for query overlaps
-u float Set the minimum alignment uniqueness, i.e. percent of
the alignment matching to unique reference AND query
sequence [0, 100], default 0
-o float Set the maximum alignment overlap for -r and -q options
as a percent of the alignment length [0, 100], default 100
Reads a delta alignment file from either nucmer or promer and
filters the alignments based on the command-line switches, leaving
only the desired alignments which are output to stdout in the same
delta format as the input. For multiple switches, order of operations
is as follows: -i -l -u -q -r -g -m -1. If an alignment is excluded
by a preceding operation, it will be ignored by the succeeding
operations.
An important distinction between the -g option and the -1 and -m
options is that -g requires the alignments to be mutually consistent
in their order, while the -1 and -m options are not required to be
mutually consistent and therefore tolerate translocations,
inversions, etc. In general cases, the -m option is the best choice,
however -1 can be handy for applications such as SNP finding which
require a 1-to-1 mapping. Finally, for mapping query contigs, or
sequencing reads, to a reference genome, use -q.
> mummerplot -h #mummerplot
$ mummerplot -h
USAGE: mummerplot [options] <match file>
DESCRIPTION:
mummerplot generates plots of alignment data produced by mummer, nucmer,
promer or show-tiling by using the GNU gnuplot utility. After generating
the appropriate scripts and datafiles, mummerplot will attempt to run
gnuplot to generate the plot. If this attempt fails, a warning will be
output and the resulting .gp and .[frh]plot files will remain so that the
user may run gnuplot independently. If the attempt succeeds, either an x11
window will be spawned or an additional output file will be generated
(.ps or .png depending on the selected terminal). Feel free to edit the
resulting gnuplot script (.gp) and rerun gnuplot to change line thinkness,
labels, colors, plot size etc.
MANDATORY:
match file Set the alignment input to 'match file'
Valid inputs are from mummer, nucmer, promer and
show-tiling (.out, .cluster, .delta and .tiling)
OPTIONS:
-b|breaklen Highlight alignments with breakpoints further than
breaklen nucleotides from the nearest sequence end
--[no]color Color plot lines with a percent similarity gradient or
turn off all plot color (default color by match dir)
If the plot is very sparse, edit the .gp script to plot
with 'linespoints' instead of 'lines'
-c
--[no]coverage Generate a reference coverage plot (default for .tiling)
--depend Print the dependency information and exit
-f
--filter Only display .delta alignments which represent the "best"
hit to any particular spot on either sequence, i.e. a
one-to-one mapping of reference and query subsequences
-h
--help Display help information and exit
-l
--layout Layout a .delta multiplot in an intelligible fashion,
this option requires the -R -Q options
--fat Layout sequences using fattest alignment only
-p|prefix Set the prefix of the output files (default 'out')
-rv Reverse video for x11 plots
-r|IdR Plot a particular reference sequence ID on the X-axis
-q|IdQ Plot a particular query sequence ID on the Y-axis
-R|Rfile Plot an ordered set of reference sequences from Rfile
-Q|Qfile Plot an ordered set of query sequences from Qfile
Rfile/Qfile Can either be the original DNA multi-FastA
files or lists of sequence IDs, lens and dirs [ /+/-]
-r|rport Specify the port to send reference ID and position on
mouse double click in X11 plot window
-q|qport Specify the port to send query IDs and position on mouse
double click in X11 plot window
-s|size Set the output size to small, medium or large
--small --medium --large (default 'small')
-S
--SNP Highlight SNP locations in each alignment
-t|terminal Set the output terminal to x11, postscript or png
--x11 --postscript --png (default 'x11')
-t|title Specify the gnuplot plot title (default none)
-x|xrange Set the xrange for the plot '[min:max]'
-y|yrange Set the yrange for the plot '[min:max]'
-V
--version Display the version information and exit
> mapview -h #mapview
$ mapview -h
USAGE: mapview [options] <coords file> [UTR coords] [CDS coords]
DESCRIPTION:
mapview is a utility program for displaying sequence alignments as
provided by MUMmer, NUCmer, PROmer or Mgaps. mapview takes the output of
show-coords and converts it to a FIG, PDF or PS file for visual analysis.
It can also break the output into multiple files for easier viewing and
printing.
MANDATORY:
coords file The output of 'show-coords -rl[k]' or 'mgaps'
OPTIONS:
UTR coords UTR coordinate file in GFF format
CDS coords CDS coordinate file in GFF format
-d|maxdist Set the maximum base-pair distance between linked matches
(default 50000)
-f|format Set the output format to 'pdf', 'ps' or 'fig'
(default 'fig')
-h
--help Display help information and exit
-m|mag Set the magnification at which the figure is rendered,
this is an option for fig2dev which is used to generate
the PDF and PS files (default 1.0)
-n|num Set the number of output files used to partition the
output, this is to avoid generating files that are too
large to display (default 10)
-p|prefix Set the output file prefix
(default "PROMER_graph or NUCMER_graph")
-v
--verbose Verbose logging of the processed files
-V
--version Display the version information and exit
-x1 coord Set the lower coordinate bound of the display
-x2 coord Set the upper coordinate bound of the display
-g|ref If the input file is provided by 'mgaps', set the
reference sequence ID (as it appears in the first column
of the UTR/CDS coords file)
-I Display the name of query sequences
-Ir Display the name of reference genes
> show-tiling
$ show-tiling
USAGE: show-tiling [options] <deltafile>
Try 'show-tiling -h' for more information.
user-no-MacBook-Pro-2:dist user$ show-tiling -h
USAGE: show-tiling [options] <deltafile>
-a Describe the tiling path by printing the tab-delimited
alignment region coordinates to stdout
-c Assume the reference sequences are circular, and allow
tiled contigs to span the origin
-h Display help information
-g int Set maximum gap between clustered alignments [-1, INT_MAX]
A value of -1 will represent infinity
(nucmer default = 1000)
(promer default = -1)
-i float Set minimum percent identity to tile [0.0, 100.0]
(nucmer default = 90.0)
(promer default = 55.0)
-l int Set minimum length contig to report [-1, INT_MAX]
A value of -1 will represent infinity
(common default = 1)
-p file Output a pseudo molecule of the query contigs to 'file'
-R Deal with repetitive contigs by randomly placing them
in one of their copy locations (implies -V 0)
-t file Output a TIGR style contig list of each query sequence
that sufficiently matches the reference (non-circular)
-u file Output the tab-delimited alignment region coordinates
of the unusable contigs to 'file'
-v float Set minimum contig coverage to tile [0.0, 100.0]
(nucmer default = 95.0) sum of individual alignments
(promer default = 50.0) extent of syntenic region
-V float Set minimum contig coverage difference [0.0, 100.0]
i.e. the difference needed to determine one alignment
is 'better' than another alignment
(nucmer default = 10.0) sum of individual alignments
(promer default = 30.0) extent of syntenic region
-x Describe the tiling path by printing the XML contig
linking information to stdout
Input is the .delta output of the nucmer program, run on very
similar sequence data, or the .delta output of the promer program,
run on divergent sequence data.
Output is to stdout, and consists of the predicted location of
each aligning query contig as mapped to the reference sequences.
These coordinates reference the extent of the entire query contig,
even when only a certain percentage of the contig was actually
aligned (unless the -a option is used). Columns are, start in ref,
end in ref, distance to next contig, length of this contig, alignment
coverage, identity, orientation, and ID respectively.
公式マニュアルでは、この他、リピート領域のアライメント、ドラフトゲノムとドラフトゲノムのアライメントなどのフローについても書かれています。
引用
MUMmer4: A fast and versatile genome alignment system
Guillaume Marçais, Arthur L. Delcher, Adam M. Phillippy, Rachel Coston, Steven L. Salzberg, Aleksey Zimin
Published online 2018 Jan 26.
Versatile and open software for comparing large genomes
Stefan Kurtz*, Adam Phillippy†, Arthur L Delcher†, Michael Smoot†‡, Martin Shumway†, Corina Antonescu† and Steven L Salzberg†
Genome Biology 2004, 5:R12
Fast algorithms for large-scale genome alignment and comparison.
Delcher AL, Phillippy A, Carlton J, Salzberg SL.
Nucleic Acids Res. 2002 Jun 1;30(11):2478-83.
Alignment of whole genomes
Nucleic Acids Research, 1999, Vol. 27, No. 11 2369–2376
関連