Orthology Benchmark Service (https://orthology.benchmarkservice.org)は、Quest for Orthologsコンソーシアムによってサポート・維持されている、orthology inference 評価のゴールドスタンダードである。これは、標準的なデータセットと共通の手順で、既存および新規のオルソロジー推定法(多くの比較ゲノム学と系統解析の基盤)を比較するために不可欠なリソースである。Quest for Orthologs Consortiumは、Reference Proteomesの定期的な更新と、OpenEBenchプラットフォームを通じてアクセス可能なデータの増加を通じて、このリソースを最新の状態に維持することに専念している。今回の更新では、Vertebrate Gene Nomenclature Committeeによるキュレートされたオルソロジーのアサーションに基づく新しいベンチマークを追加し、プラットフォーム上に存在する公開予測のメタ解析例を提供した。
Quest for Orthologs (QfO)コンソーシアムは、オルソロジー推論ソフトウェアとデータベースの開発者とそのユーザーを集めたコミュニティ活動である(ref.4-8)。このコンソーシアムは10年以上活動しており(ref.9)、科学コミュニティの多様な利害関係者が共有する共通の標準を定義することを継続的に追求してきた。最も広く認知されている取り組みの一つは、様々な指標を用いていくつかのオルソロジー推定法の性能を報告する、公開ベンチマークプラットフォームである(ref.10)。
このベンチマーキングプラットフォーム(https://orthology.benchmarkservice.org/)は、一般に公開されているオルソロジー推論手法の常に変化する状況を反映するために、時間をかけて進化してきた。具体的には、リファレンスゲノムデータセットの漸進的な改良を利用し、グランドトゥルースの代理として利用できるキュレーションデータセットを活用することを目的としている(ref.10,11)。本論文では、過去2年間に行われたベンチマークプラットフォームの更新について報告する。(以下省略)
Documentation
https://orthology.benchmarkservice.org/proxy/doc
ここではベンチマークプラットフォームのサイトを簡単に見ていきます。
https://orthology.benchmarkservice.org/proxy/にアクセスする。
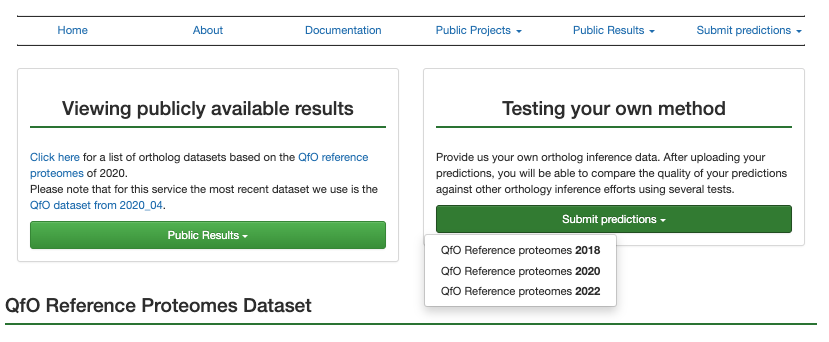
いくつかの確立されたオルソロジー推論ツールは、リファレンスプロテオームセットを使ってすでにベンチマークされている。 関心のあるツールの結果を見たいならPublic Projectsを、プロジェクトから探したいならPublic Resultsをクリックする。

Public Projectsの2020を選択した。
ベンチマークされたプログラムが1つずつ説明されている。スクロールすると26個分ある。

...
OrthoMCLの解説:”OrthoMCLは、真核生物、細菌、古細菌の幅広い生物種にわたってオルソロググループを構築するためのスケーラブルな方法を提供する。このアルゴリズムは、推定されるオルソログおよびパラログを同定するために、すべてのタンパク質のペア間のBlastスコアを計算する。Blastスコアは種間の配列分岐を考慮して正規化される。次に、OrthoMCLはマルコフクラスターアルゴリズムを用いて、密接に関連するオルソログとパラログを適切にグループ化する。"
Uploadデータをクリックすると2020のベンチマークコンペに参加者が提出したオリジナルデータを保持しているB2SHAREにアクセスできる。B2SHAREは、メタデータ付きの研究データを格納・保存・公開するためのリポジトリ(*1)。画像はOrthoMCLのアップロードデータとなる。
今度はPublic Resultsを見てみる。画像は2020の結果。

各ベンチマーク内容はオープンアクセスの論文で説明されている。Species Tree Discordance Benchmarkをクリックした。
画面の下に結果が表示される。Eukaryota、Fungi、Bacteriaに分かれている。
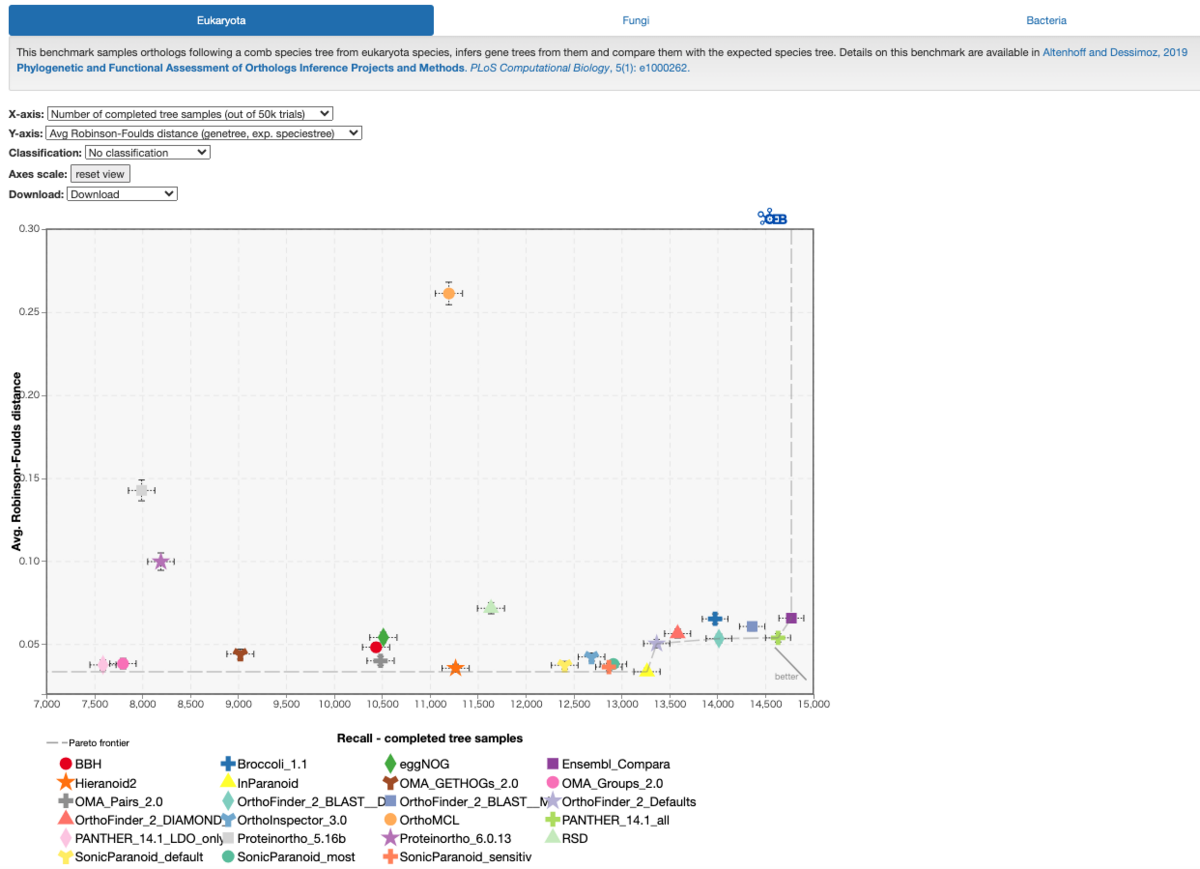
y軸はaverage Robinson-Foulds distance(wiki)、x軸はベンチマークされたツール。
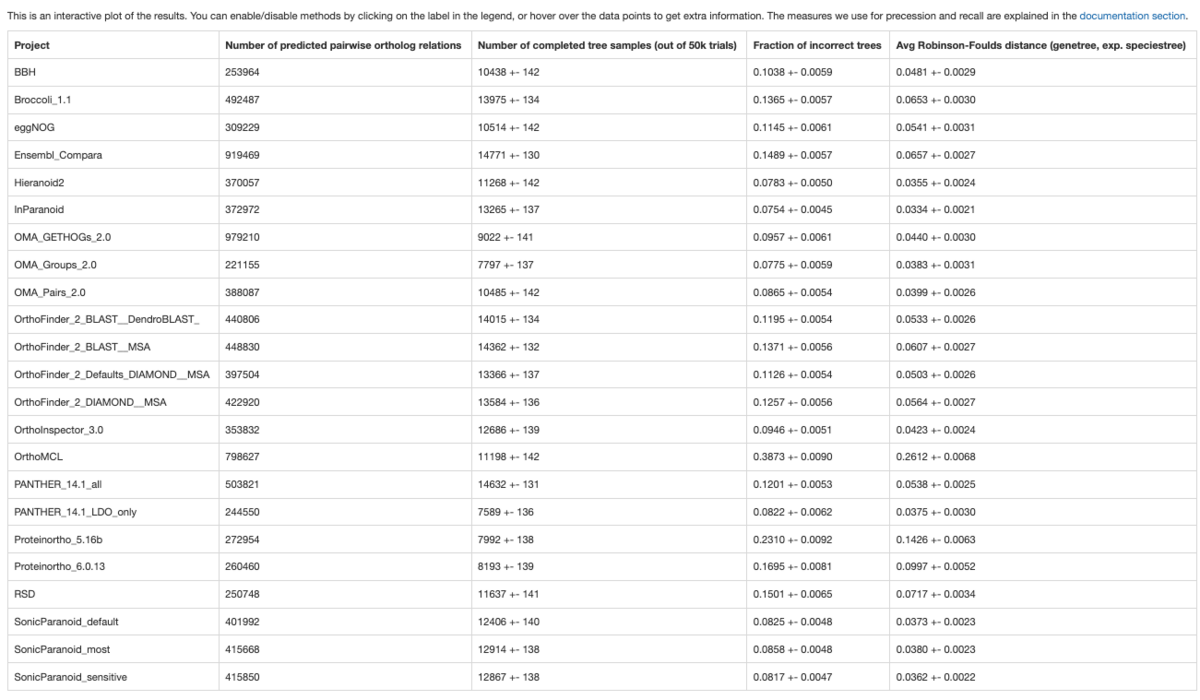
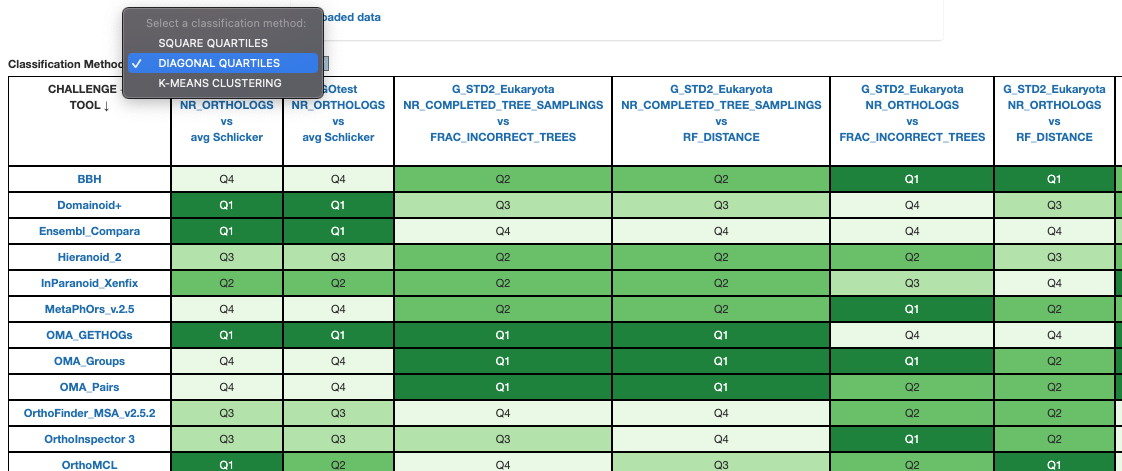
Summary tableではクラスタリングされたすべての結果の表を閲覧できる。
自分らが開発したオルソログ推論ツールを他のツールと比較して評価するなら、Testin your own methodから結果をサブミットできる。
Ortholog Benchmarking Webservice
Documentより
- Documentでは、ベンチマークで使用された3つの尺度と4つの代用尺度について説明されている。
- オルソロジー手法の開発者は、Quest for Orthologs(QfO)リファレンスプロテオームデータセットを用いてオルソログを推定する。結果はBenchmarkingサービスにアップロードされ、性能が統計解析と一般公開されているデータセットとの比較によって評価される。
- リファレンスプロテオームセットは、よく研究されているモデル生物と、生物医学研究や系統学にとって興味深いその他の生物を網羅するように選択されている。
- リファレンス・プロテオームは毎年4月に発表される。自分らが開発したオルソロジー推定法を評価したい時のベストプラクティスは、前年のリファレンスプロテオームデータセットを使ってベンチマークを実行することである。例えば、2020年6月に評価する時は、通常2019年のデータセットで解析を行う。そうすることで、すでにいくつかの比較ポイントが存在することになる。
- リファレンスプロテオームの全リリースはUniProtKBのアーカイブFTPサーバーから入手できる(HPのトップにFTPサーバーへのリンクあり)。
引用
The Quest for Orthologs orthology benchmark service in 2022
Yannis Nevers, Tamsin E M Jones, Dushyanth Jyothi, Bethan Yates, Meritxell Ferret, Laura Portell-Silva, Laia Codo, Salvatore Cosentino, Marina Marcet-Houben, Anna Vlasova, Laetitia Poidevin, Arnaud Kress, Mark Hickman, Emma Persson, Ivana Piližota, Cristina Guijarro-Clarke, the OpenEBench team the Quest for Orthologs Consortium , Wataru Iwasaki, Odile Lecompte, Erik Sonnhammer, David S Roos, Toni Gabaldón, David Thybert, Paul D Thomas, Yanhui Hu, David M Emms, Elspeth Bruford, Salvador Capella-Gutierrez, Maria J Martin, Christophe Dessimoz, Adrian Altenhoff
Nucleic Acids Research, Volume 50, Issue W1, 5 July 2022, Pages W623–W632
参考