このホワイトペーパーでは、Cytoscape用のオープンソースWikiPathwaysアプリ(http://apps.cytoscape.org/apps/wikipathways)を紹介する。WikiPathwaysアプリは、データの視覚化とネットワーク分析のためにバイオロジカルパスウェイをインポートするために使用できる。 WikiPathwaysは、手動でダウンロードしたりWebサービスを使用したりするための完全にアノテーションが付けられたパスウェイマップを提供する、オープンでcollaborativeなバイオロジカルパスウェイデータベースである。 WikiPathwaysアプリを使用すると、ユーザーは2つの異なるビューでパスウェイを読み込むことができる。
2013年8月の最初のリリースから2014年5月の論文のサブミットまでの2000以上のダウンロードは、ネットワーク生物学分野でのアプリの重要性を強調している。
Tutorial
https://cytoscape.org/cytoscape-tutorials/protocols/wikipathways-app/#/title
Visualizing Expression Data
https://cytoscape.org/cytoscape-tutorials/protocols/mapping-data/index.html#/
Cytoscape User manual
WikiPathways
インストール
macos10.14にて、Cytoscape v3.7.2を使ってテストした。
1、リンク先の右端にあるdowloadから、ビルド済みの.jarファイルをダウンロードする。
2、cytoscape3.7(link)を立ち上げ、.jarファイルを読み込む。Apps => App Manager...

3、ウィンドウ左下のinstall from fileを選択。

4、プラグインとしてcytoscapeにインストールされた。ウィンドウを閉じる。

使い方
ここからはcytoscapeで作業する。Networkの左下にある▼マークをクリック、一覧からwikipathwaysを選択。

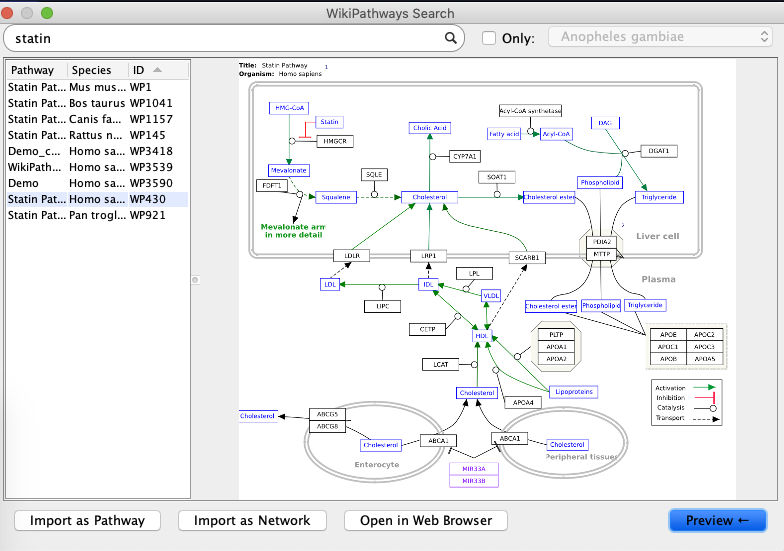
ウィンドウ内にタイプして、pathwayを検索する。ここではstatin(スタチン)とタイプ。 ウィンドウが出現するので、読み込むパスウェイを選択する。

ここではヒトのStatin pathwayを選択した。左下のimport as Pathwayボタンをクリックしてロードする。
補足==============================================================
ウィンドウ右下のpreviewボタンを押すとパスウェイを確認できる。
==============================================================
読み込まれた。ロード時、ノードには対応するEnsemblの識別子がつけられる(下の表を見ると、右端のカラムにEnsembl identifiersがついている)。

Ensemblの識別子が付いているため、対応するEnsembl識別子が付いたCSVを読み込めば、その識別子に対応するnodeやedgeに、数値の大小に応じた色をアサインできる(nodeやedgeの色、太さなどを変えるのはcytoscape標準の機能)。

ここでは発現量情報のテーブルを読み込んで、nodeやedgeに発現量に応じた色を付ける。こちらのデモデータを使用する。1カラム目はEnsembl識別子、2カラム目はcase/controlのlog2 fold changeになっている。

読み込んでいく。File => import => Table from Fileを選択。

テーブル読み込みウインドウが出てきたらTo a Network Collectionを選択。

Key Column fot NetworkはEnsemblを選択 (判別に使う情報の列がどれかを教えてあげる)。

実際にノードに色をつけていく。Style タブ=>Fill colorを選択。

Fil Color左の>をクリックすれば上のように展開される。
columnはhyper〜を選択。

Mapping typeはContinuous mappingを選択。

fold changeに応じて色がついた。

細かな色指定もできます。グラデーションのパレットをクリック。

最大と最小の値とその時の色、境界、など全てマニュアル指定できる。
パスウェイデータを読み込むときにimport as networkを選ぶと、エッジとノードからなる純粋なグラフ(ネットワーク)として読み込まれる。

ネットワークとして読み込み、ノードにfold changeによる色をつけた(手順は上の通り)。
引用
WikiPathways App for Cytoscape: Making biological pathways amenable to network analysis and visualization
Kutmon M, Lotia S, Evelo CT, Pico AR
Version 2. F1000Res. 2014 Jul 1 [revised 2014 Sep 11];3:152
参考
CYTOSCAPEを使ったデータの可視化
PDF版
http://motdb.dbcls.jp/?plugin=attach&pcmd=open&file=AJACS62_Cytoscape.pdf&refer=AJACS62