2019 6/17 インストール追記
2020 5/15 help追加
2020 7/9 ローカルでの実行例記載
2021 5/13 v6について追記
二次代謝産物または特殊代謝産物とも呼ばれる天然の産物(Natural products)は、多くの薬の基礎であり、農業および栄養学の応用にとって重要な分子でもある。さらに、分子生物学および細胞生物学の多くの側面を研究する化学プローブとして科学研究に重要な役割を果たす。多くの微生物ゲノムがこのような分子の産生をコードする複数の生合成遺伝子クラスター(BGCs: biosynthetic gene clusters)を含んでいるという科学的洞察は、天然産物研究のパラダイムシフトにつながった: ここ10年で、バイオアッセイおよびケミストリー主導の古典的な天然産物の探索手法(論文より ref.1)を補完する目的でゲノムマイニングが重要な技術として確立された。この基礎的な部分の変更は、wetの実験室の微生物研究者や化学研究者が使用可能な様々なゲノムマイニングソフトウェアツールの開発と公開によりサポートされてきた、それらのツールにはNP.searcher(ref.5)、antiSMASH(ref.6)、NaPDoS(ref.9)、そして最近のPRISM / GNP(ref.10,11)などがある。
総合的なオープンソースのBGCマイニングプラットフォームantiSMASH(ref.6-8)は、2011年に初めてリリースされ、て定期的に拡張機能付きで更新されてきた。 antiSMASHは、バクテリアや真菌のゲノムマイニングを容易にし、plantiSMASH、a new variant for BGC mining in plants (ref.12)、antiSMASHデータベース(ref.13)、実験的に「特徴づけされたBCGsのリポジトリである Minimum Information on Biosynthetic Gene Cluster (MIBiG) などと相互接続している(ref.14)。
ここではantiSMASHのバージョン4を報告する。主要な拡張機能のいくつかは、真菌BGCの遺伝子クラスター境界予測、テルペンの改善されたケミストリー予測、リボソームペプチドおよび非リボソームペプチドのBGC、トランスATポリケチドシンターゼ(PKS)のアセンブリとTTAコドンアノテーションとの比較アライメントなどのいくつかの主要な拡張を含む。さらに、改良されたユーザインタフェースが導入され、他にもいくつかのユーザビリティと効率改善が導入された。公式のantiSMASH Webサーバーはhttp://antismash.secondarymetabolites.orgから自由にアクセスできる。
Documentationより
バクテリアおよび真菌の二次代謝は、抗生物質、コレステロール低下薬または抗腫瘍薬の豊富な供給源を構成しており、現在利用されている多くの化学物質の生合成経路を含む潜在的医薬価値の生物活性化合物の構成源となっている。 興味深いことに、このような二次代謝産物の産生に関与する生合成経路をコードする遺伝子は、染色体上の特定の位置に密接に集まっていることがしばしばある。 このような遺伝子群は、「二次代謝産物生合成遺伝子クラスター」と呼ばれる。 この遺伝的アーキテクチャにより、遺伝子クラスターを突き止めることによる二次代謝産物生合成経路の直接的な検出の可能性の扉が開かれた。 近年、バクテリアおよび真菌の全ゲノムシーケンシングコストが劇的に低下し、多くのゲノム配列が利用可能になっている。antiSMASHは、特定のタイプの遺伝子クラスターに特異的な遺伝子の 隠れマルコフモデルに基づいて、既知の広範なケミカルクラスの二次代謝産物をコードする遺伝子クラスターを正確に同定することができる。 antiSMASHは、遺伝子クラスターを検出するだけでなく、詳細な配列解析も提供する。
antiSMASHが検出できるのは既知の二次代謝産物合成遺伝子クラスターである。既知の代謝産物合成遺伝子クラスターでも、検出されないものがある点に注意する。例えば代謝産物のクラスターは脂肪酸生合成やCofactor生合成系は一次代謝産物 合成系に属するため、検出されない。
antiSMASH Documentation
https://docs.antismash.secondarymetabolites.org
Using antiSMASH
https://docs.antismash.secondarymetabolites.org/using_antismash/
(2012) antiSMASH: Searching for New Antibiotics Using Open Source Tools - Kai Blin
antiSMASHに関するツイート。
使い方
ローカルサーバーを立てて利用することもできるが、ここではweb版を紹介する。
antiSMASH bacterial versionにアクセスする。
https://antismash.secondarymetabolites.org/#!/start
3つのツールがあり、antiSMASH bacterial version、antiSMASH fungal version、Plant Secondary Metabolite Analysisがあり、それぞれ、バクテリア、真菌類、植物(pubmed)がターゲットになっている。
fungal version
https://fungismash.secondarymetabolites.org/#!/start
Plant Secondary Metabolite Analysis
http://plantismash.secondarymetabolites.org
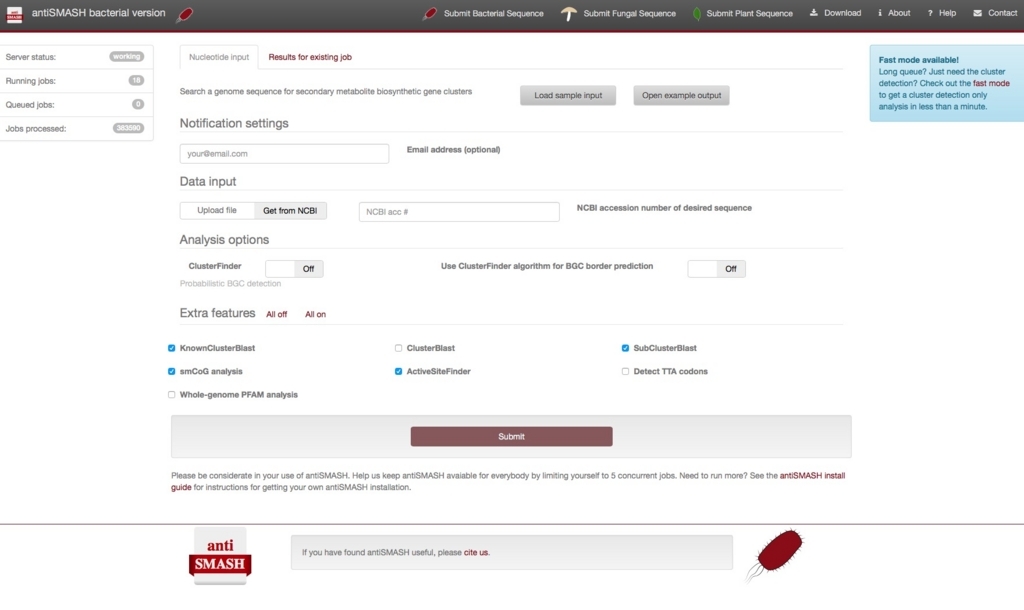
ここではbacterial versionを例に流れを確認する。

遺伝子クラスターを調べたいゲノムのGenbankファイル、GFF3ファイルをアップロードするか、NCBI accession numberを指定する。右上のexample データを選択すると、"Amycolatopsis balhimycina biosynthetic gene cluster for balhimycin"(リンク)genbankファイルのaccession number "Y16952"が読み込まれる。

データをアップロードする場合、genbank、gff3の他にアセンブリして得たFASTAもアップロードできる。FASTAはantiSMASHサーバーサイドでProdigalを使ってアノテーションされてから使用される。
他にも利用できる形式がある。詳細はantiSMASH Documentationを参照。
他の条件はdefaultのままランする。ジョブが終わると、resultsがアクティブになる。クリックすると結果の画面に移行する。
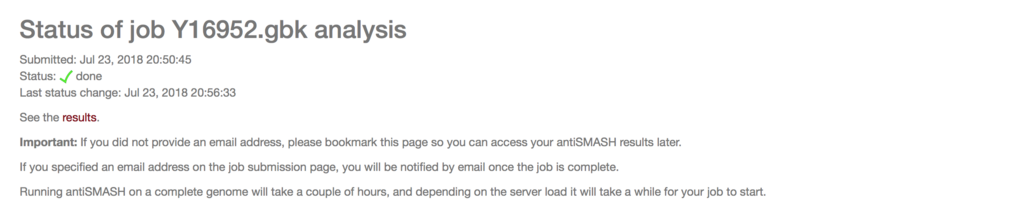
exampleデータは1つの生合成遺伝子クラスターだけ使われている。resultsには、そのクラスターがそのまま1つの遺伝子クラスターとして検出される。
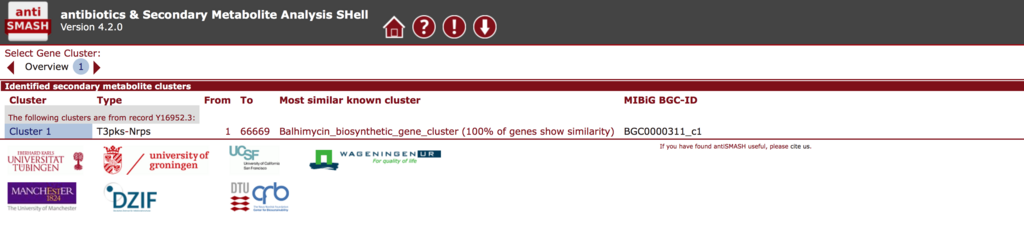
Cluster1をクリックする。遺伝子クラスターの詳細が表示される。
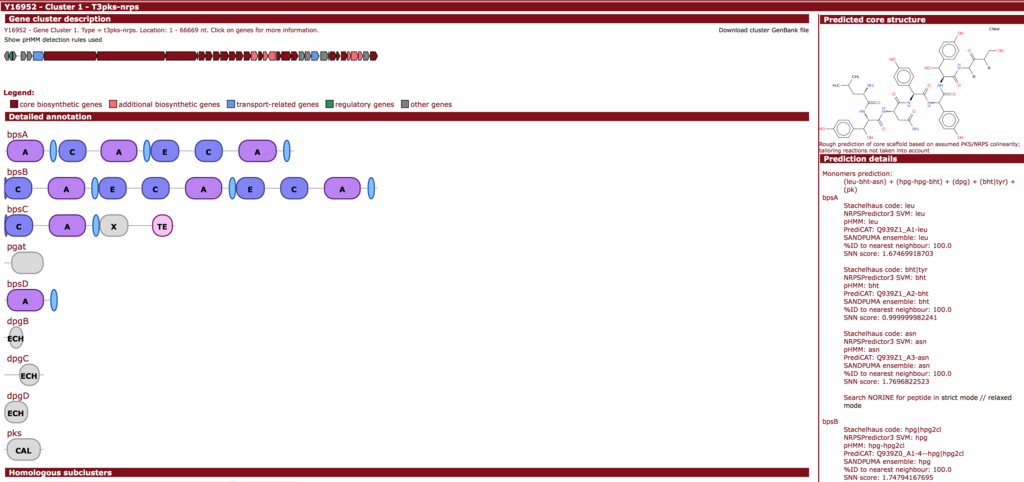
一番上が検出された遺伝子クラスターの全体像である。赤色ORFはcore biosynthetic genes、ピンクORFはadditional biosynthetic genesと予測されたORFになる。antiSMASHの予測では末端に関係ない遺伝子が余分に1−2個検出されることがある(Q&A参照)。

赤色のORFについてはdomain情報に関するアノテーションも表示される(情報がある場合のみ)。
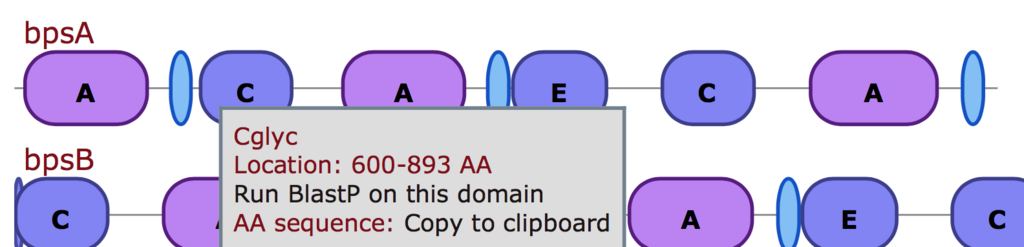
下の方にスクロールすると、検出されたクラスターと、antiSMASHのデータベース内で相同性が高かったTOP10のクラスターが表示される。

clsuter classについては次のリンク先にまとめられています。
https://docs.antismash.secondarymetabolites.org/glossary/
追記
ローカルマシンへのインストール
#bioconda (link)
#依存が多いので環境を作って導入するのが無難
conda create -n antismash -y
conda activate antismash
conda install -c bioconda -y antismash
#database (小さめ)
download-antismash-databases
> antismash
$ antismash -h
/home/kazu/anaconda3/envs/antismash/lib/python3.7/site-packages/scss/selector.py:54: FutureWarning: Possible nested set at position 329
''', re.VERBOSE | re.MULTILINE)
########### antiSMASH 5.1.2 #############
usage: antismash [-h] [options ..] sequence
arguments:
SEQUENCE GenBank/EMBL/FASTA file(s) containing DNA.
--------
Options
--------
-h, --help Show this help text.
--help-showall Show full lists of arguments on this help text.
-c CPUS, --cpus CPUS How many CPUs to use in parallel. (default: 56)
Basic analysis options:
--taxon {bacteria,fungi}
Taxonomic classification of input sequence. (default:
bacteria)
Additional analysis:
--fullhmmer Run a whole-genome HMMer analysis.
--cassis Motif based prediction of SM gene cluster regions.
--cf-borders-only Only annotate borders of existing clusters.
--cf-create-clusters Find extra clusters.
--clusterhmmer Run a cluster-limited HMMer analysis.
--smcog-trees Generate phylogenetic trees of sec. met. cluster
orthologous groups.
Lowest GC content to annotate TTA codons at (default:
0.65).
--cb-general Compare identified clusters against a database of
antiSMASH-predicted clusters.
--cb-subclusters Compare identified clusters against known subclusters
responsible for synthesising precursors.
--cb-knownclusters Compare identified clusters against known gene
clusters from the MIBiG database.
--asf Run active site finder analysis.
--pfam2go Run Pfam to Gene Ontology mapping module.
Output options:
--output-dir OUTPUT_DIR
Directory to write results to.
--html-title HTML_TITLE
Custom title for the HTML output page (default is
input filename).
--html-description HTML_DESCRIPTION
Custom description to add to the output.
Gene finding options (ignored when ORFs are annotated):
--genefinding-tool {glimmerhmm,prodigal,prodigal-m,none,error}
Specify algorithm used for gene finding: GlimmerHMM,
Prodigal, Prodigal Metagenomic/Anonymous mode, or
none. The 'error' option will raise an error if
genefinding is attempted. The 'none' option will not
run genefinding. (default: error).
--genefinding-gff3 GFF3_FILE
Specify GFF3 file to extract features from.
配列のgenbankファイルを指定する。prokkaでアノテーション付したgbkファイルも使用可能になっている。
antismash input.gbk
出力
metagenome assemblyであれば短い配列を除いてアノテーションをつけ、出力のgenbankファイルを使用する。
seqkit seq -m 5000 megahit-asssembly.fa > megahit_long_contig.fa
prokka megahit_long_contig.fa -o out_dir --metagenome --cpus 20
antismash out_dir/iPROKKA.gbk
2021 5/13
現在はベータ。submit前に以下のチェックをつける。

引用
antiSMASH 4.0-improvements in chemistry prediction and gene cluster boundary identification
Blin K, Wolf T, Chevrette MG, Lu X, Schwalen CJ, Kautsar S, Suarez Duran HG, de Los Santos ELC, Kim HU, Nave M8, Dickschat JS, Mitchell DA, Shelest E, Breitling R, Takano E, Lee SY, Weber T, Medema MH
Nucleic Acids Res. 2017 Jul 3;45(W1):W36-W41. doi: 10.1093/nar/gkx319.
The antiSMASH database, a comprehensive database of microbial secondary metabolite biosynthetic gene clusters
Blin K, Medema MH, Kottmann R, Lee SY, Weber T
Nucleic Acids Res. 2017 Jan 4;45(D1):D555-D559.
antiSMASH 3.0—a comprehensive resource for the genome mining of biosynthetic gene clusters
Tilmann Weber, Kai Blin, Srikanth Duddela, Daniel Krug, Hyun Uk Kim, Robert Bruccoleri, Sang Yup Lee,Michael A Fischbach, Rolf Müller, Wolfgang Wohlleben, Rainer Breitling, Eriko Takano, and Marnix H Medema
Nucleic Acids Res. 2015 Jul 1; 43(Web Server issue): W237–W243.
antiSMASH 2.0--a versatile platform for genome mining of secondary metabolite producers
Blin K1, Medema MH, Kazempour D, Fischbach MA, Breitling R, Takano E, Weber T.
Nucleic Acids Res. 2013 Jul;41(Web Server issue):W204-12.
antiSMASH: rapid identification, annotation and analysis of secondary metabolite biosynthesis gene clusters in bacterial and fungal genome sequences
Marnix H. Medema, Kai Blin, Peter Cimermancic, Victor de Jager, Piotr Zakrzewski, Michael A. Fischbach,4Tilmann Weber, Eriko Takano, and Rainer Breitling
Nucleic Acids Res. 2011 Jul 1; 39(Web Server issue): W339–W346.
関連