前回k-means法でクラスタリングするところまでやった。
k=9の結果が以下の通り。
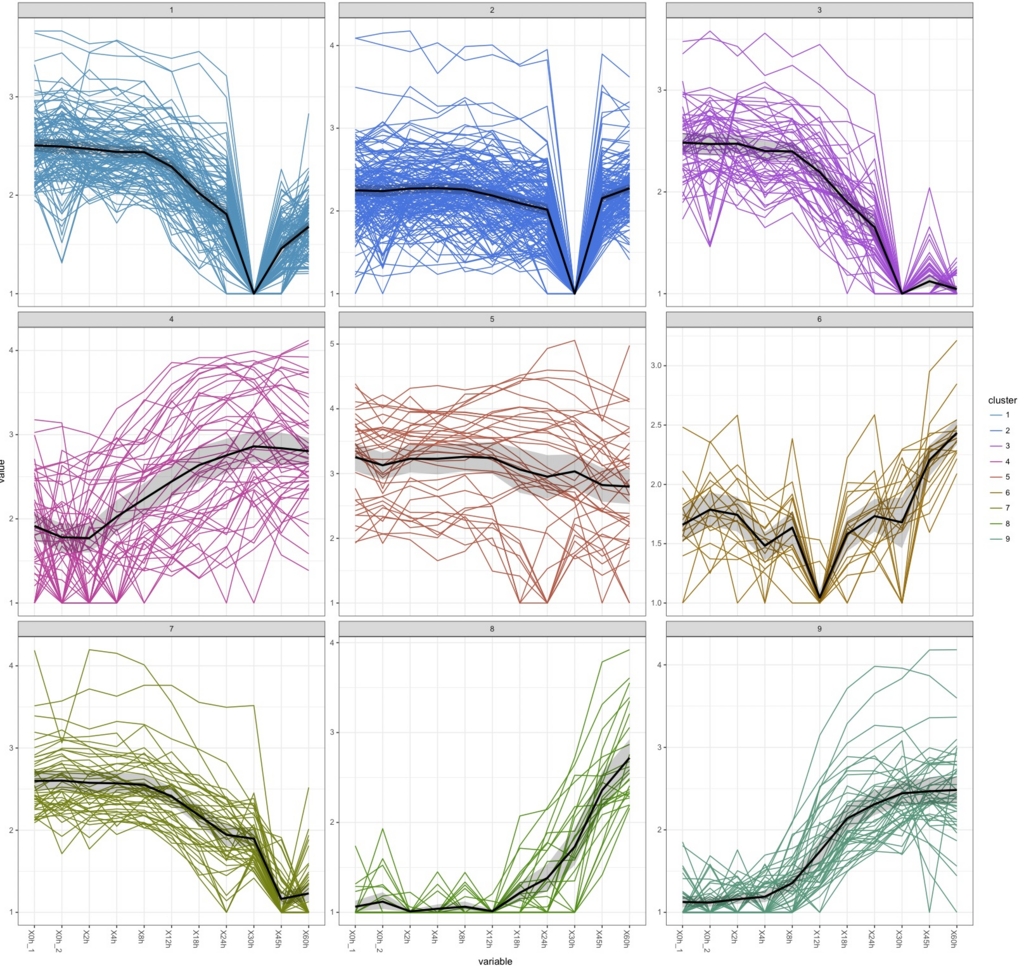
k=9の結果についてヒートマップを書いて見る。
全部を1つのヒートマップに書くとしんどいので、分けて書く。まずは1、2、3のデータパターンが似たものをヒートマップ化する。
k.meansと打つと分析結果が表示されるので、
$silinfo
$silinfo$widths
cluster neighbor sil_width
CHLREDRAFT_138936 1 2 0.4642057561
CHLREDRAFT_13257 1 2 0.4273940431
CHLREDRAFT_206075 1 2 0.4219634073
CHLREDRAFT_195343 1 2 0.4197930334
CHLREDRAFT_167270 1 2 0.4180063266
CHLREDRAFT_178353 1 2 0.4158867327
CHLREDRAFT_120099 1 2 0.4109362252
と書いてあるところをテキストエディタにコピーし、エクセルに読み込む。区分けはカンマかタブ。cluster neighbor のカラムでフィルターをかけ、1,2,3のclusterだけ取り出してcsv形式で保存 (1-3.csv)。
#読み込み
csv <- read.table("1-3.csv" , header=F, sep="\t")
前回と同じ
csv2 <- t(csv)
csv2data <-getGenes(cuff, csv2)
csHeatmap(csv2data)

まだデータが多い。
1のクラスタだけで描いてみる。

ようやく見える解像度になってきた。しかしデータに問題がある可能性がある30hのデータの落ち込みで分類されたぽいクラスタ1なので、生物的な応答パターンを見えているか怪しい。30hのデータを除いてcufflinksの定量からやり直してみる。
次回へ