2019 4/12 quast-lg追記
QUASTはspadesアセンブラで有名なAlgorithmic Biology Labのチームが発表した、アセンブリの性能や精度を評価するツール。web版とコマンドラインでランするローカル版がある。
ダウンロード
ローカル版はbrewでも導入できる。
Assembliesのところにcontig.fastaを指定、Genomeのところにリファレンスゲノムを指定してランする。リファレンスゲノムがなくても、N(G)50などのアセンブルの基礎的な評価情報は出力してくれる。
=> 解析例 (E.coli)。
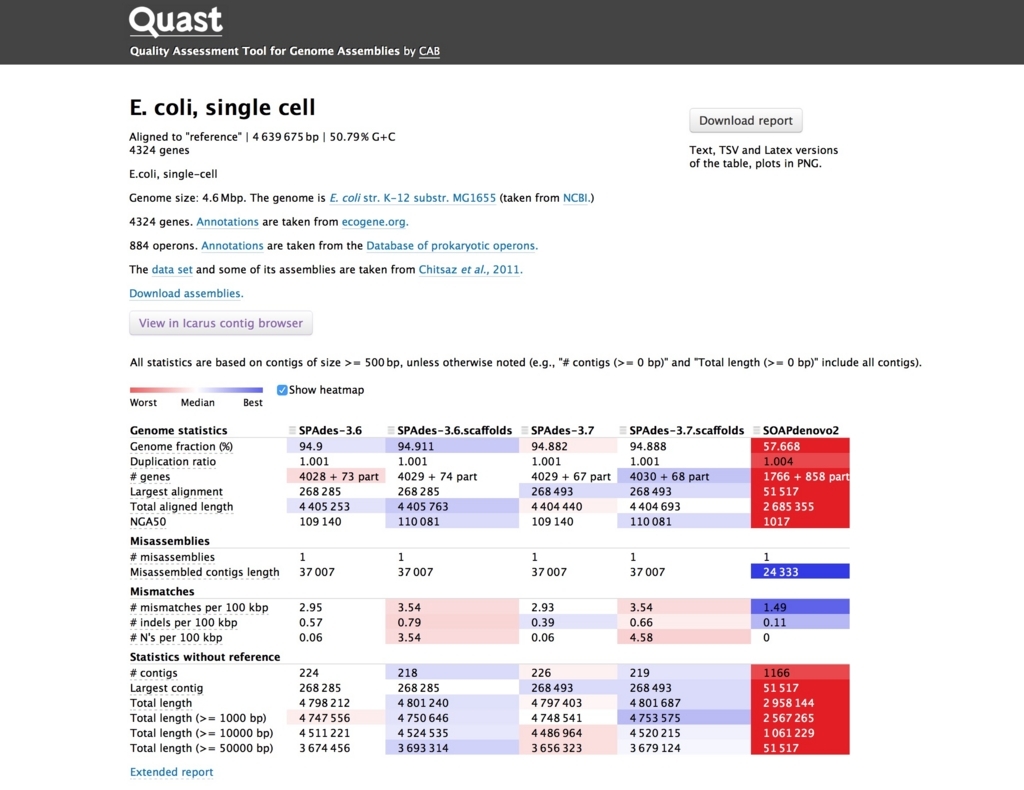
Extended reportをクリックすると詳細な情報を表示。

contigの長さの累積サイズ。左に寄っているほどアセンブル効率が良いことを表す。この例ではSOAPdenovo2は他より劣ることがわかる。
レファレンスを指定して解析した場合、viewerに移動するとアセンブルのエラーが起こっていそうな部位を確認できる。

赤い領域がリファンレスとどこかでマッチしなかったcontig。上ではcontig左末端でエラーが起こっている。上記では遺伝子予測も行なっているので、orfも表示されている。
ローカル環境でのラン
quast Contigs.fa -R reference.fa -o results -t 8
- -o Directory to store all result files [default: quast_results/results_<datetime>]
- -R Reference genome file
- -t Maximum number of threads [default: 25% of CPUs]
指定したディレクトリにhtmlファイルとPDFファイルが出力される。
Icarus Browserで確認する(htmlを開く)。
N50やNG50などのcontigは違う色がつき、N50などと表記される。

report.pdfにもいくつかの図が出力される。




bedやgbkファイルがあれば指定することでgeneの絵もviewerに表示される。
quast Contigs.fa -R reference.fa -o results -G reference.bed -t 8
- -G File with gene coordinates in the reference (GFF, BED, NCBI or TXT)
- -m Lower threshold for contig length [default: 500]
引用
QUAST: quality assessment tool for genome assemblies,
Alexey Gurevich, Vladislav Saveliev, Nikolay Vyahhi, Glenn Tesler
Bioinformatics (2013) 29 (8): 1072-1075.
アノテーション情報がない生物の場合、はRASTやprokkaなどで作成できます。
prokkaは以下でアノテーションをつけてgenbankファイルやbed、gffファイルなど出力することができます。
関連
ラージゲノムに対応